[1] 12Introdução à Modelos Estatísticos no R
Análise de dados univariados
Teste t
15 de maio de 2023
Maurício Vancine
Análises Ecológicas no R (2022)

15 capítulos: perguntas em ecologia, linguagem R, tidyverse, análises univariadas, multivariadas e geoespaciais
Conteúdo
Tempo: 2 horas
- Linguagem R (30 min.)
- Análise exploratória de dados (30 min.)
- Análise de dados multivariados (60 min.)
IMPORTANTE!!!
Estamos num espaço seguro e amigável
Sintam-se à vontade para me interromper e tirar dúvidas

1. Linguagem R
Definição
O R é uma linguagem de programação livre (open source), direcionada à manipulação, análise e visualização de dados, com diversas expansões (pacotes) para dados ou análises específicas
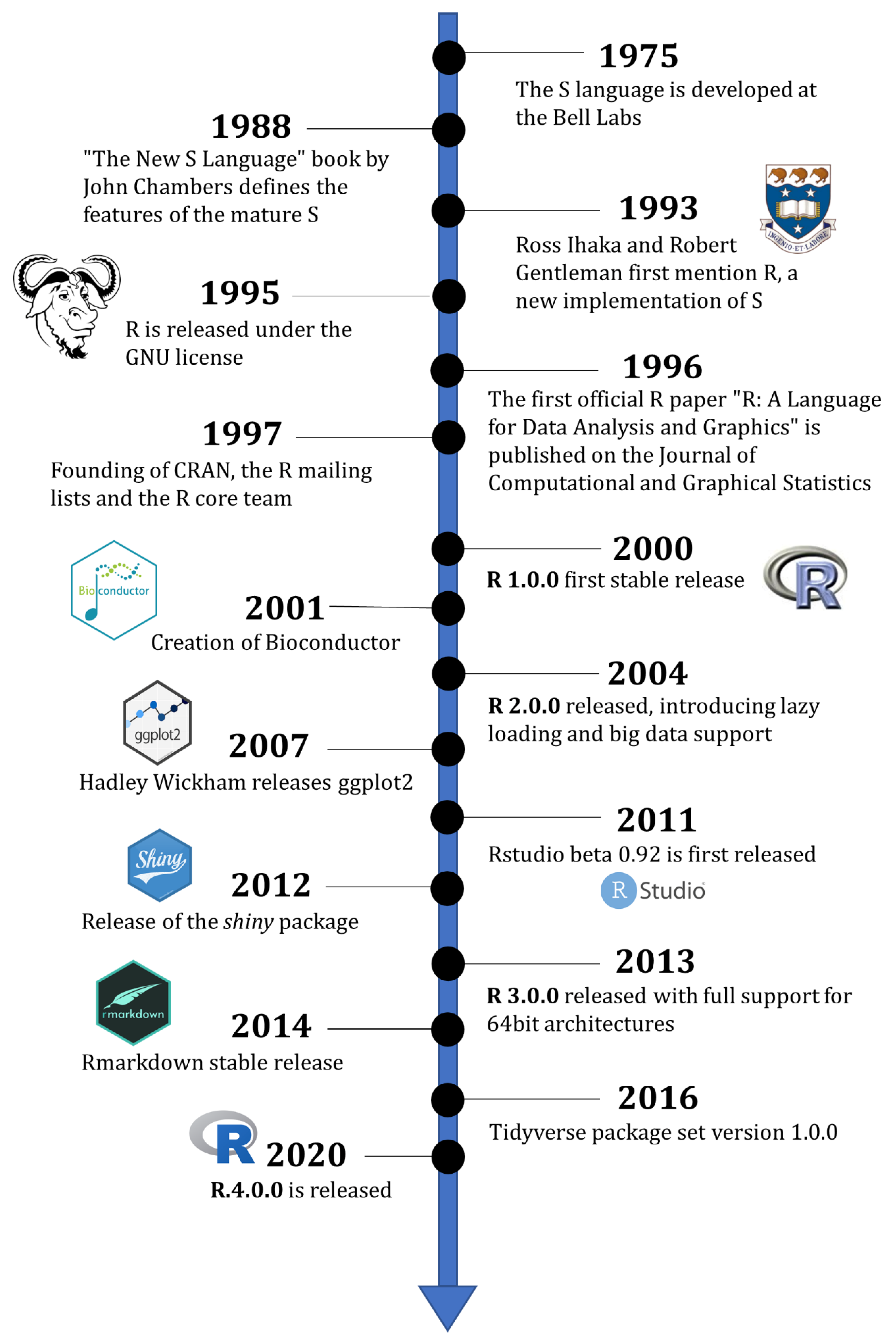
Histórico - Linguagem S
John M. Chambers (Stanford University, CA, EUA)
Versões
- Old S (1976-1987)
- New S (1988-1997)
- S4 (1998)
IDE (Integrated Development Environment)
- Interface: S-PLUS (1988-2008)

Histórico - Linguagem R
Robert Gentleman e Ross Ihaka (Auckland University, NZ)
Versões
- Desenvolvimento (1993-2000)
- Versão 1 (2000-2004)
- Versão 2 (2004-2013)
- Versão 3 (2013-2020)
- Versão 4 (2020-atual)
IDE (Integrated Development Environment)
- Interface: RStudio (2011-atual)
- Atualmente: R Core Team

Histórico - Linguagem R
Aplicações
Manipulação, visualização e análise de dados
- Estatísticas univariadas e multivariadas
- Análises de dados ecológicos
- Análise de dados espaciais, temporais e sonoros
- Análise de dados funcionais, genéticos e filogenéticos
- Análise de dados geoespaciais e sensoriamento remoto
- Visualização de todos os tipos de dados anteriores
R Markdown e quarto
- Textos em HTML, PDF, Word, ODT, Markdown
- Slides, Websites, Blogs, Livros e Artigos
- Shiny

IDE
Ambiente de Desenvolvimento Integrado (Integrated Development Environment)
IDE
Ambiente de Desenvolvimento Integrado (Integrated Development Environment)


Interface
Projeto R (.Rproj)
- Facilita o trabalho em múltiplos ambientes
- Cada projeto possui seu diretório, documentos e workspace
- Permite controle de versão (git e GitHub)

Antes de começarmos…
Conferindo os computadores

Console
O console é onde a linguagem R instalada é carregada para executar os códigos
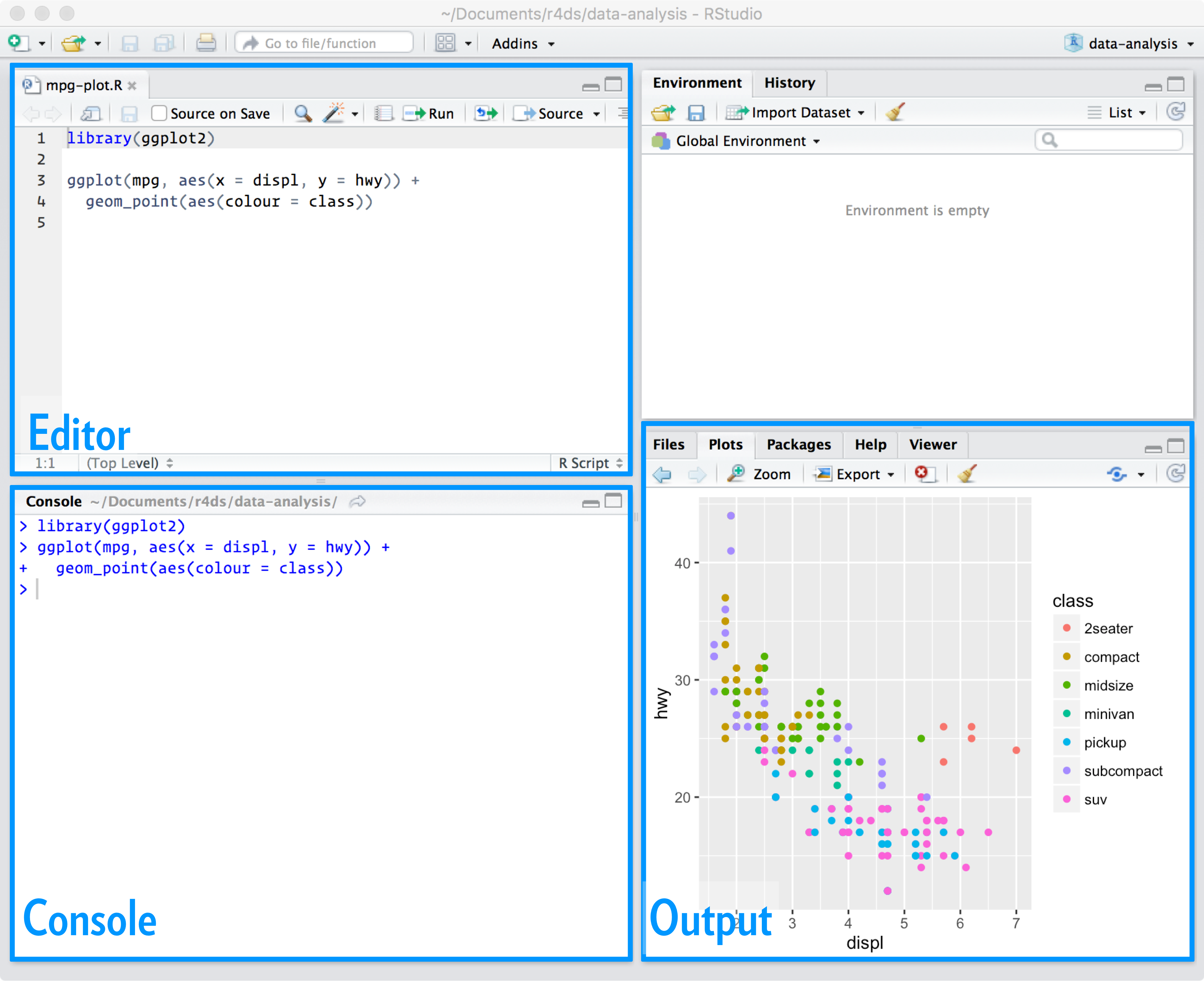
Console
Na janela do console aparece o símbolo
>, seguido de uma barra vertical|que fica piscando (cursor), onde digitamos ou enviamos nossos códigos do scriptVamos digitar
10 + 2e apertar a teclaEnterpara que essa operação seja executadaO resultado retorna o valor
12, precedido do valor1entre colchetes[1]
Console
Os colchetes
[]demonstram a posição do elemento numa sequência de valoresVamos criar uma sequência usando o operador
:para demonstrar issoO número que aparecer nos colchetes vai depender da largura das janelas
Console
Noções de programação
Número inteiro (integer)
Texto entre aspas simples ('') ou duplas ("") (character ou string)
Script
Onde os códigos são escritos e salvos no formato .R
- Atalho:
ctrl + shift + N
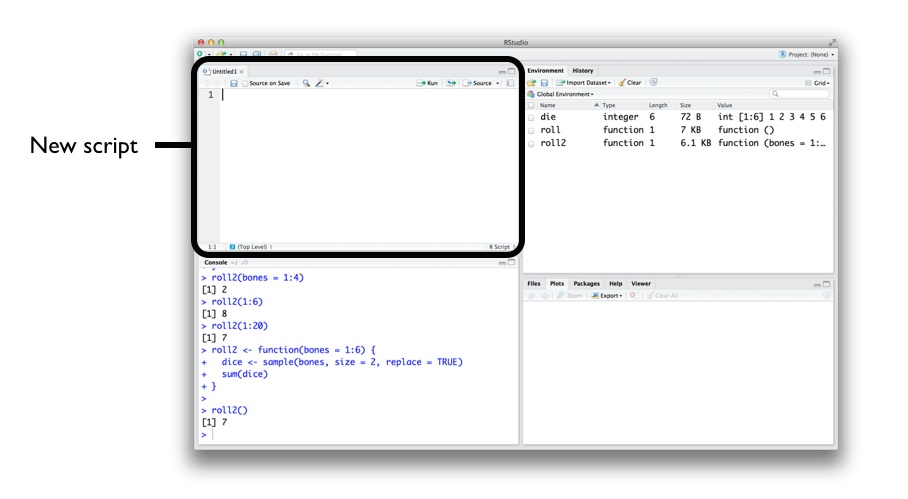
Script
Os códigos devem ser digitados preferencialmente no script
Para executar um código, deixem o cursor em qualquer lugar da linha
Atalho:
ctrl + enter
Script
Salvar um script
- Atalho:
ctrl + S

Script
Comentários (#)
Comentários não são lidos pelo R e descrevem informações em nosso script
São representados pelo
#(hash) ou#'(hash-linha)
Script
Comentários (#)
Sempre comece um script com um cabeçalho
Ajuda a lembrar o que o script faz e quando foi escrito

Operadores
Operadores aritméticos (retorna números)
| Operador | Descrição | Uso |
|---|---|---|
| + | Adição | a + b |
| – | Subtração | a - b |
| * | Multiplicação | a * b |
| / | Divisão | a / b |
| %% | Resto da divisão | a %% b |
| %/% | Quociente da divisão | a %/% b |
| ^ | Potenciação | a^b |
Operadores
Operadores relacionais (retorna TRUE|FALSE)
| Operador | Descrição | Uso |
|---|---|---|
| < | Menor | a < b |
| > | Maior | a > b |
| <= | Menor ou igual | a <= b |
| >= | Maior ou igual | a > = b |
| == | Igual | a == b |
| != | Não igual (diferente) | a!=b |
Operadores
Ordem das operações aritméticas
() > ^ > * ou / > + ou -
Objetos
Palavras que atribuímos (guardamos) dados possibilitando sua manipulação
Atribuição (
<-)palavra <- dados
Atalho:
alt + -

Objetos
Vamos atribuir o valor 10 à palavra eco

Objetos
Sempre confira a atribuição
Dica: chame o objeto novamente
Objetos
Seja criativo
O R sobrescreve os valores dos objetos com o mesmo nome
Objetos
Seja criativo, mas nem tanto…
O R tem limitações ao nomear objetos!
Começar por letras (
a-zouA-Z) ou pontos (.)Conter letras (
a-zouA-Z), números (0-9), underscores (_) ou pontos (.)Case-sensitive, i.e., ele difere letras maiúsculas de minúsculas
Evitar utilizar letras maiúsculas, acentos ou cedilha (
ç)Não podem ser iguais a nomes especiais:
break, else, FALSE, for, function, if, Inf, NA, NaN, next, repeat, return, TRUE, while
Objetos
Ambiente (Environment)
Os objetos podem ser visualizados no painel Environment

Objetos
Podemos utilizar objetos para fazer operações
Objetos
Podemos utilizar objetos para atribuir resultados de operações
Objetos
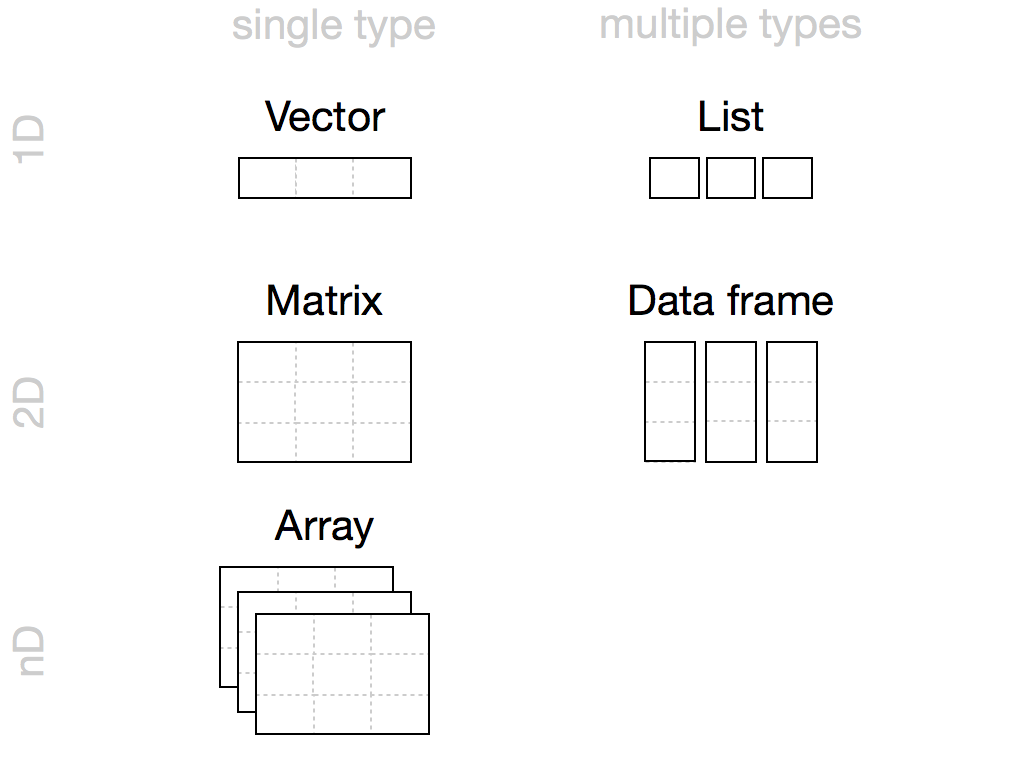
Tipos de objetos
Funções
Códigos que realizam operações em argumentos
- Estrutura de uma função:
nome_da_funcao(argumento1, argumento2)
- Nome da função: remete ao que ela faz (inglês)
- Parênteses: limitam a função
- Argumentos: onde a função atuará
- Vírgulas: separam os argumentos

Funções
Exemplos
Funções
Argumentos
Os argumentos podem ser de dois tipos:
Objetos ou valores: dados onde a função irá atuar
Parâmetros: mudam o comportamento da função (texto =
TRUE,FALSEou"texto")
Funções
Argumentos como valores
Funções
Argumentos como parâmetros
Funções
Atribuição de resultados a objetos
[1] 1 2 3 4 5 1 2 3 4 5 1 2 3 4 5 1 2 3 4 5 1 2 3 4 5Funções
Atribuição, função e linha temporal
Criar dois objetos
Somar esses objetos e atribuição
Raiz quadrada e atribuição
Funções
Atribuição, função e linha temporal
Atribuição de dados a objetos
Funções que operam e mudam esses dados
Nova atribuição desses resultados a novos objetos
Ajuda
Descreve as informações de uma função
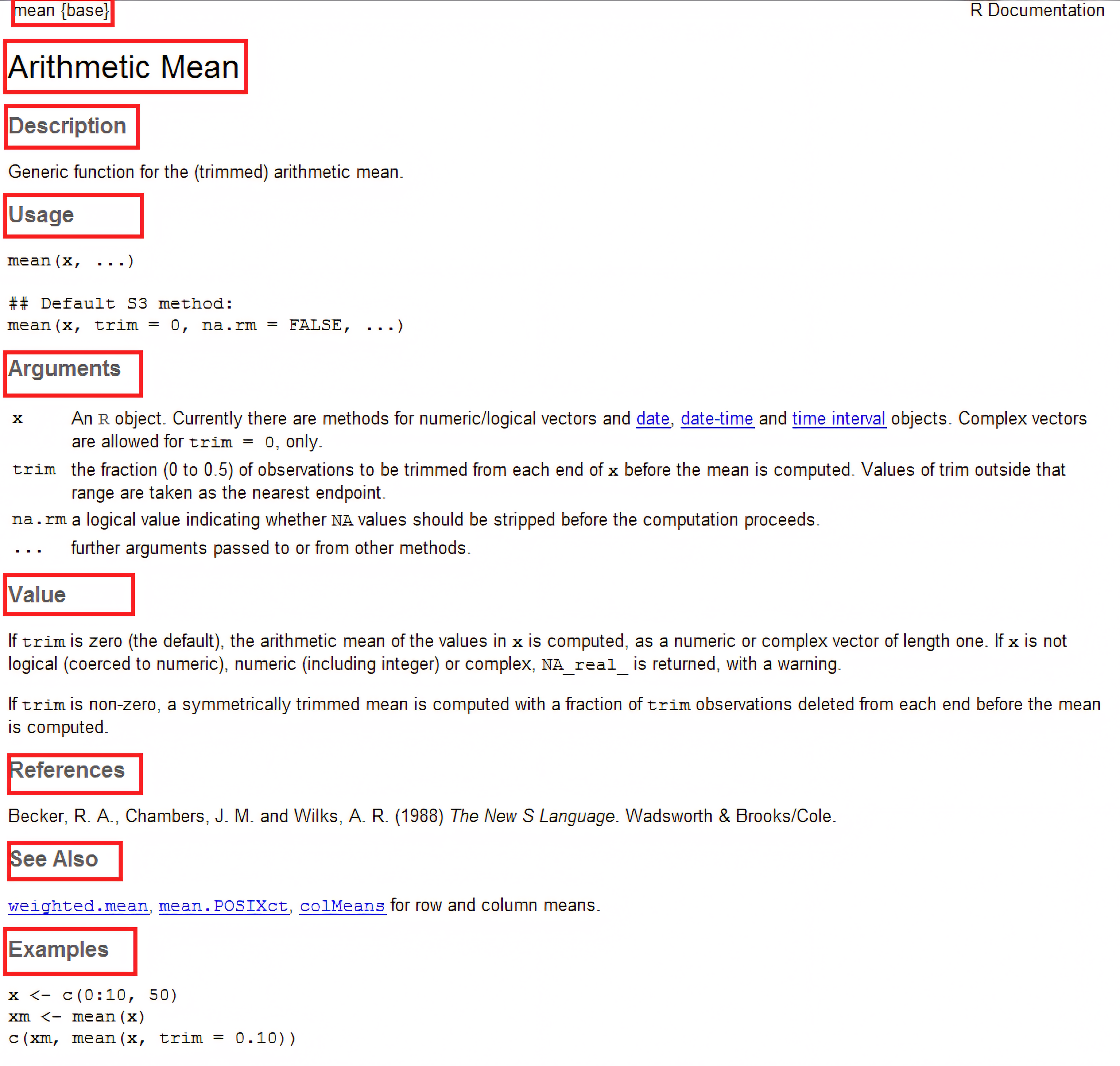
Description: descrição da função
Usage: uso da função e argumentos
Arguments: argumentos e suas especificações
Details: detalhes da função
Value: interpretar a saída (output)
Note: notas sobre a função
Authors: autores da função
References: referências bibliográficas da função
See also: funções relacionadas
Examples: exemplos do uso da função
Pacotes
Conjunto de funções extras para executar tarefas específicas
Pacotes
Duas fontes
- CRAN (Comprehensive R Archive Network)
- GitHub (Repositório de códigos)
Pacotes
Instalação de pacotes
- Download do pacote para o computador (como instalar um software/APP)
- Precisa estar conectado à internet
- O nome do pacote precisa estar entre aspas
- Função (CRAN):
install.packages("pacote")
Instalar o pacote vegan
Verificar pacotes instalados
Pacotes
Carregamento de pacotes
- Carregar o pacote para o R (como abrir software/APP)
- Carrega-se toda vez que se abre o R
- Não precisa estar conectado à internet
- O nome do pacote não precisa estar entre aspas
- Funções:
library(pacote)ourequire(pacote)
Carregar o pacote vegan
Verificar pacotes carregados
Principais erros
Se seu script rodou sem erros, tem algo errado…



Help me help you: um bestiário para entender erros e pedir ajuda no R

Principais erros
1. Esquecer de completar um código (+)
Parênteses
Aspas
::: {.cell}
```{.r .cell-code}
"string
+
```
::: {.cell-output .cell-output-error}
```
Error: <text>:1:1: unexpected INCOMPLETE_STRING
1: "string
2: +
^
```
:::
:::Principais erros
2. Esquecer da vírgula
3. Chamar um objeto errado
Principais erros
4. Esquecer de carregar um pacote
Principais erros
4. Esquecer de carregar um pacote
# carregar o pacote
library(vegan)
## carregar dados
data(dune)
## funcao do pacote vegan
decostand(dune, "hell") Achimill Agrostol Airaprae Alopgeni Anthodor Bellpere Bromhord
1 0.2357023 0.0000000 0.0000000 0.0000000 0.0000000 0.0000000 0.0000000
2 0.2672612 0.0000000 0.0000000 0.2182179 0.0000000 0.2672612 0.3086067
3 0.0000000 0.3162278 0.0000000 0.4183300 0.0000000 0.2236068 0.0000000
4 0.0000000 0.4216370 0.0000000 0.2108185 0.0000000 0.2108185 0.2581989
5 0.2156655 0.0000000 0.0000000 0.0000000 0.3049971 0.2156655 0.2156655
6 0.2041241 0.0000000 0.0000000 0.0000000 0.2500000 0.0000000 0.0000000
7 0.2236068 0.0000000 0.0000000 0.0000000 0.2236068 0.0000000 0.2236068
8 0.0000000 0.3162278 0.0000000 0.3535534 0.0000000 0.0000000 0.0000000
9 0.0000000 0.2672612 0.0000000 0.2672612 0.0000000 0.0000000 0.0000000
10 0.3049971 0.0000000 0.0000000 0.0000000 0.3049971 0.2156655 0.3049971
11 0.0000000 0.0000000 0.0000000 0.0000000 0.0000000 0.0000000 0.0000000
12 0.0000000 0.3380617 0.0000000 0.4780914 0.0000000 0.0000000 0.0000000
13 0.0000000 0.3892495 0.0000000 0.3892495 0.0000000 0.0000000 0.0000000
14 0.0000000 0.4082483 0.0000000 0.0000000 0.0000000 0.0000000 0.0000000
15 0.0000000 0.4170288 0.0000000 0.0000000 0.0000000 0.0000000 0.0000000
16 0.0000000 0.4605662 0.0000000 0.3481553 0.0000000 0.0000000 0.0000000
17 0.3651484 0.0000000 0.3651484 0.0000000 0.5163978 0.0000000 0.0000000
18 0.0000000 0.0000000 0.0000000 0.0000000 0.0000000 0.2721655 0.0000000
19 0.0000000 0.0000000 0.3110855 0.0000000 0.3592106 0.0000000 0.0000000
20 0.0000000 0.4016097 0.0000000 0.0000000 0.0000000 0.0000000 0.0000000
Chenalbu Cirsarve Comapalu Eleopalu Elymrepe Empenigr Hyporadi
1 0.0000000 0.0000000 0.0000000 0.0000000 0.4714045 0.0000000 0.0000000
2 0.0000000 0.0000000 0.0000000 0.0000000 0.3086067 0.0000000 0.0000000
3 0.0000000 0.0000000 0.0000000 0.0000000 0.3162278 0.0000000 0.0000000
4 0.0000000 0.2108185 0.0000000 0.0000000 0.2981424 0.0000000 0.0000000
5 0.0000000 0.0000000 0.0000000 0.0000000 0.3049971 0.0000000 0.0000000
6 0.0000000 0.0000000 0.0000000 0.0000000 0.0000000 0.0000000 0.0000000
7 0.0000000 0.0000000 0.0000000 0.0000000 0.0000000 0.0000000 0.0000000
8 0.0000000 0.0000000 0.0000000 0.3162278 0.0000000 0.0000000 0.0000000
9 0.0000000 0.0000000 0.0000000 0.0000000 0.3779645 0.0000000 0.0000000
10 0.0000000 0.0000000 0.0000000 0.0000000 0.0000000 0.0000000 0.0000000
11 0.0000000 0.0000000 0.0000000 0.0000000 0.0000000 0.0000000 0.2500000
12 0.0000000 0.0000000 0.0000000 0.0000000 0.0000000 0.0000000 0.0000000
13 0.1740777 0.0000000 0.0000000 0.0000000 0.0000000 0.0000000 0.0000000
14 0.0000000 0.0000000 0.2886751 0.4082483 0.0000000 0.0000000 0.0000000
15 0.0000000 0.0000000 0.2948839 0.4662524 0.0000000 0.0000000 0.0000000
16 0.0000000 0.0000000 0.0000000 0.4923660 0.0000000 0.0000000 0.0000000
17 0.0000000 0.0000000 0.0000000 0.0000000 0.0000000 0.0000000 0.3651484
18 0.0000000 0.0000000 0.0000000 0.0000000 0.0000000 0.0000000 0.0000000
19 0.0000000 0.0000000 0.0000000 0.0000000 0.0000000 0.2540003 0.4016097
20 0.0000000 0.0000000 0.0000000 0.3592106 0.0000000 0.0000000 0.0000000
Juncarti Juncbufo Lolipere Planlanc Poaprat Poatriv Ranuflam
1 0.0000000 0.0000000 0.6236096 0.0000000 0.4714045 0.3333333 0.0000000
2 0.0000000 0.0000000 0.3450328 0.0000000 0.3086067 0.4082483 0.0000000
3 0.0000000 0.0000000 0.3872983 0.0000000 0.3535534 0.3872983 0.0000000
4 0.0000000 0.0000000 0.3333333 0.0000000 0.2981424 0.3333333 0.0000000
5 0.0000000 0.0000000 0.2156655 0.3409972 0.2156655 0.3735437 0.0000000
6 0.0000000 0.0000000 0.3535534 0.3227486 0.2500000 0.2886751 0.0000000
7 0.0000000 0.2236068 0.3872983 0.3535534 0.3162278 0.3535534 0.0000000
8 0.3162278 0.0000000 0.3162278 0.0000000 0.3162278 0.3162278 0.2236068
9 0.3086067 0.3086067 0.2182179 0.0000000 0.3086067 0.3450328 0.0000000
10 0.0000000 0.0000000 0.3735437 0.2641353 0.3049971 0.3049971 0.0000000
11 0.0000000 0.0000000 0.4677072 0.3061862 0.3535534 0.0000000 0.0000000
12 0.0000000 0.3380617 0.0000000 0.0000000 0.0000000 0.3380617 0.0000000
13 0.0000000 0.3015113 0.0000000 0.0000000 0.2461830 0.5222330 0.2461830
14 0.0000000 0.0000000 0.0000000 0.0000000 0.0000000 0.0000000 0.2886751
15 0.3611576 0.0000000 0.0000000 0.0000000 0.0000000 0.0000000 0.2948839
16 0.3015113 0.0000000 0.0000000 0.0000000 0.0000000 0.2461830 0.2461830
17 0.0000000 0.0000000 0.0000000 0.3651484 0.2581989 0.0000000 0.0000000
18 0.0000000 0.0000000 0.2721655 0.3333333 0.3333333 0.0000000 0.0000000
19 0.0000000 0.0000000 0.0000000 0.0000000 0.0000000 0.0000000 0.0000000
20 0.3592106 0.0000000 0.0000000 0.0000000 0.0000000 0.0000000 0.3592106
Rumeacet Sagiproc Salirepe Scorautu Trifprat Trifrepe Vicilath
1 0.0000000 0.0000000 0.0000000 0.0000000 0.0000000 0.0000000 0.0000000
2 0.0000000 0.0000000 0.0000000 0.3450328 0.0000000 0.3450328 0.0000000
3 0.0000000 0.0000000 0.0000000 0.2236068 0.0000000 0.2236068 0.0000000
4 0.0000000 0.3333333 0.0000000 0.2108185 0.0000000 0.1490712 0.0000000
5 0.3409972 0.0000000 0.0000000 0.2641353 0.2156655 0.2156655 0.0000000
6 0.3535534 0.0000000 0.0000000 0.2500000 0.3227486 0.3227486 0.0000000
7 0.2738613 0.0000000 0.0000000 0.2738613 0.2236068 0.2236068 0.0000000
8 0.0000000 0.2236068 0.0000000 0.2738613 0.0000000 0.2236068 0.0000000
9 0.2182179 0.2182179 0.0000000 0.2182179 0.0000000 0.2672612 0.0000000
10 0.0000000 0.0000000 0.0000000 0.2641353 0.0000000 0.3735437 0.1524986
11 0.0000000 0.2500000 0.0000000 0.3952847 0.0000000 0.3061862 0.2500000
12 0.2390457 0.3380617 0.0000000 0.2390457 0.0000000 0.2927700 0.0000000
13 0.0000000 0.2461830 0.0000000 0.2461830 0.0000000 0.2461830 0.0000000
14 0.0000000 0.0000000 0.0000000 0.2886751 0.0000000 0.5000000 0.0000000
15 0.0000000 0.0000000 0.0000000 0.2948839 0.0000000 0.2085144 0.0000000
16 0.0000000 0.0000000 0.0000000 0.0000000 0.0000000 0.0000000 0.0000000
17 0.0000000 0.0000000 0.0000000 0.3651484 0.0000000 0.0000000 0.0000000
18 0.0000000 0.0000000 0.3333333 0.4303315 0.0000000 0.2721655 0.1924501
19 0.0000000 0.3110855 0.3110855 0.4399413 0.0000000 0.2540003 0.0000000
20 0.0000000 0.0000000 0.4016097 0.2540003 0.0000000 0.0000000 0.0000000
Bracruta Callcusp
1 0.0000000 0.0000000
2 0.0000000 0.0000000
3 0.2236068 0.0000000
4 0.2108185 0.0000000
5 0.2156655 0.0000000
6 0.3535534 0.0000000
7 0.2236068 0.0000000
8 0.2236068 0.0000000
9 0.2182179 0.0000000
10 0.2156655 0.0000000
11 0.3535534 0.0000000
12 0.3380617 0.0000000
13 0.0000000 0.0000000
14 0.0000000 0.4082483
15 0.4170288 0.0000000
16 0.3481553 0.3015113
17 0.0000000 0.0000000
18 0.4714045 0.0000000
19 0.3110855 0.0000000
20 0.3592106 0.3110855Principais erros
5. Usar o nome da função de forma errônea
Achimill Agrostol Airaprae Alopgeni Anthodor Bellpere Bromhord Chenalbu
16 48 5 36 21 13 15 1
Cirsarve Comapalu Eleopalu Elymrepe Empenigr Hyporadi Juncarti Juncbufo
2 4 25 26 2 9 18 13
Lolipere Planlanc Poaprat Poatriv Ranuflam Rumeacet Sagiproc Salirepe
58 26 48 63 14 18 20 11
Scorautu Trifprat Trifrepe Vicilath Bracruta Callcusp
54 9 47 4 49 10 Dúvidas?
2. Análise exploratória de dados (AED)
Dados
| id | especie | peso | comp_corpo | comp_asa | … |
|---|---|---|---|---|---|
| LECAVE035 | Columbina talpacoti | 40.64 | 158.5 | 90.51 | … |
| LECAVE036 | Columbina talpacoti | 41.05 | 149.73 | 89.42 | … |
| LECAVE037 | Columbina talpacoti | 43.96 | 147.62 | 89.32 | … |
| LECAVE038 | Columbina talpacoti | 48.01 | 153.44 | 90.57 | … |
| … | … | … | … | … | … |
Variáveis
Cada coluna dos nossos dados é uma variável (valores variam)
Rows: 16
Columns: 8
$ id <chr> "LECAVE040", "LECAVE041", "LECAVE043", "LECAVE044", "LECAVE…
$ especie <chr> "Thraupis sayaca", "Thraupis sayaca", "Thraupis sayaca", "T…
$ peso <dbl> 22.77, 18.22, 24.09, 17.34, 20.75, 26.90, 23.90, 26.80, 32.…
$ comp_corpo <dbl> 146.58, 139.79, 120.63, 133.33, 139.80, 140.45, 136.39, 119…
$ comp_bico <dbl> 16.57, 17.14, 19.06, 18.49, 15.68, 9.69, 14.94, 11.13, 13.0…
$ larg_bico <dbl> 6.96, 6.94, 6.64, 7.11, 7.13, 7.75, 6.59, 6.55, 6.77, 8.40,…
$ alt_bico <dbl> 6.60, 8.51, 8.23, 6.97, 7.12, 5.92, 7.12, 7.30, 7.35, 7.95,…
$ sexo <chr> "F", "F", "F", "F", "F", "M", "F", "M", "M", "F", "M", "M",…Medidas-resumo
Medidas de posição
Moda: valor mais frequente das observações
Mediana: valor que divide as observações ordenadas em duas partes iguais
Média aritmética: soma das observações dividida pelo número de observações
Outras médias: ponderada, geométrica e harmônica
Medidas-resumo
Medidas de dispersão
Variância: medida de quantas observações diferem do valor central (média)
Desvio padrão: raiz quadrada da variância (mantêm a mesma unidade de medida)
Erro padrão: variação da média amostral em relação à média populacional (confiabilidade da média amostral)
Tabelas
Tabelas de frequência
Dados brutos: conjunto dos valores numéricos coletados na pesquisa
Classes: intervalo de valores contínuos (0 |— 1)
Frequência absoluta (fi): número de vezes que uma observação aparece ou pertencente a um intervalo ou classe
Frequência total: soma de todas as frequências absolutas (fi)
Frequência relativa (fri): valor da razão de cada frequência absoluta (fi) com a frequência total (somatório de fi)
Frequência acumulada (Fi): obtida somando (acumulando) os valores da frequência absoluta (fi)
Frequência relativa acumulada (Fri): obtida somando (acumulando) os valores da frequência relativa (fri)
Tabelas
Tabelas de frequência
| Classes | Freq. absoluta (fi) | Freq. relativa (fri) | Freq. acumulada (Fi) | Freq. relativa acumulada (Fri) |
|---|---|---|---|---|
| 0|—1 | 4 | 0.13 | 4 | 0.13 |
| 1|—2 | 8 | 0.26 | 12 | 0.39 |
| 2|—3 | 12 | 0.39 | 24 | 0.77 |
| 3|—4 | 7 | 0.23 | 31 | 1.00 |
| Total | 31 | 1.00 |
Gráficos
Melhor forma de apresentar, sintetizar, discutir e interpretar seus dados
Necessário em quase todas as análises estatísticas
Necessário em quase todas as publicações, trabalhos de consultoria, TCC, dissertação, tese, etc.
Existem vários tipos de gráficos para representar os padrões em seus dados para diferentes tipos de finalidades
De forma simplificada, os gráficos são representações dos nossos dados tabulares
Como mentir com estatística?

Elementos de um gráfico
Representações das colunas (eixos) e linhas (elementos)
Tipos de variáveis
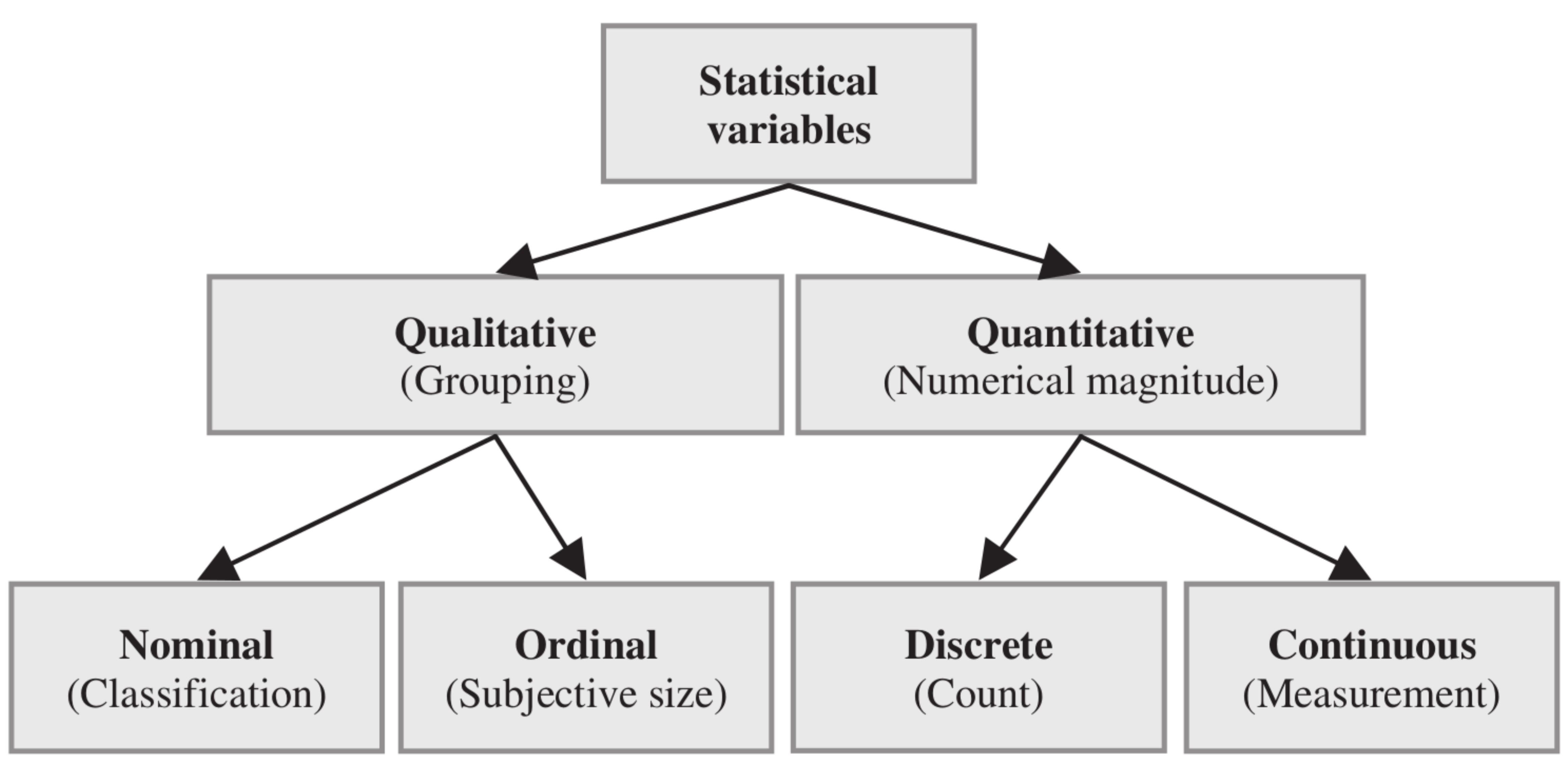
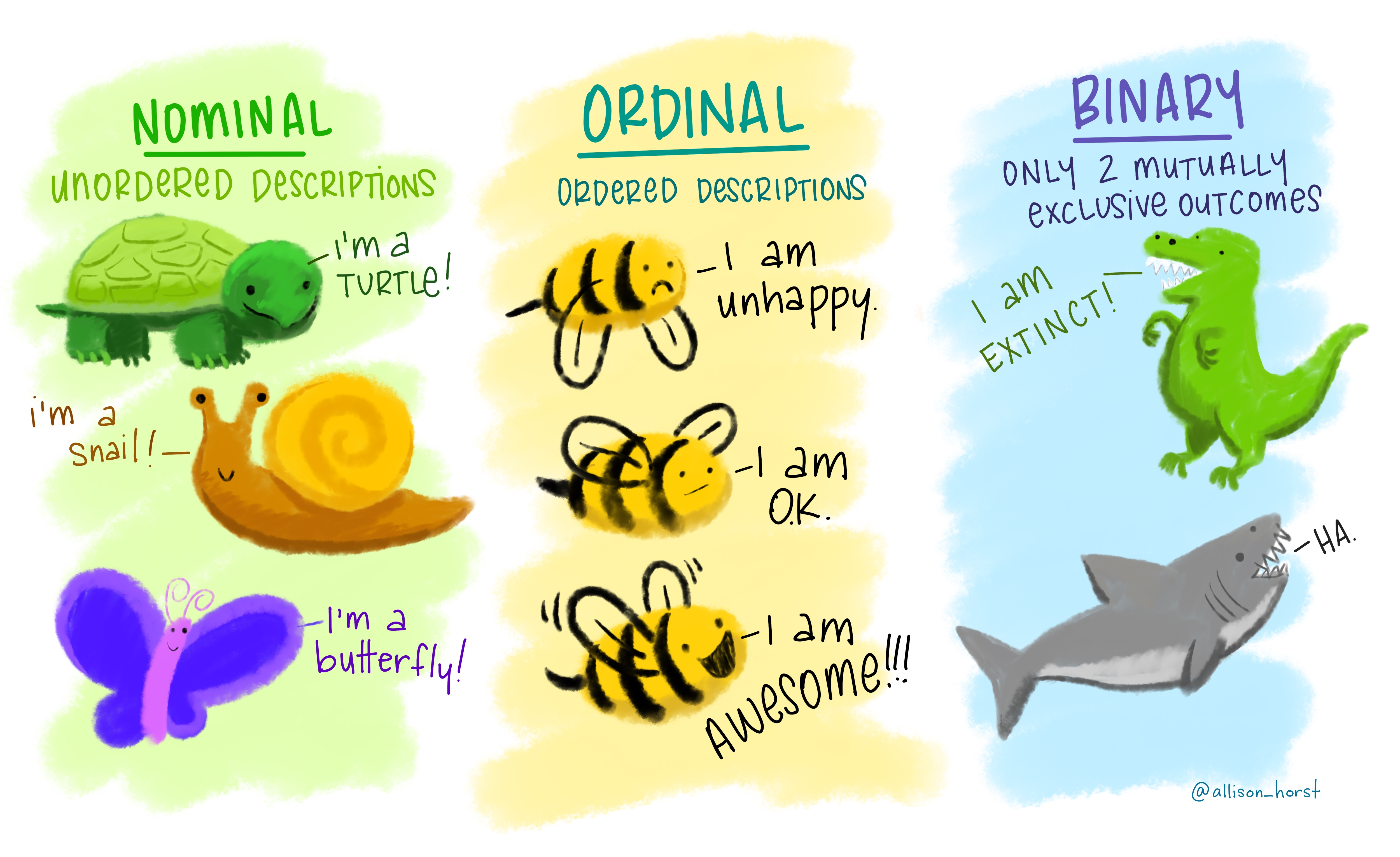

Tipos de variáveis e tipos gráficos
Tipos de variáveis e tipos gráficos
R CHARTS
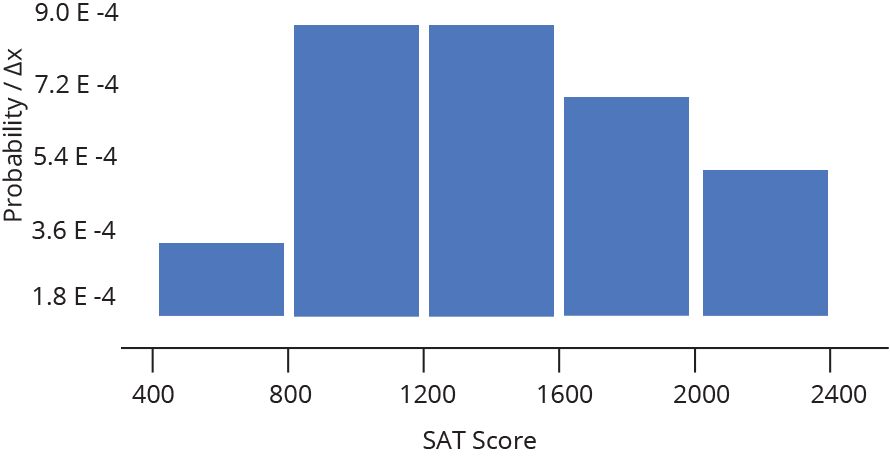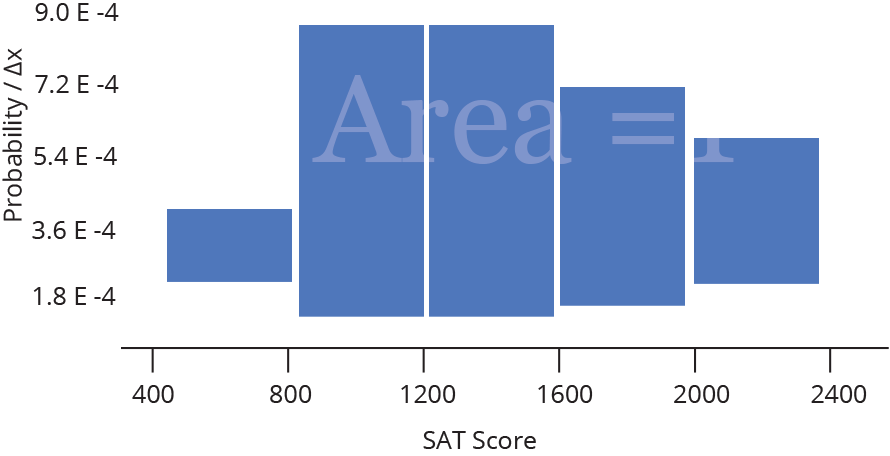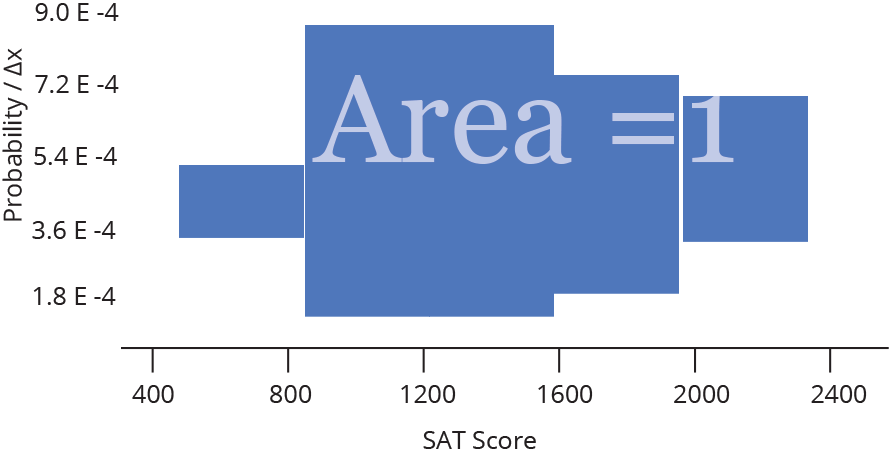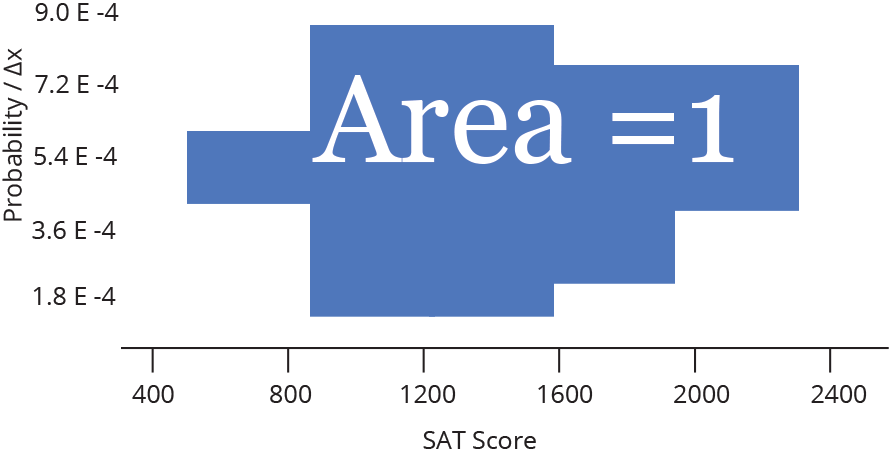
Histograma ou densidade
Representa dados de uma coluna
Dados do tipo discreto ou contínuo
Distribuição de frequência ou densidade
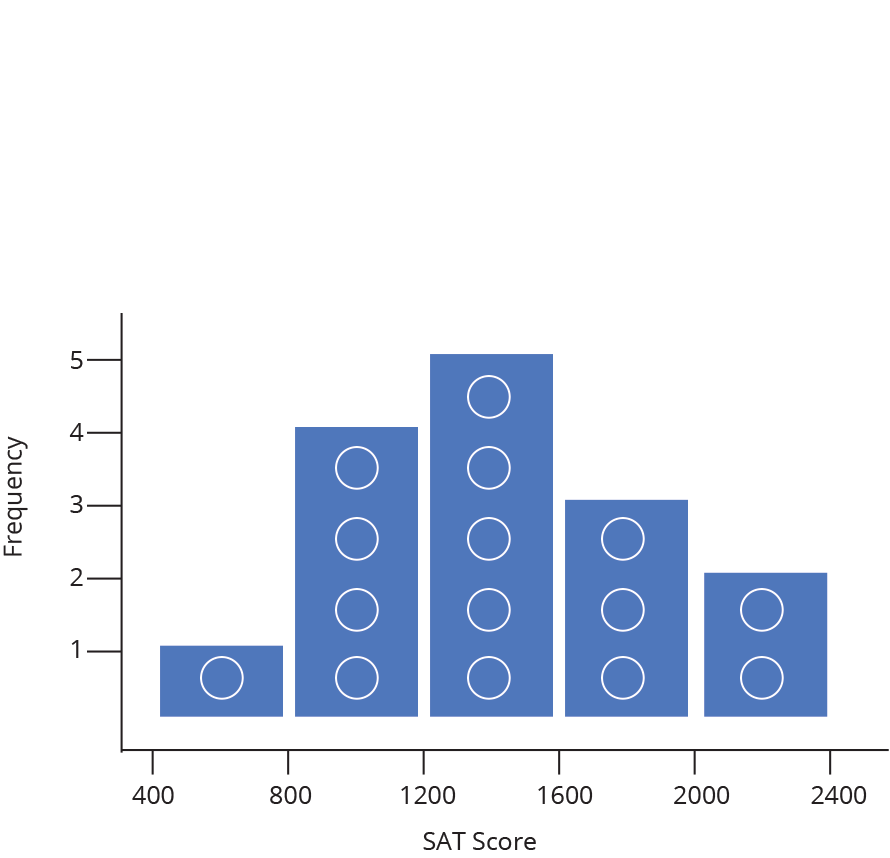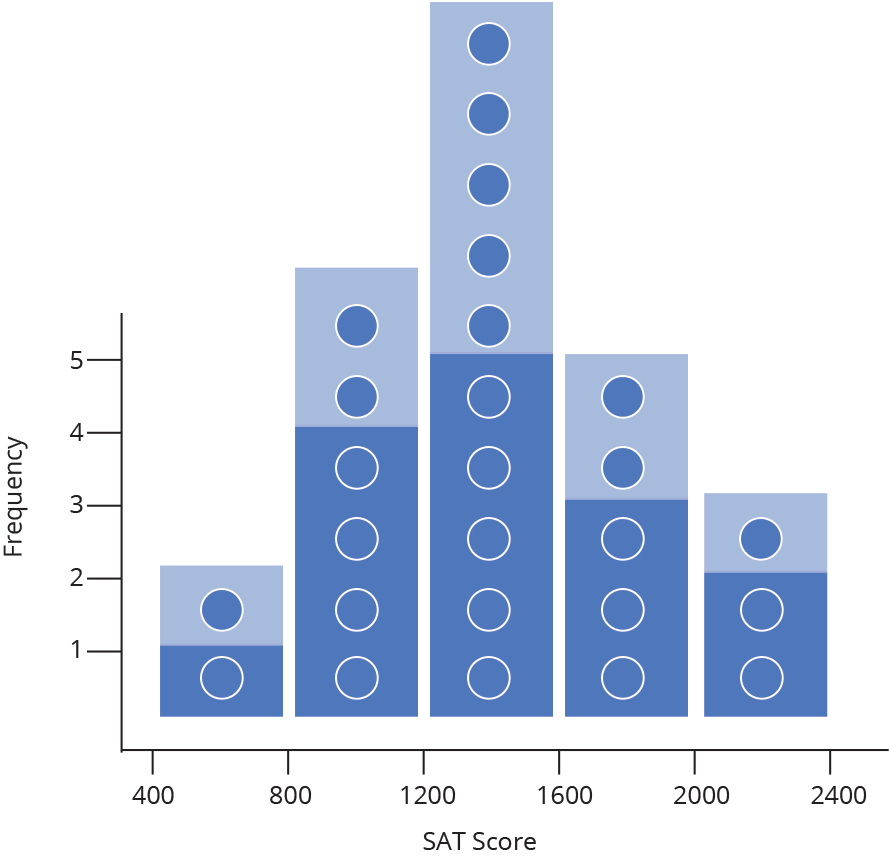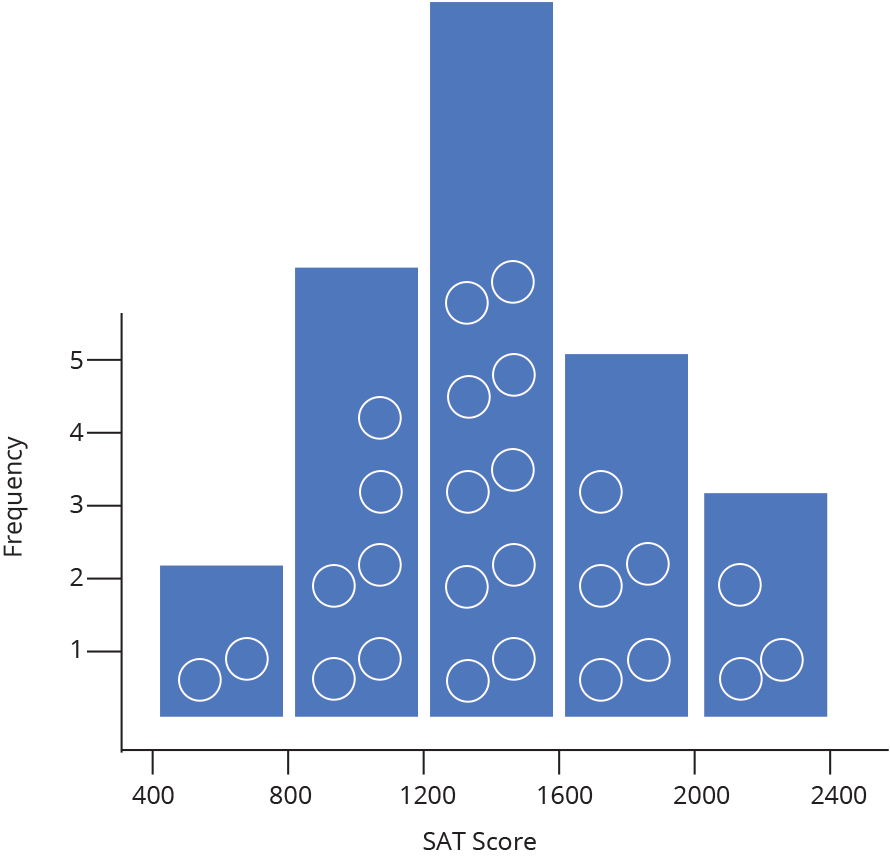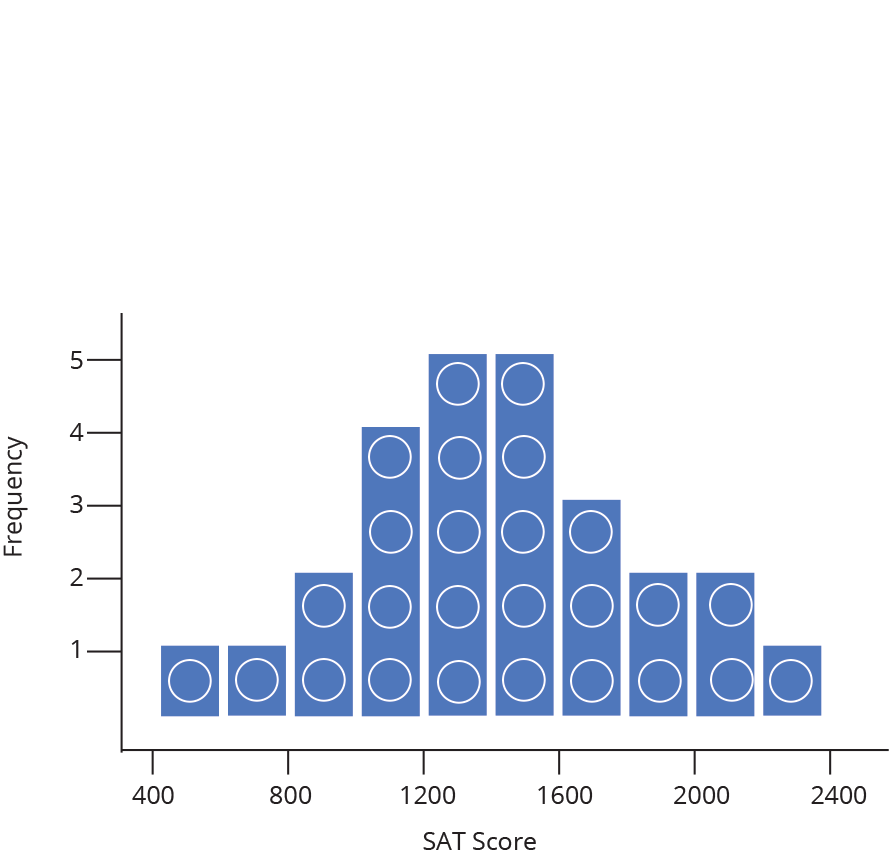
Histograma

Histograma
Densidade
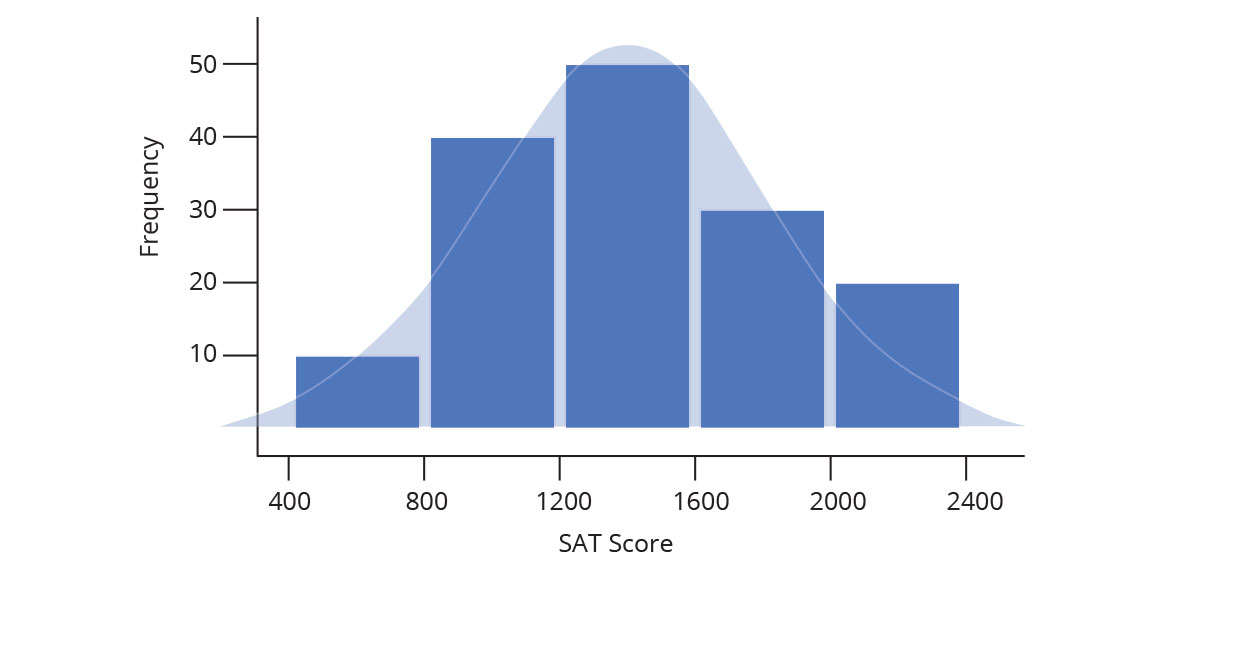
Densidade
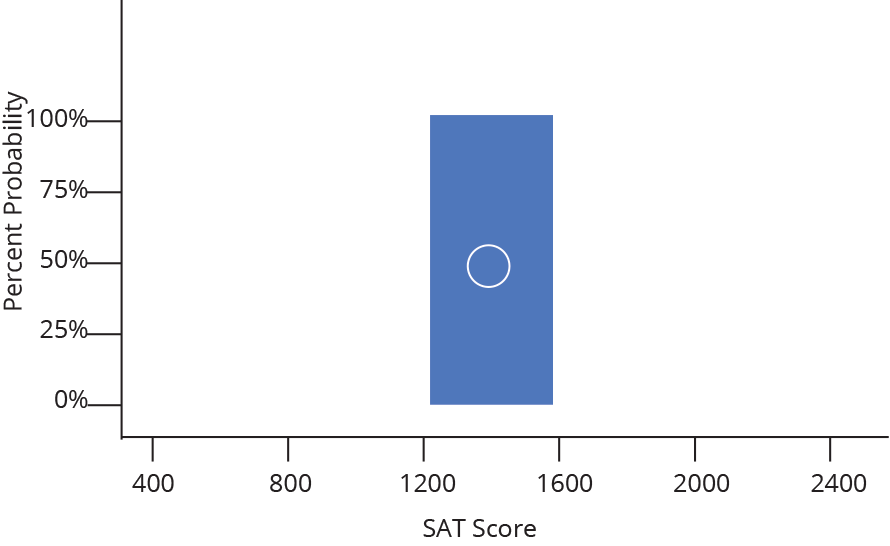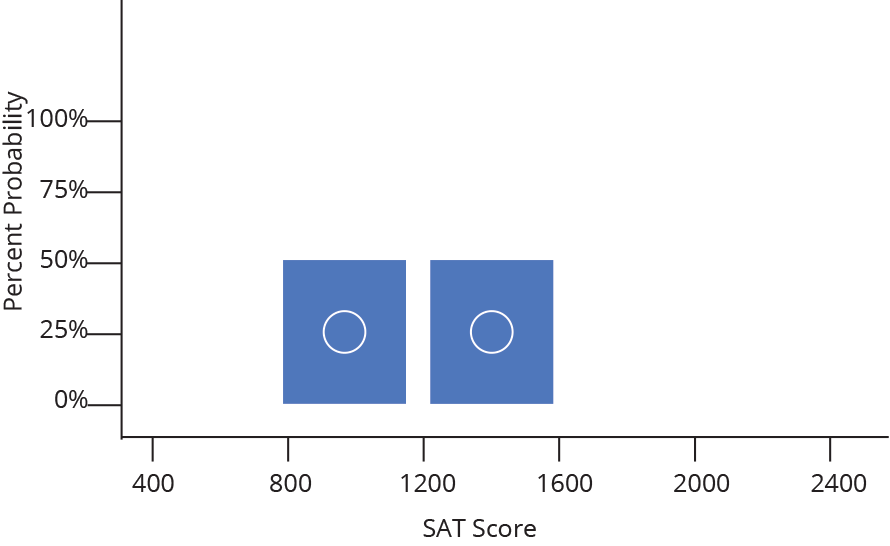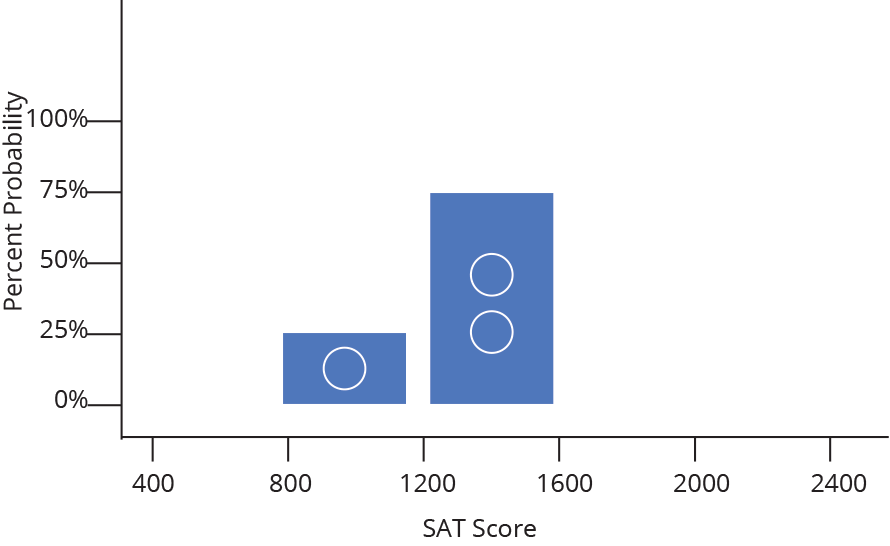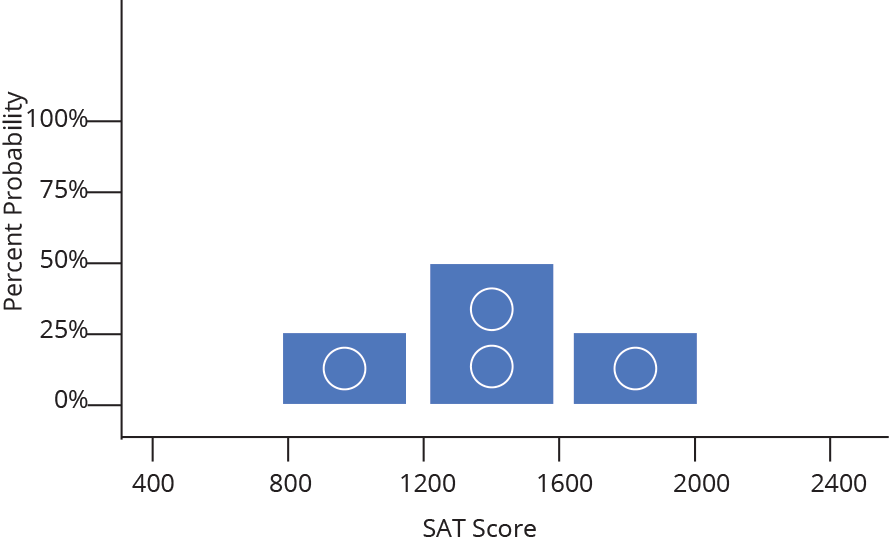
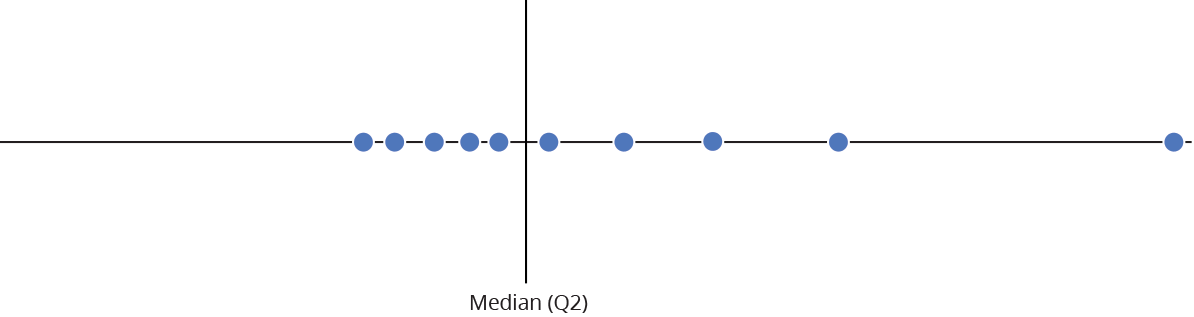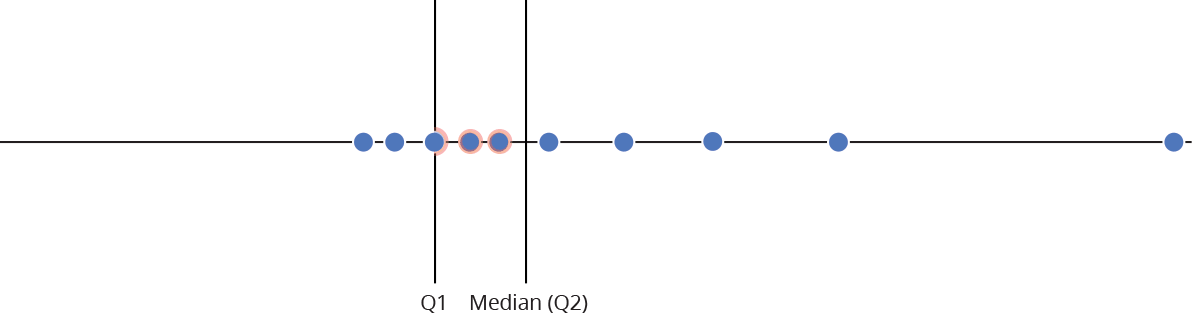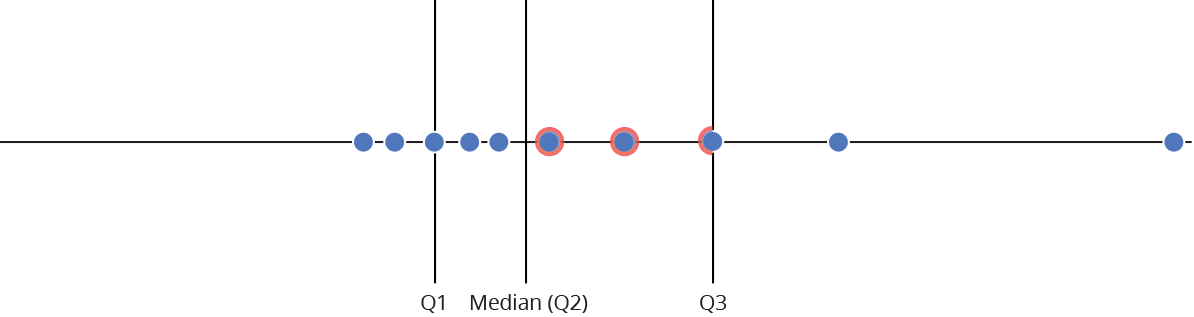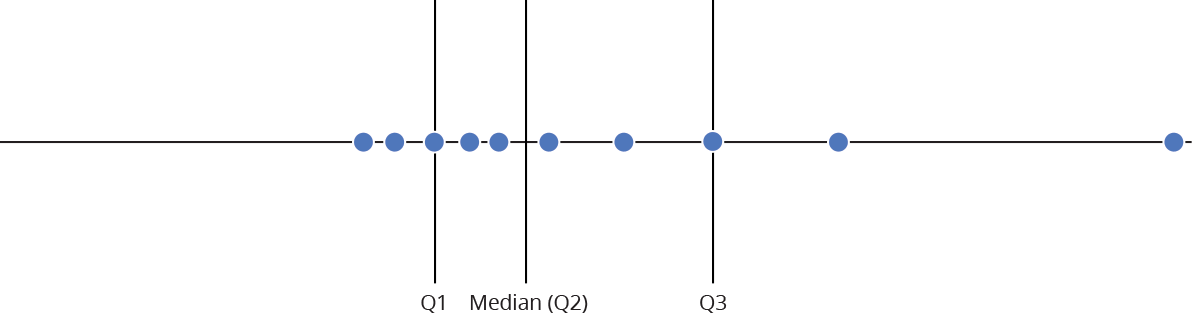
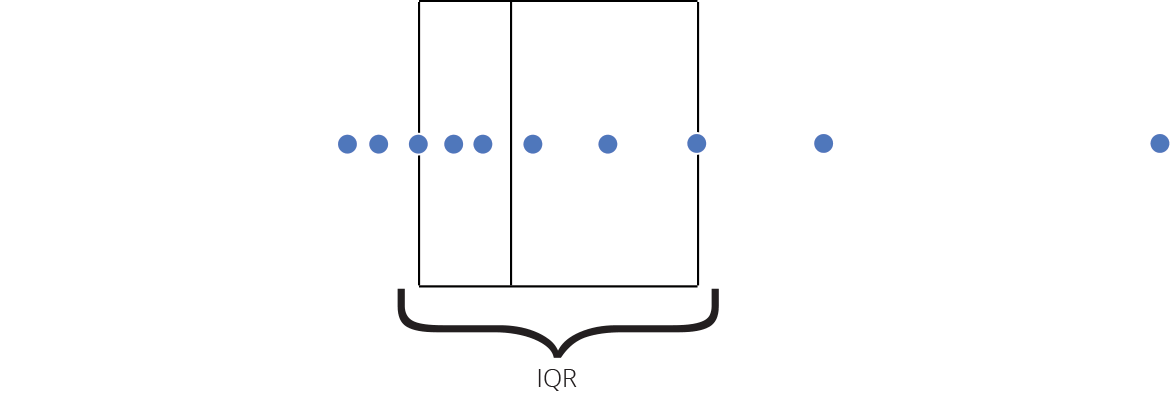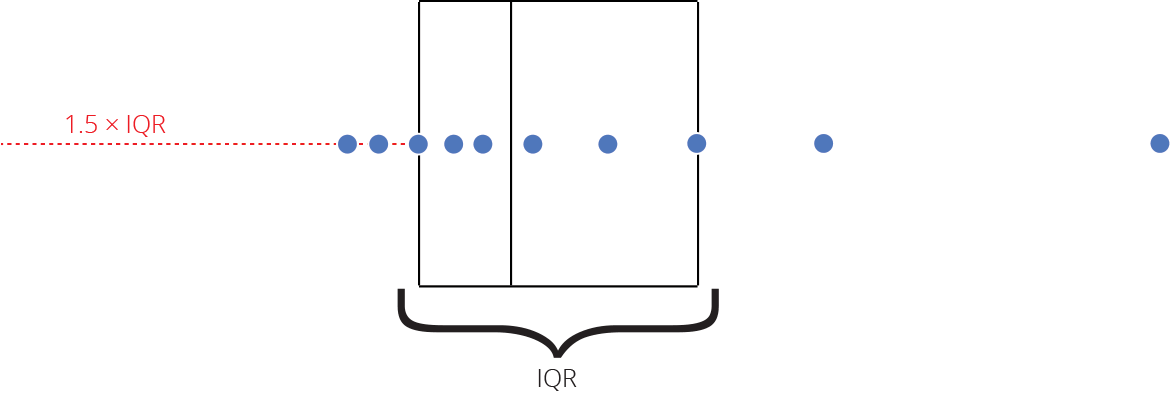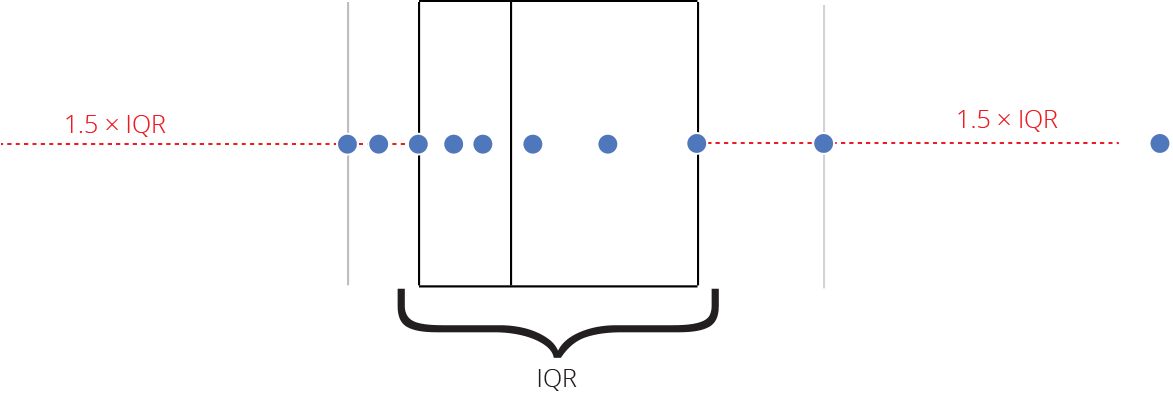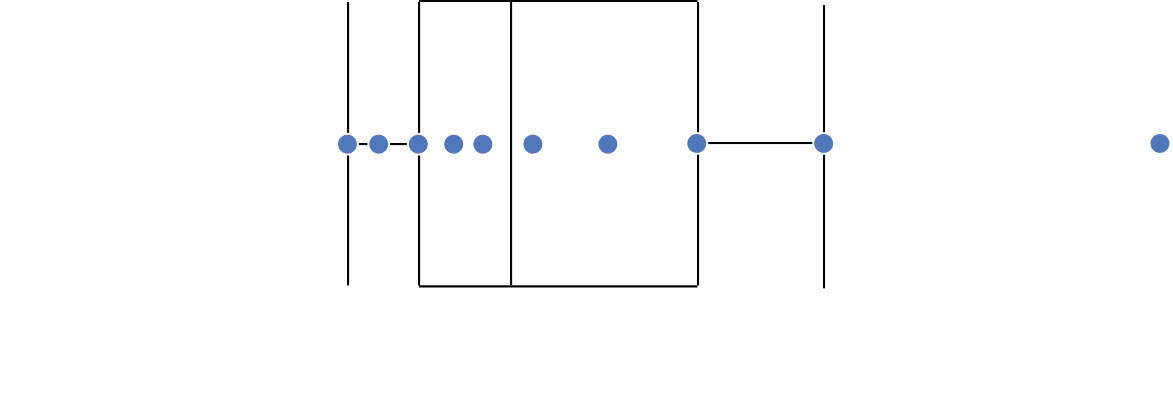
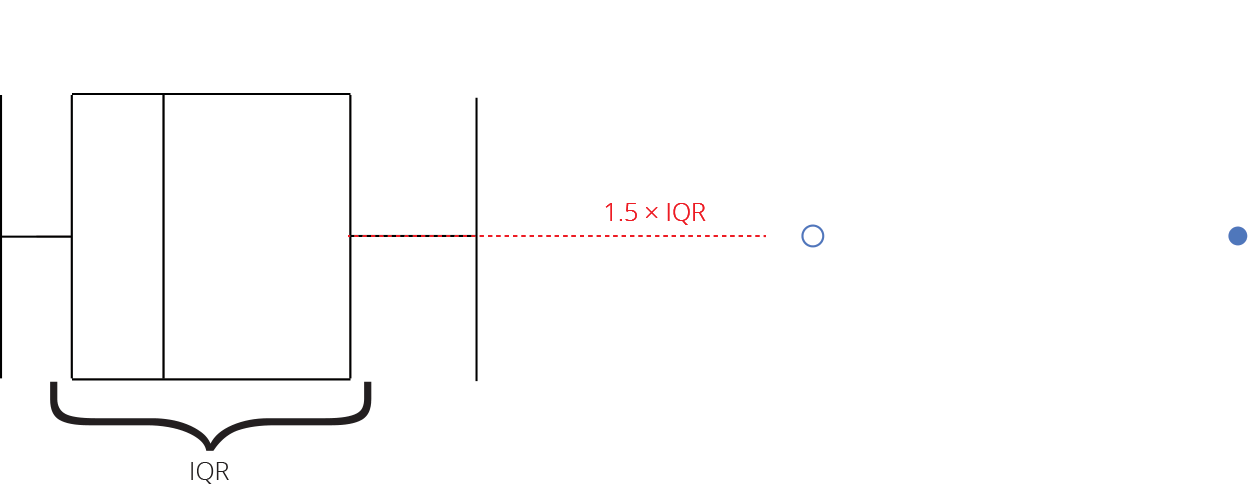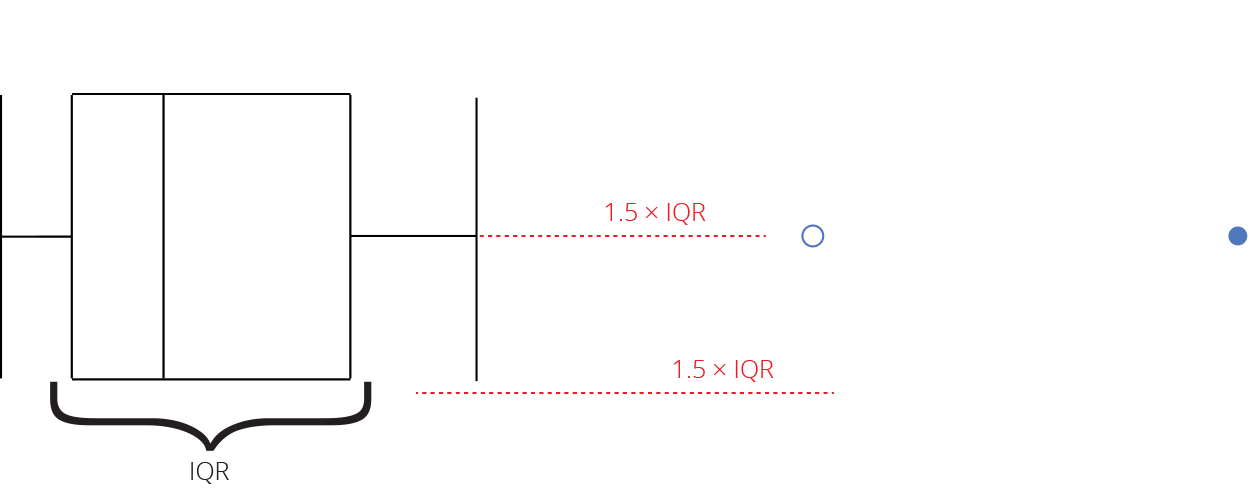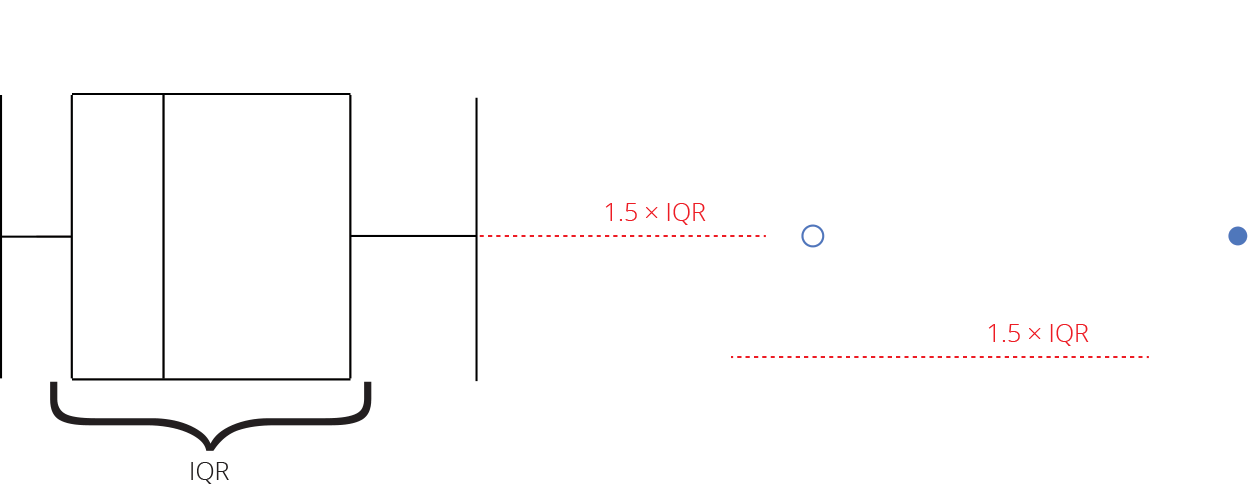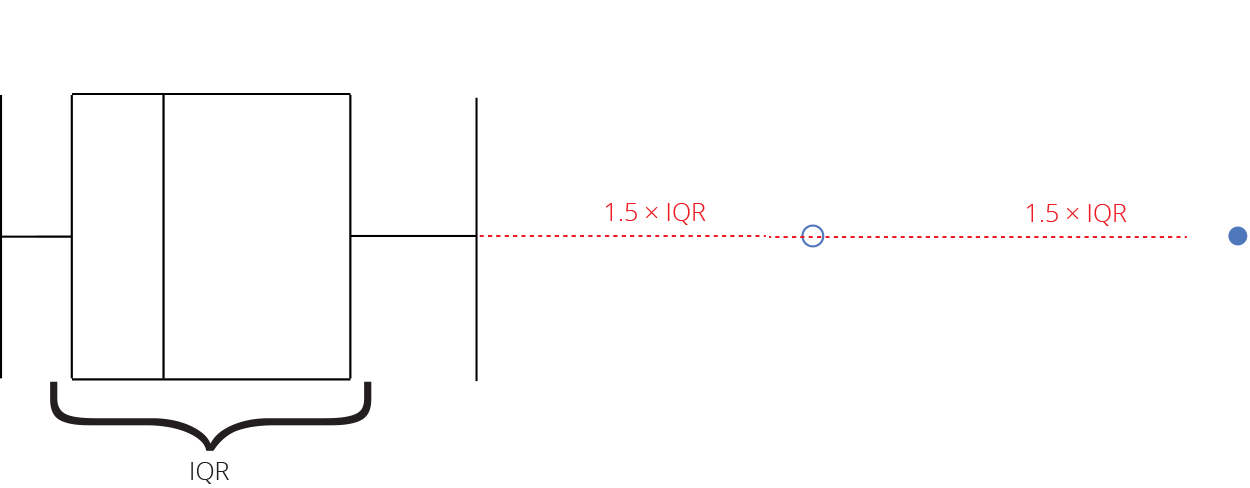
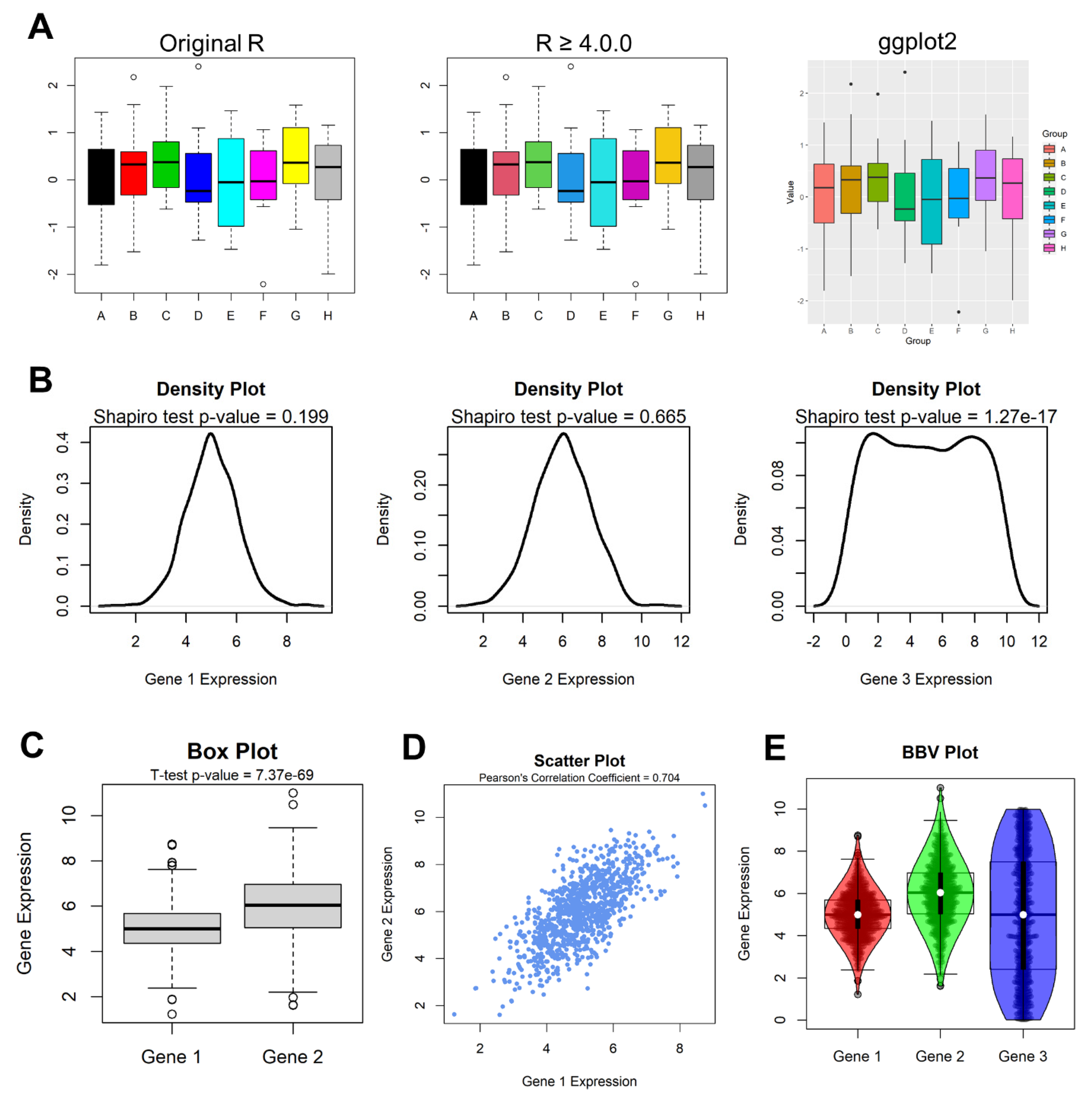
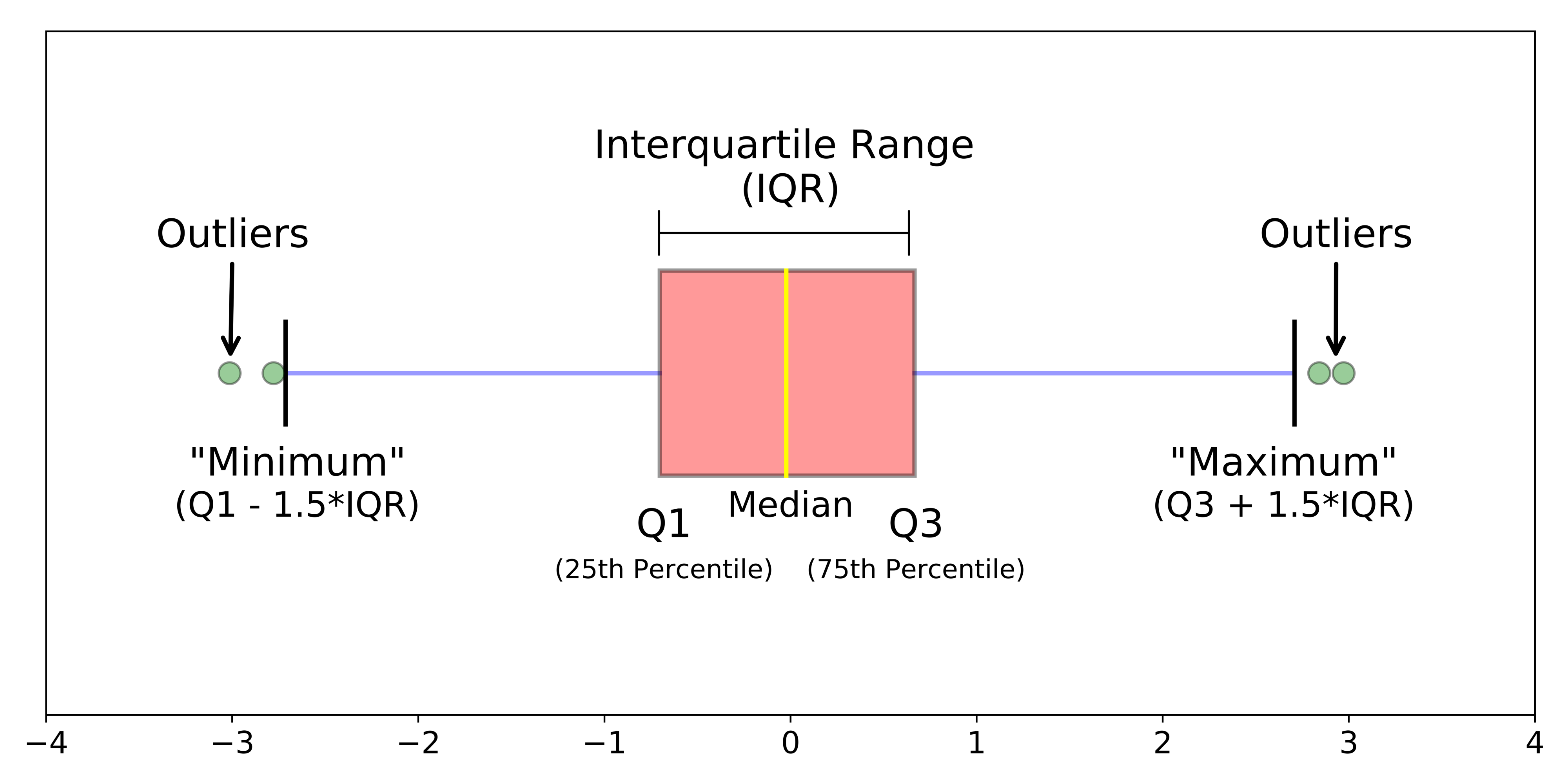
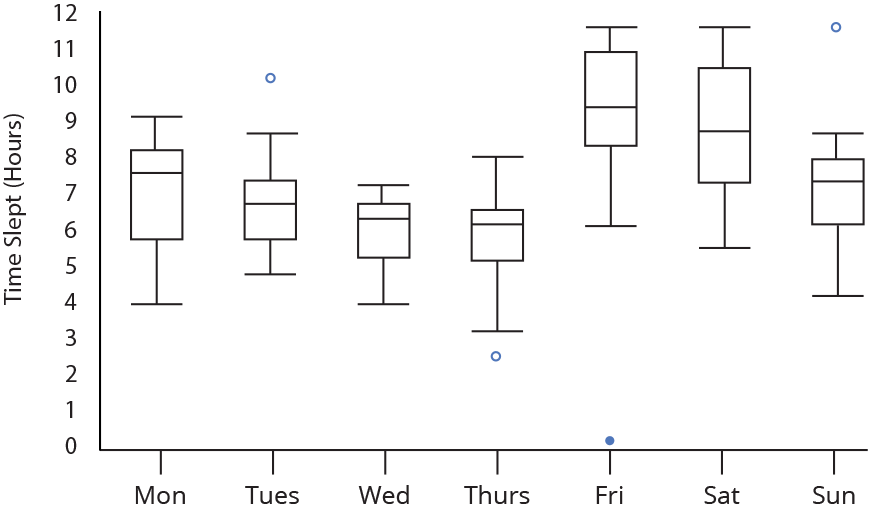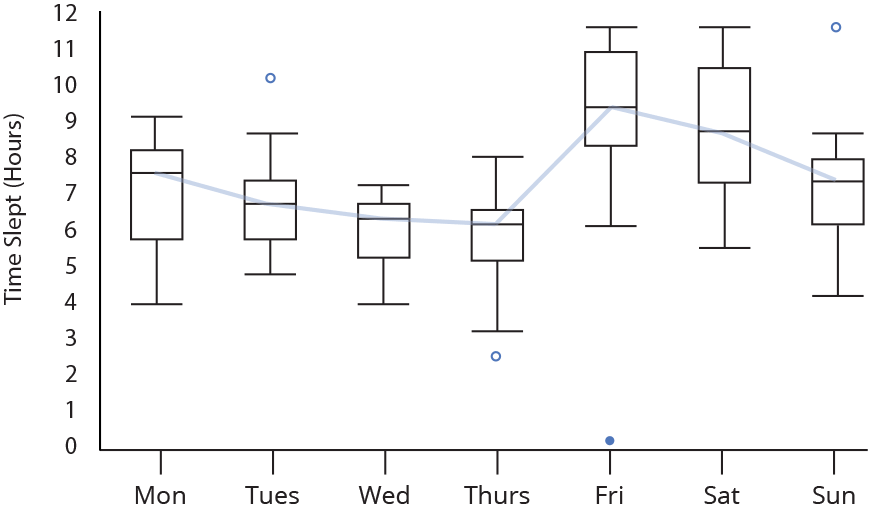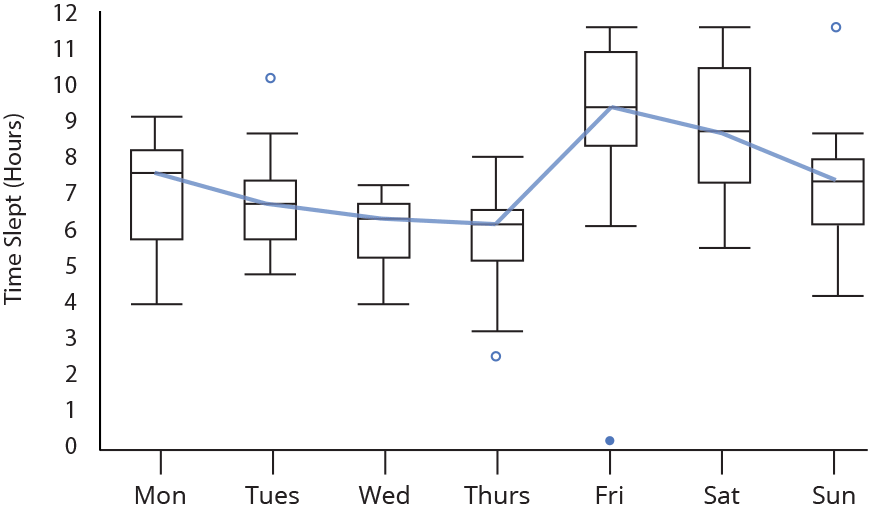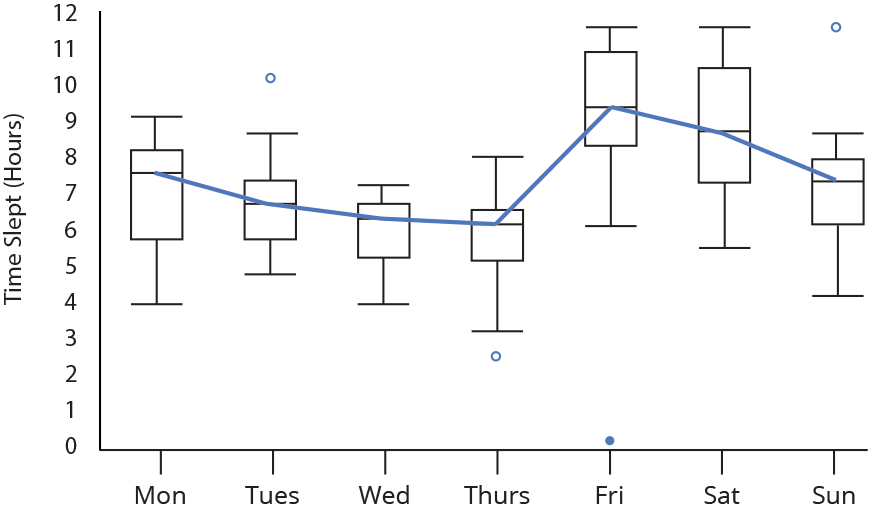
Gráfico de caixas (Box plot)
Representa dados de duas colunas
Dados do tipo categóricos: X = categórico e Y = contínuo
Resume informações de medidas contínuas para dois ou mais fatores categóricos
Gráfico de caixas (Box plot)
Intervalo inter-quartil (interquartile range - IQR)
Limite inferior e limite superipor (1.5 x IQR)
Valores exteriores (outliers)
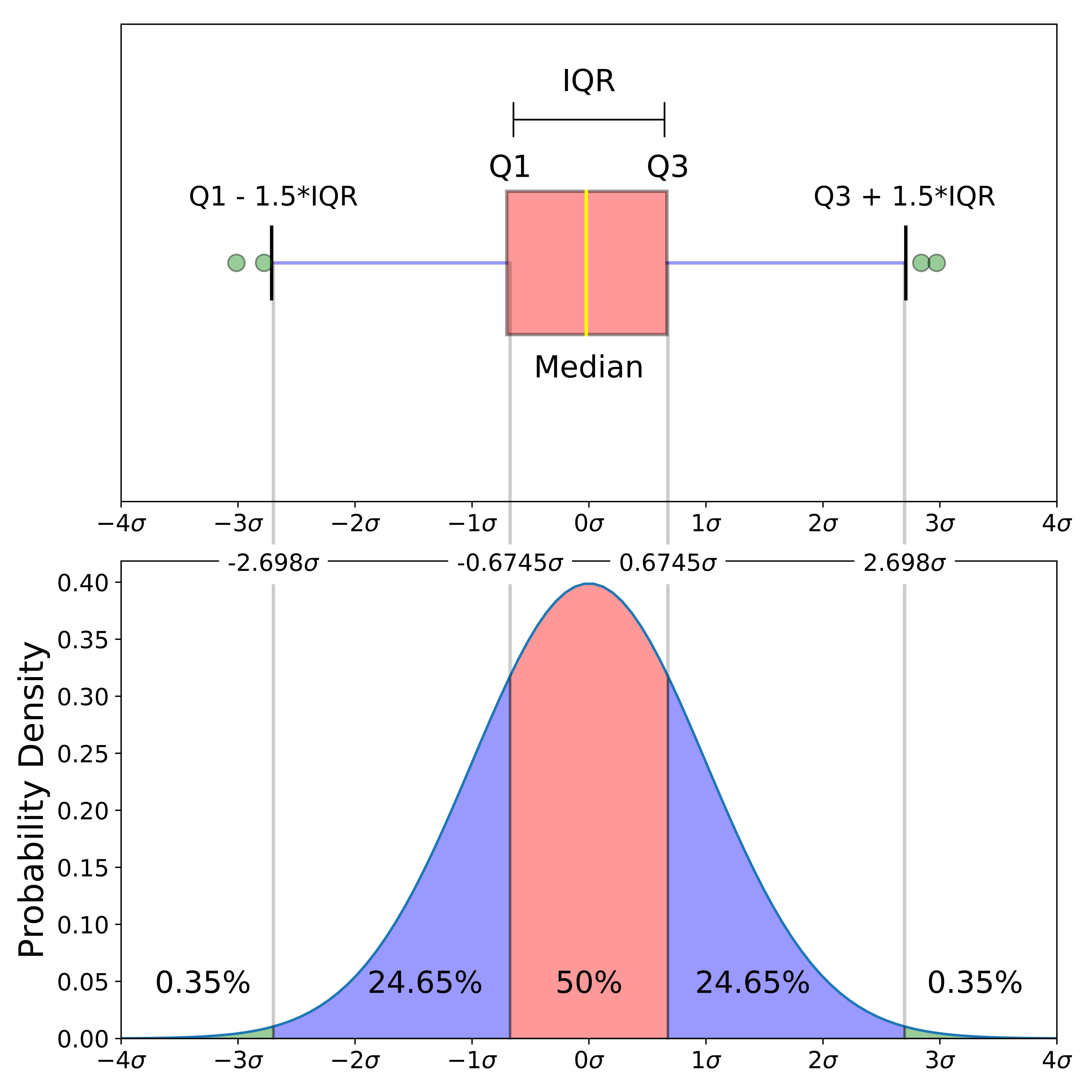
Outlier
Observação coletado de forma errônea ou devido à arredondamentos

Gráfico de caixas (Box plot)
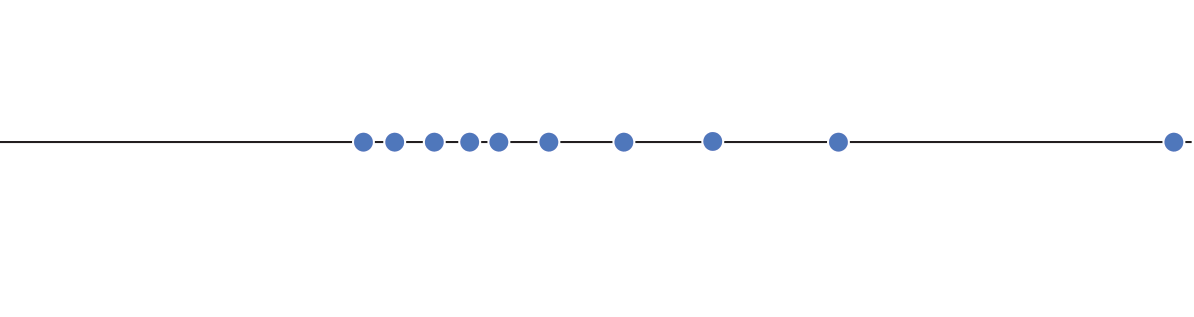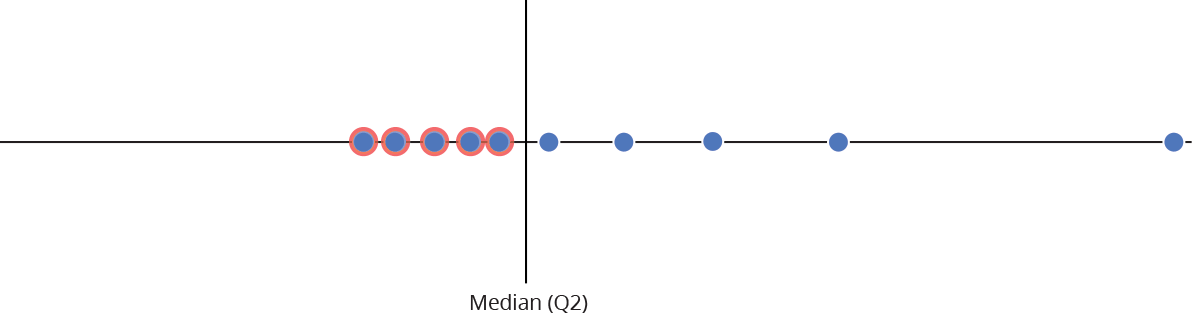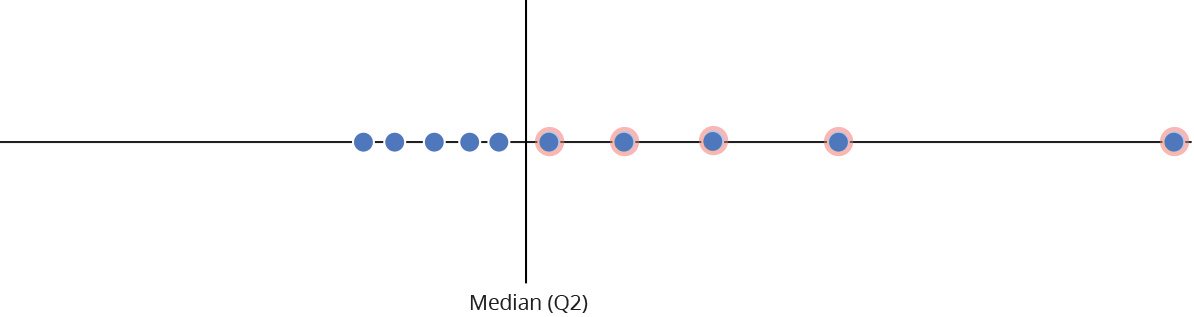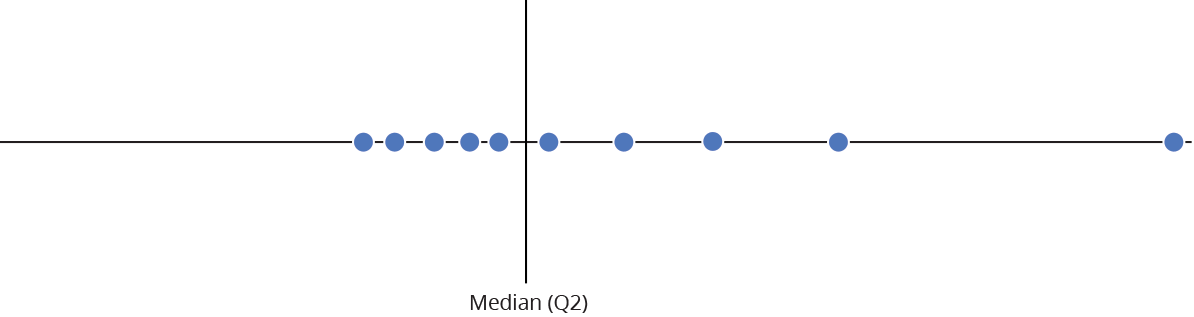
Gráfico de caixas (Box plot)
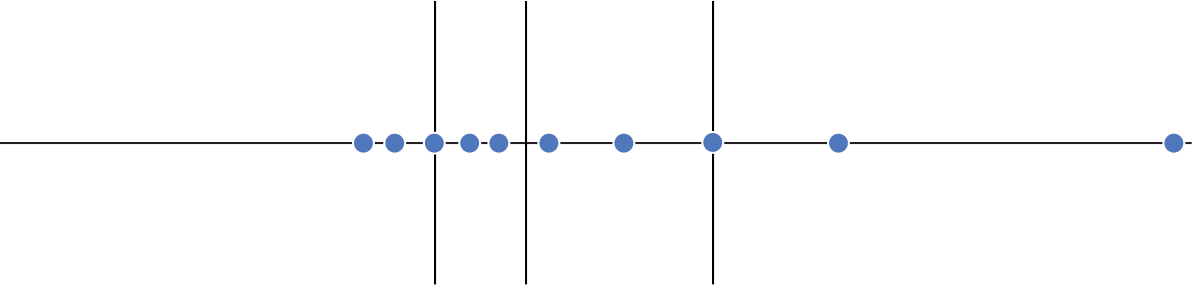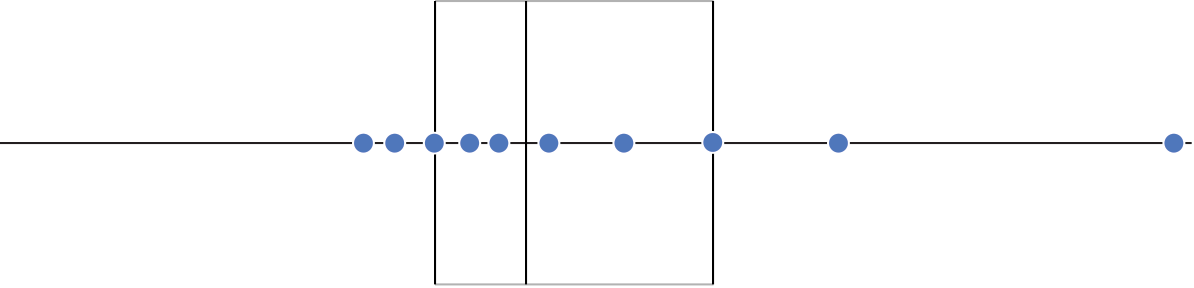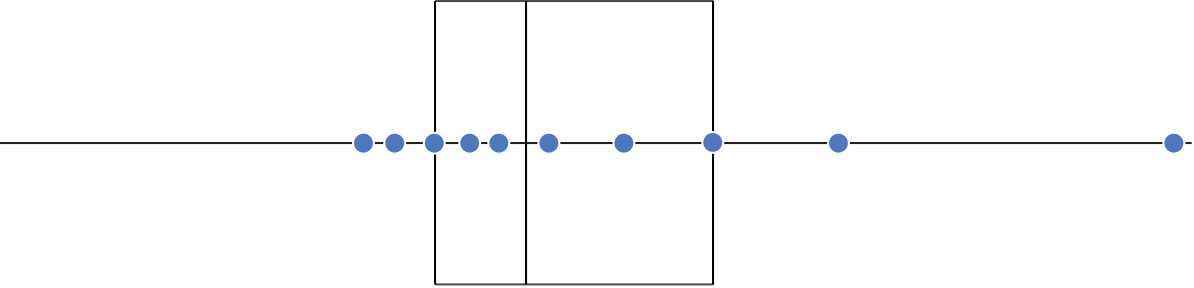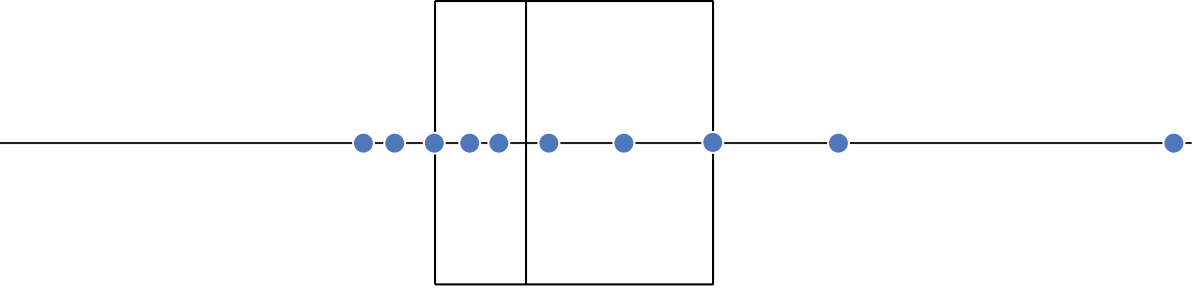
Gráfico de caixas (Box plot)
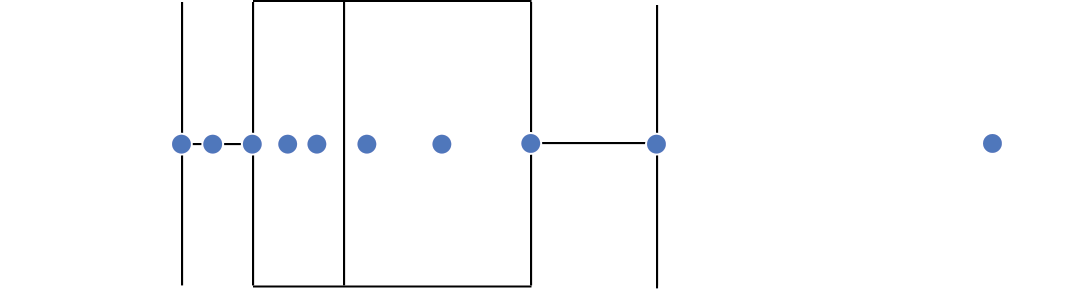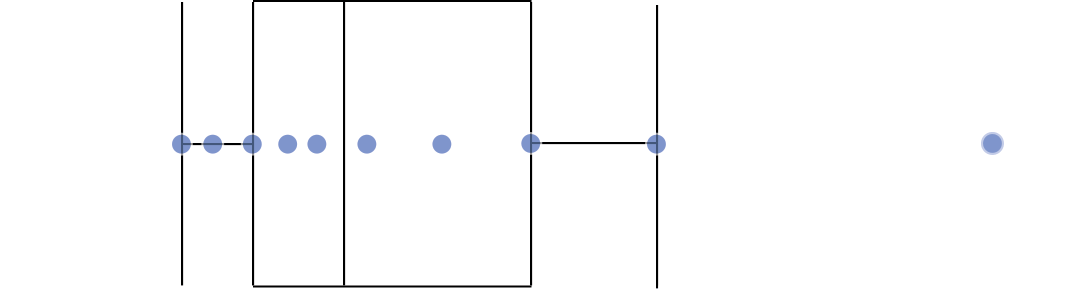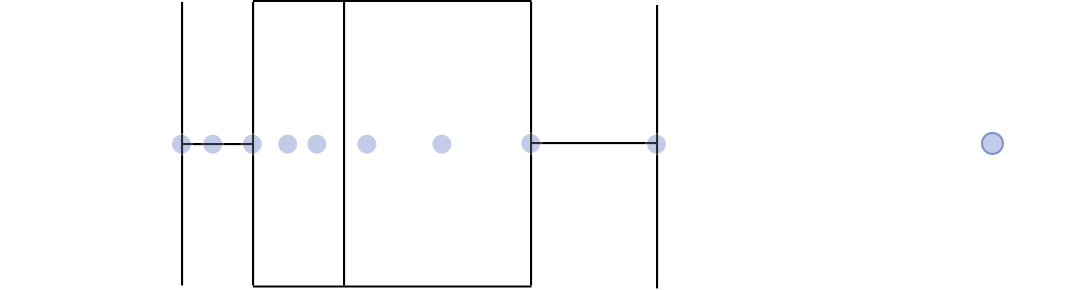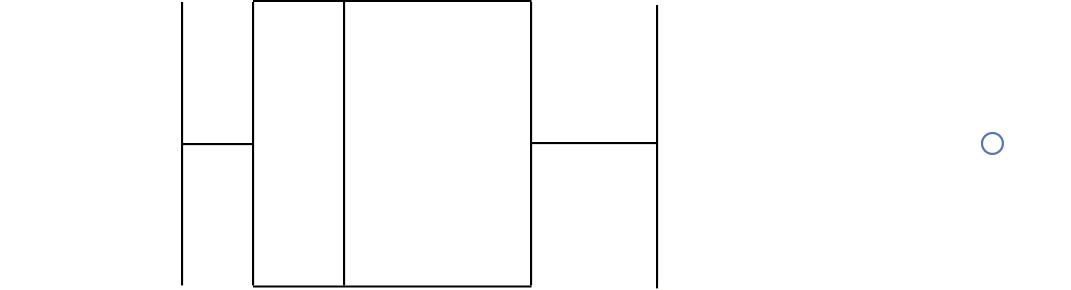
Gráfico de caixas (Box plot)
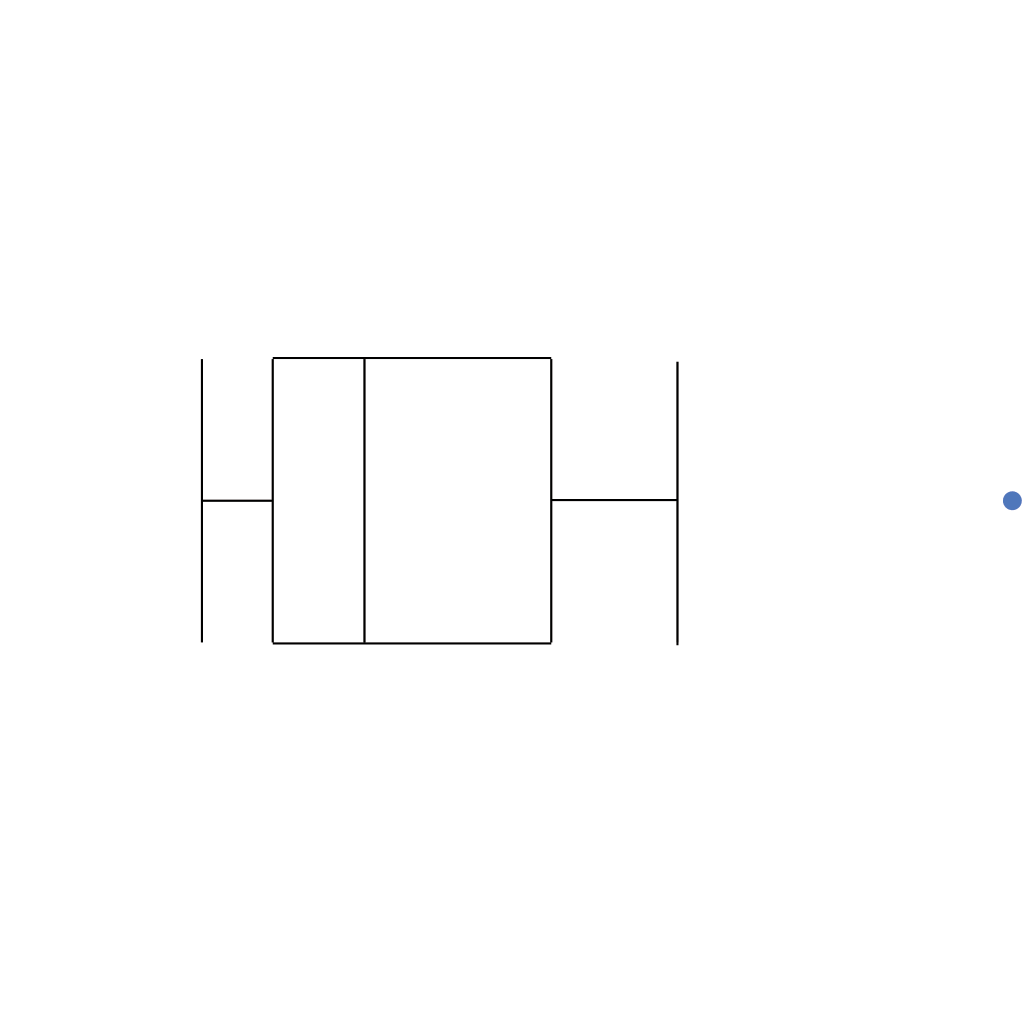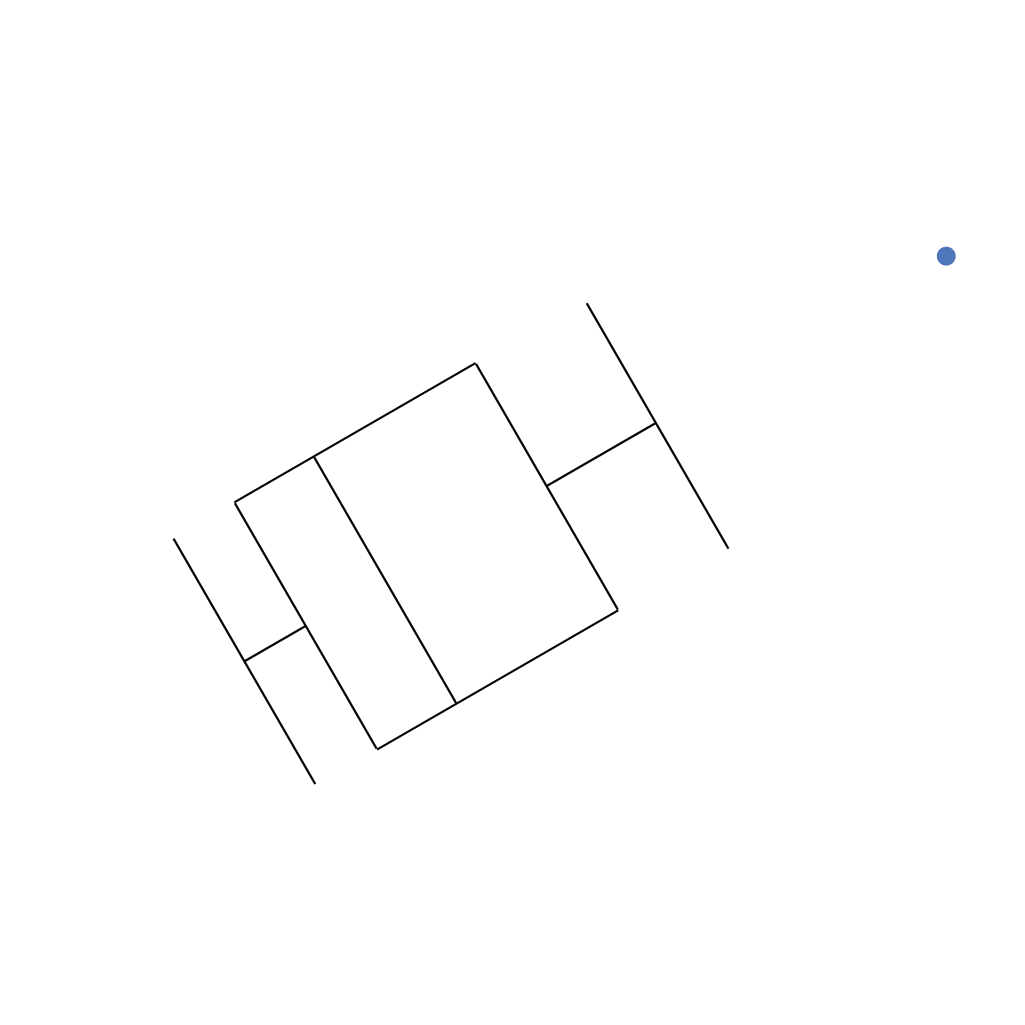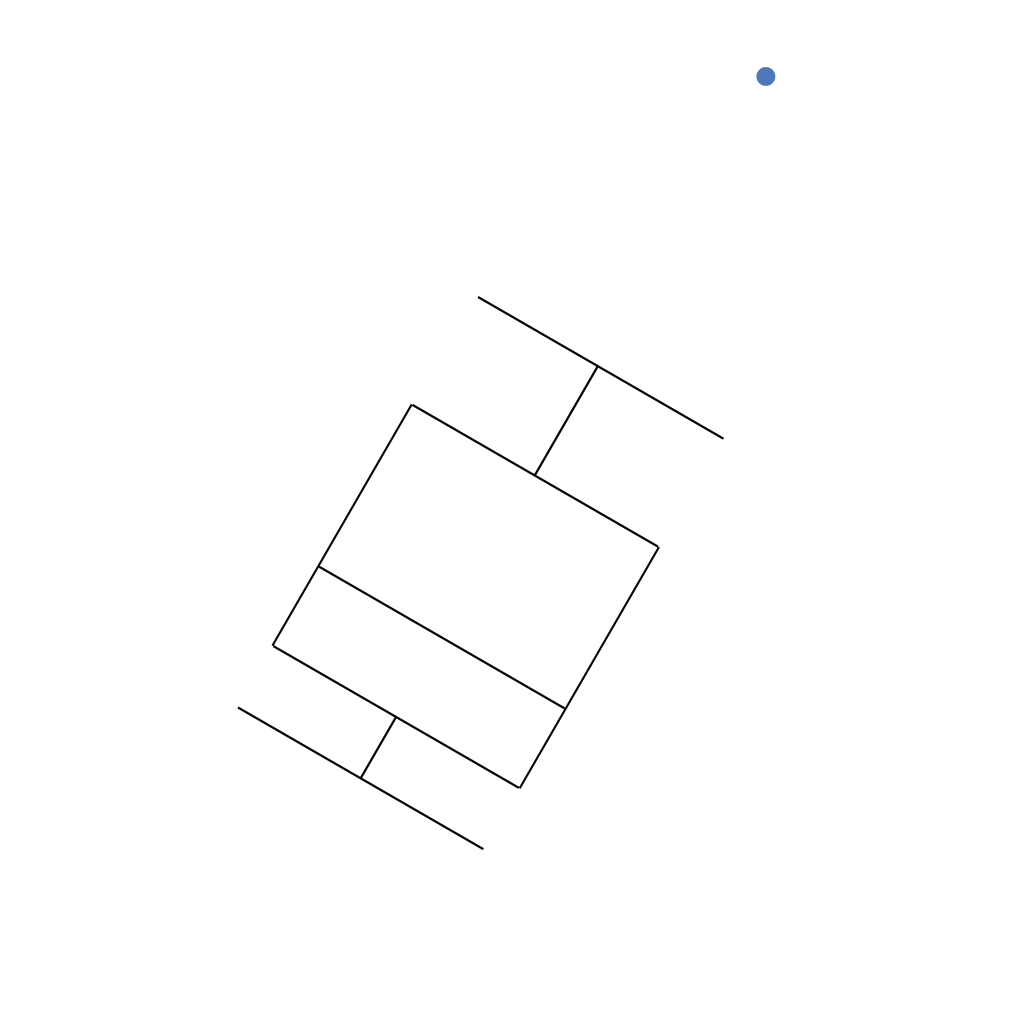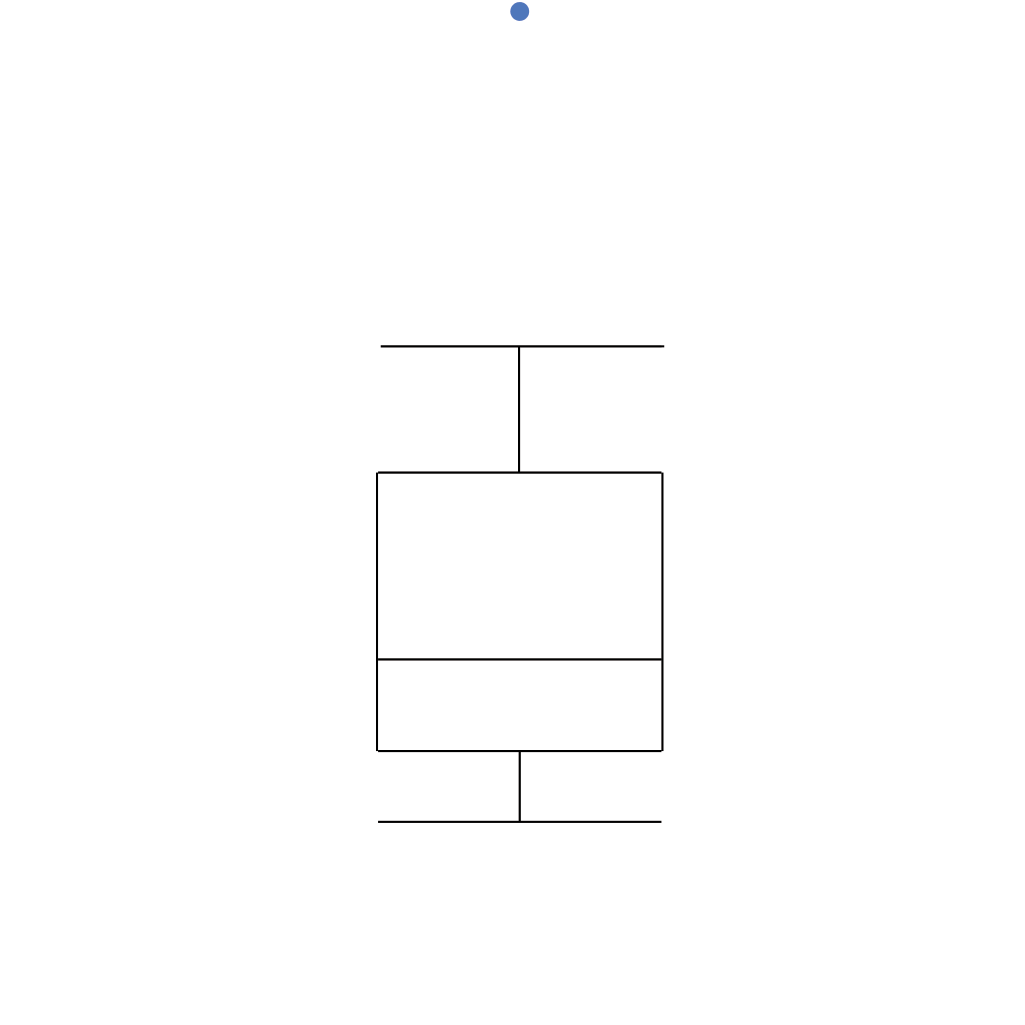
Gráfico de caixas (Box plot)
Dúvidas?
3. Análise de dados univariados
Pergunta e hipótese
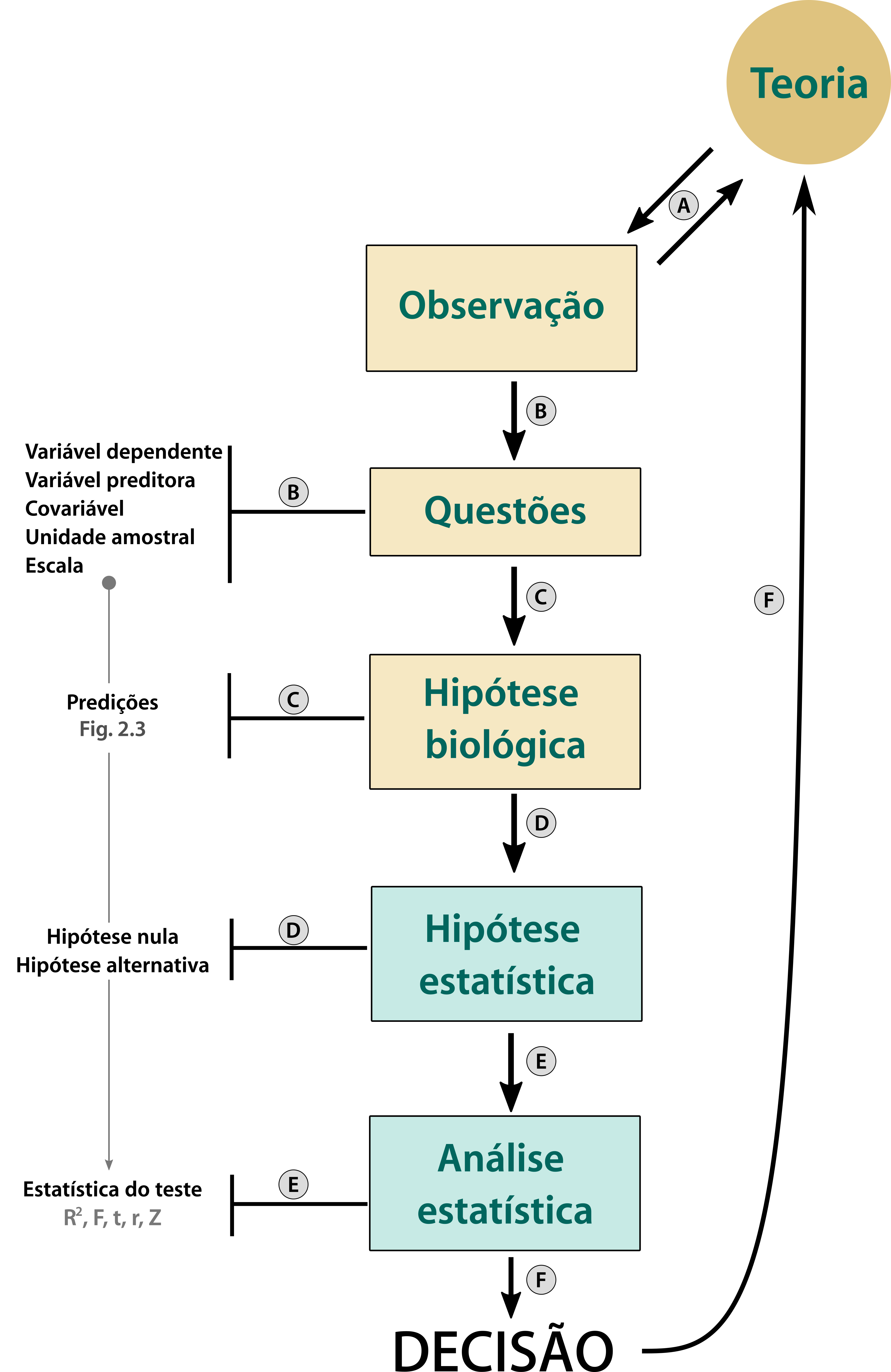
Método hipotético-dedutivo
A. Teoria ecológica/biológica
B. Desenho amostral (variáveis, unidade amostral, esforço, escala - temporal e espacial)
C. Hipótese ecológica/biológica e predições
D. Hipótese estatística - hipótese nula (H0) e hipótese alternativa (H1)
E. Análise estatística - inferência (estatística ou estimador): p, R², F, t, z, r…
F. Decisão: interpretação à luz da teoria ecológica/biológica
Hipótese ecológica e estatística
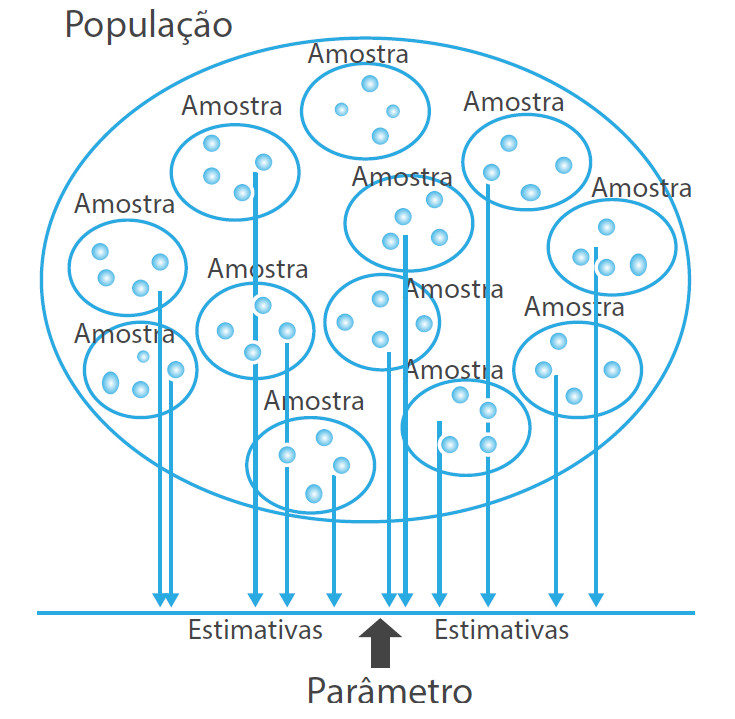
População e amostra
População: todos dados existentes
Amostra: parte dos dados coletados
Amostragem: processo de coleta de dados
Estatística descritiva: análise descritiva dos dados coletados (medidas-resumo, tabela e gráficos)
Inferência estatística: análise inferencial da amostra para afirmar algo sobre a população (testes estatísticos)
Parâmetros: medidas sobre a população (média, variância, desvio padrão) (letras gregas)
Estatísticas (estimadores): medidas sobre a amostra (média, variância, desvio padrão, erro padrão) (letras romanas)
Parâmetro e estatística (estimador)
Média
Média da população (parâmetro)
\[\mu=\sum \frac{x_{i}}{n}\]
Média da amostra (estatística)
\[\bar{x}=\sum \frac{x_{i}}{n}\]
Variância

Variância da população (parâmetro)
\[\sigma^2=\sum \frac{(x_{i}-\bar{x})^2}{n}\]
Variância da amostra (estatística)
\[s^2=\sum \frac{(x_{i}-\bar{x})^2}{n}\]
Desvio padrão

Desvio padrão da população (parâmetro)
\[\sigma =\sqrt {\sum \frac{(x_{i}-\bar{x})^2}{n}}\]
Desvio padrão da amostra (estatística)
\[s =\sqrt {\sum \frac{(x_{i}-\bar{x})^2}{n}}\]
Erro padrão
Erro padrão da média amostral
Variação da média amostral em relação à média populacional (confiabilidade da média amostral)
\[s_\bar{x} =\frac{s}{\sqrt{n}}\]
Inferência estatística
Conclusões sobre características da população (parâmetros) através de estatísticas (estimadores) calculadas a partir da(s) amostra(s)

Inferência estatística
Duas categorias:
- Estimação pontual com um intervo (intervalo de confiança)
- Testes de hipóteses (testar afirmações)
Intervalo de confiância (IC)
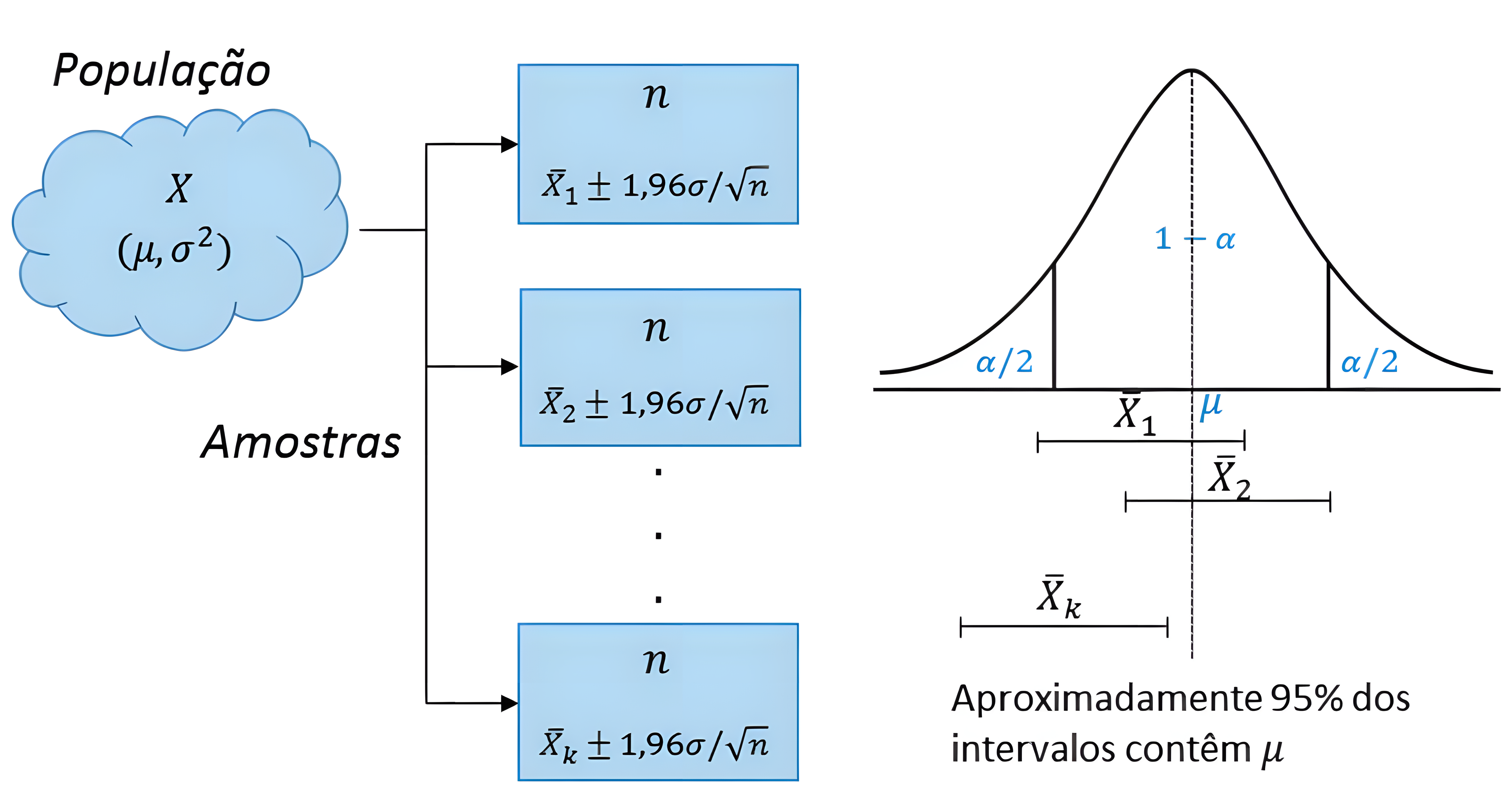
- Intervalo de valores com alta probabilidade (grau de confiança) de conter o valor do parâmetro da população
Intervalo de confiância = [Estivativa pontual ± Erro da estimativa]
Grau de confiança: probabilidade do intervalo de confiança conter o parâmetro populacional (1−α)
α: probabilidade de erro ao se afirmar que o intervalo contém o valor do parâmetro
Ex. A estimativa da média foi 2.5 ± 0.5 para um nível de significância de 5% (α = 0.05)
Intervalo de confiância (IC)
1. Variância σ² da população é conhecida
Erro segue a distribuição z normal padronizada (μ = 0 e σ² = 1)
\[IC^{\mu}_{100(1 - \alpha)\%} = (\bar{x} - z_{\alpha/2}\times\frac{\sigma}{\sqrt{n}}; \bar{x} + z_{\alpha/2}\times\frac{\sigma}{\sqrt{n}}); z_{\alpha/2} = 1.96\]
Ex.: x=10 (amostra), n=100 (amostra), σ=2 (população), α=5%
\[IC^{\mu}_{95} = (10 - 1.96\times\frac{2}{\sqrt{100}}; 10 + 1.96\times\frac{2}{\sqrt{100}})\]
\[IC^{\mu}_{95} = [9.608; 10.392]\]
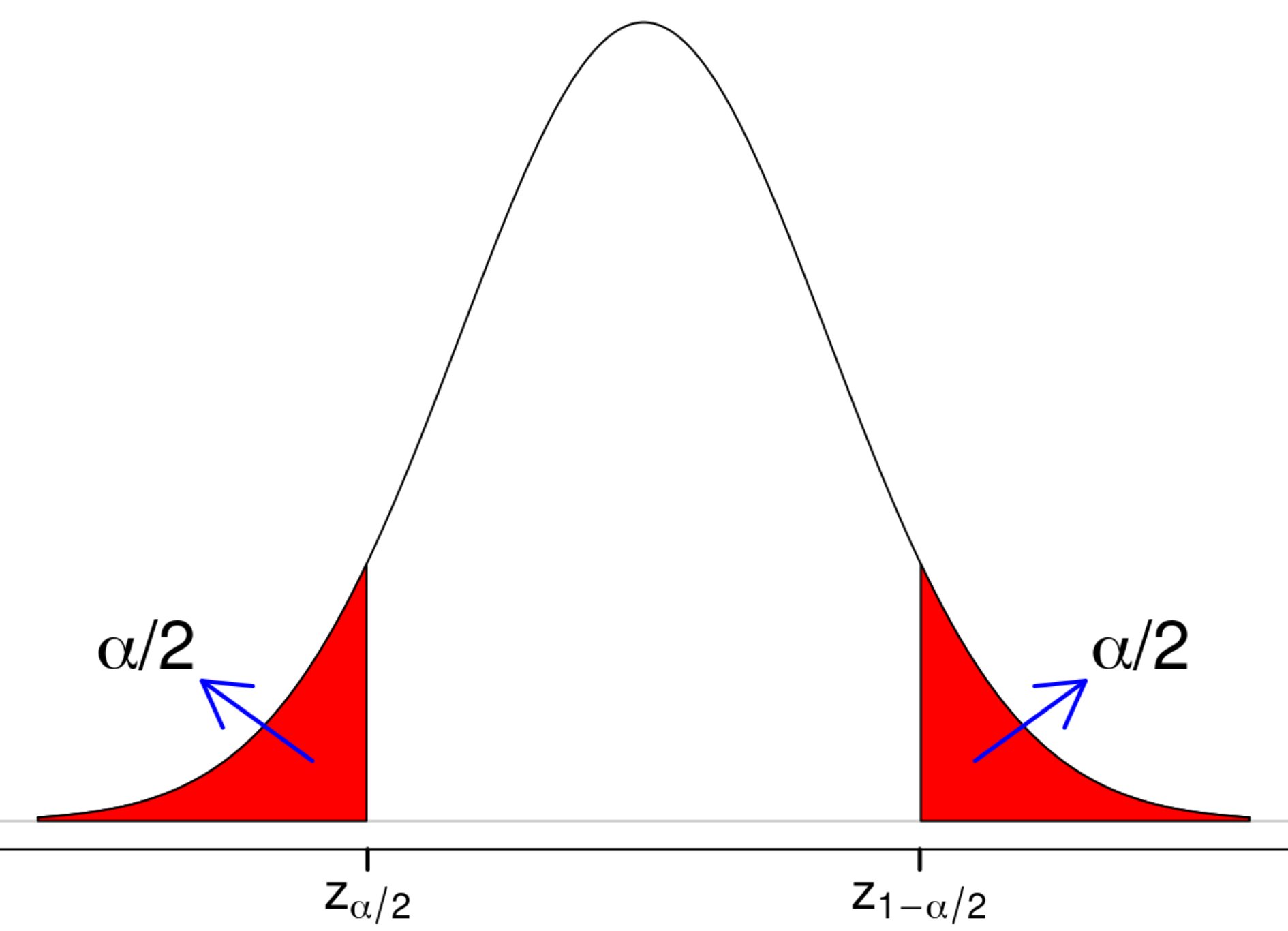
Distribuição z
Distribuição de probabilidades de z, com média de z (μ = 0) e desvio padrão de z (σ² = 1)
Como estimamos valores acima e abaixo da média populacional, e para um nível de significância de 5% (α = 0.05), dividimos (α/2 = 0.025) e (1 - α/2 = 0.975)
Intervalo de confiância (IC)
1. Variância σ² da população é conhecida
Intervalo de confiância (IC)
2. Variância σ² da população é desconhecida
Erro segue a distribuição t
- Variância amostral (s²) ou desvio padrão amostral (s)
- Graus de liberdade (n - 1), sendo n o número de observações
\[IC^{\mu}_{100(1 - \alpha)\%} = (\bar{x} - t_{(n - 1, \alpha/2)}\times\frac{s}{\sqrt{n}}; \bar{x} + t_{(n - 1,\alpha/2)}\times\frac{s}{\sqrt{n}})\]
\[s = \sqrt\frac{\sum_{i = 1}^n{(x_i - \bar{x})^2}}{n - 1}\]
Distribuição t
Graus de liberdade (g.l.): quantidade de observações (n) menos a quantidade de parâmetros estimados (μ)
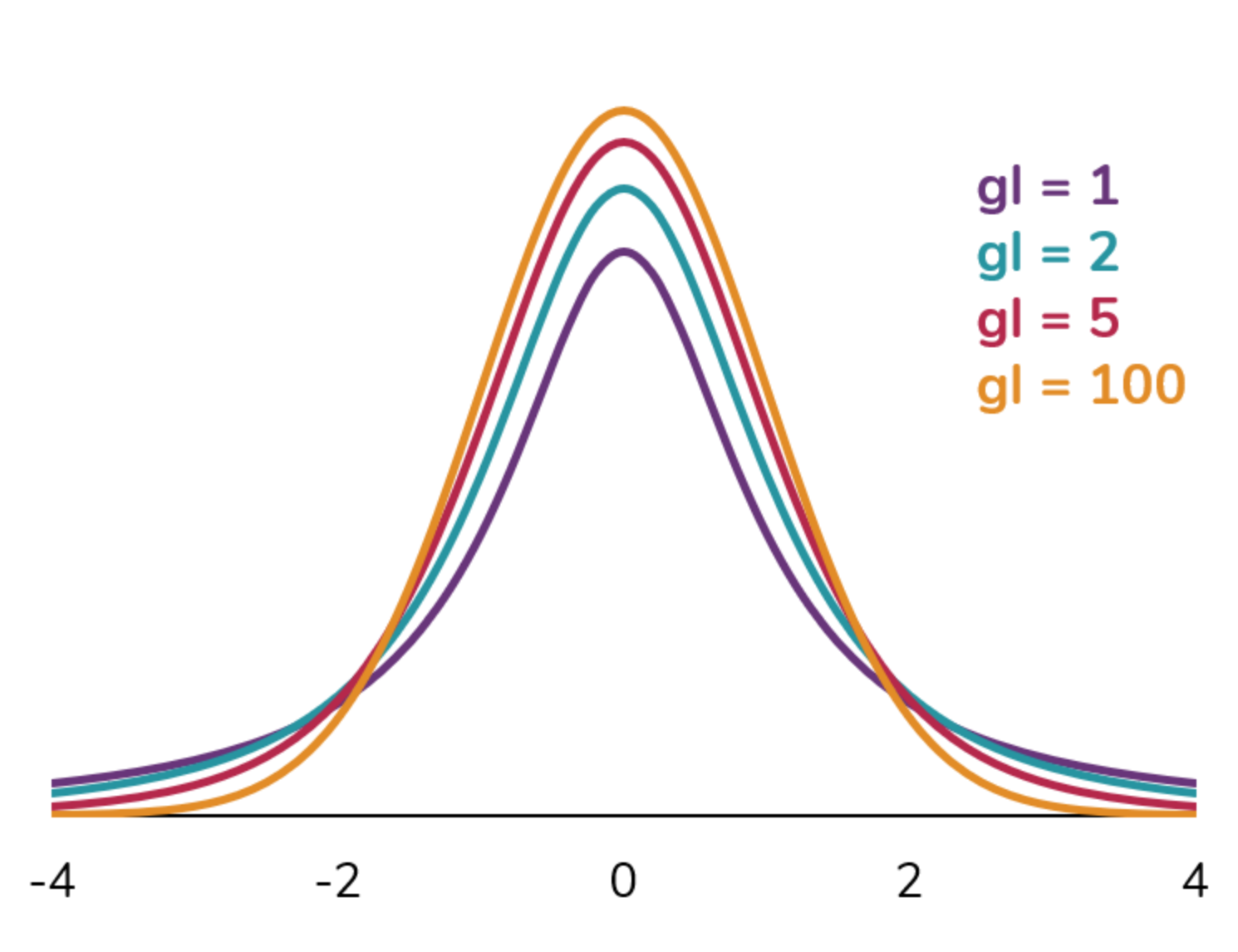
Intervalo de confiância (IC)
2. Variância σ² da população é desconhecida
Ex.: [36.4, 35.7, 37.2, 36.5, 34.9, 35.2, 36.3, 35.8, 36.6, 36.9]
\[\bar{x} = \frac{\sum_{i=1}^n x_i}{n} = 36.15\] \[s = \sqrt\frac{\sum_{i = 1}^n{(x_i - \bar{x})^2}}{n - 1} = \sqrt\frac{4.865}{9} = 0.7352\] \[t_{(n−1,α/2)} = t_{(9,0.025)} = 2.26\]
Intervalo de confiância (IC)
2. Variância σ² da população é desconhecida
Ex.: [36.4, 35.7, 37.2, 36.5, 34.9, 35.2, 36.3, 35.8, 36.6, 36.9]
\[IC^{\mu}_{95} = (36.15 - 2.26\times\frac{0.7352}{\sqrt{10}}; 36.15 + 2.26\times\frac{0.7352}{\sqrt{10}})\]
\[IC^{\mu}_{95} = [35.624; 36.676]\]
Intervalo de confiância (IC)
2. Variância σ² da população é desconhecida
[1] 10[1] 36.15[1] 0.7352248[1] 2.262157[1] 35.62405[1] 36.67595Dúvidas?
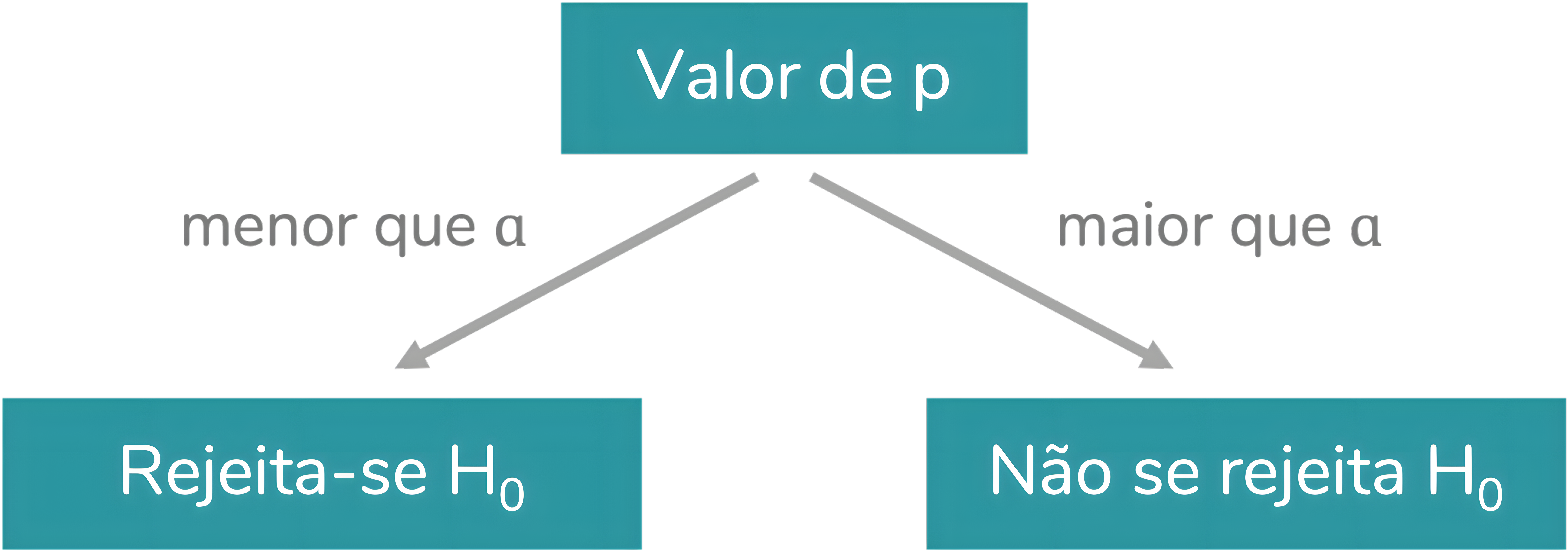
Teste de hipóteses
Regra de decisão para rejeitar (ou não) uma afirmação (hipótese) feita sobre um parâmetro populacional desconhecido, com base numa amostra aleatória
Ex:
- Testar se agroflorestas retêm mais caborno que a agricultura convencional
- Verificar se paisagens mais fragmentadas possuem menos espécies
- Testar se as pessoas gostam mais de Harry Potter ou Senhor dos Anéis
Teste de hipóteses
Regra de decisão para rejeitar (ou não) uma afirmação (hipótese) feita sobre um parâmetro populacional desconhecido, com base numa amostra aleatória
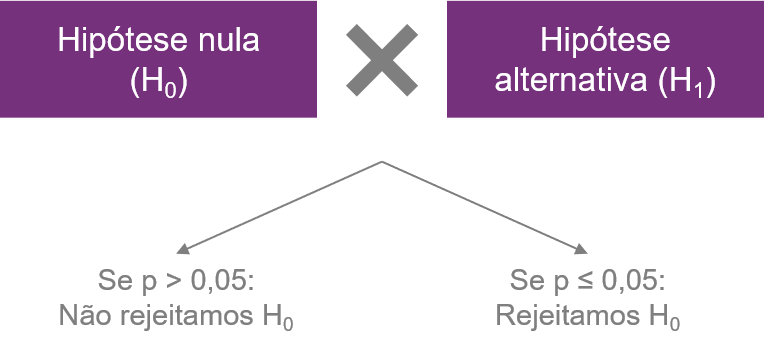
Hipótese Nula (H0): hipótese a ser testada
Hipótese Alternativa (H1): hipótese a ser confrontada com H0
- Teste feito para concluir na rejeição (ou não) de H0
- Decisão é feita com base em informações de uma amostra, estaremos sujeitos a dois tipos de erros
Teste de hipóteses
Erros
- Erro do tipo I: rejeitar H0 quando H0 é verdadeira
α = P(erro do tipo I) = P(rejeitar H0|H0 é verdadeira)
- Erro do tipo II: não rejeitarmos H0 quando H0 é falsa
β = P(erro do tipo II) = P(não rejeitar H0|H0 é falsa)
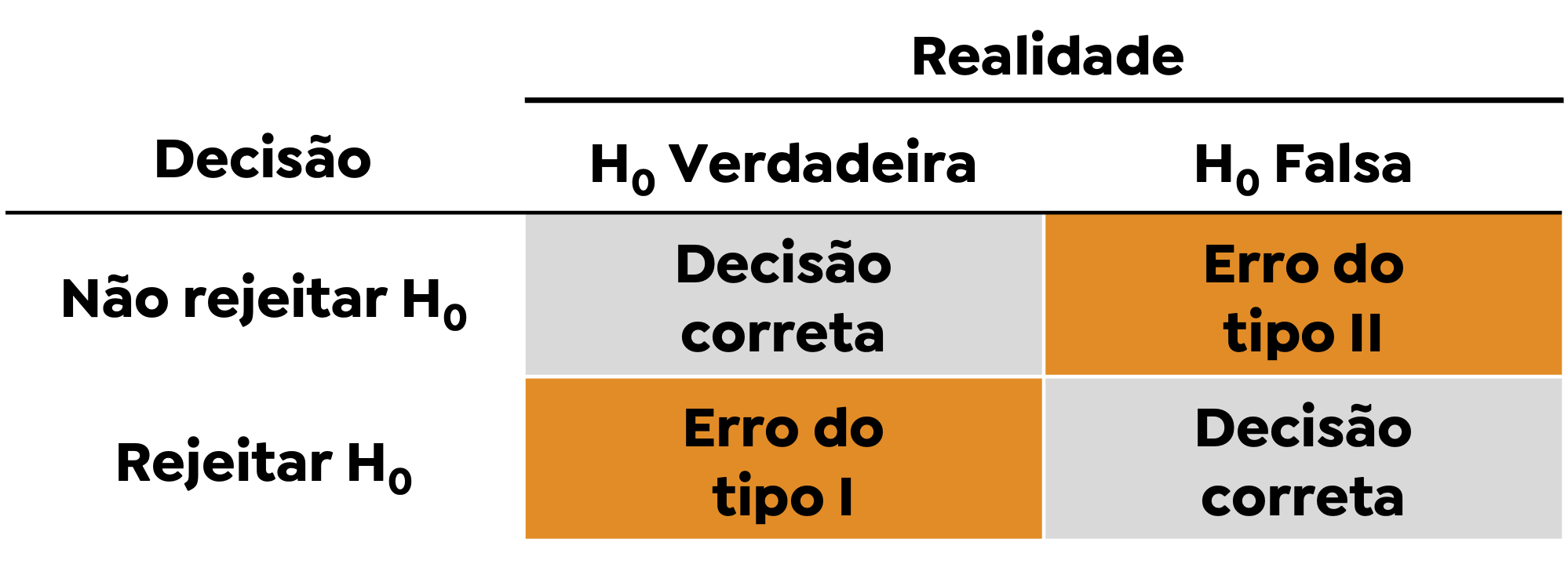
Teste de hipóteses
Etapas
- Definir as hipóteses nula (H0) e alternativa (H1)
- Fixar o nível de significância (α) (e.g. α = 5% ou 0.05)
- Determinar a estatística do teste (e.g. t)
- Determinar a região crítica do teste (RC)
- Calcular o valor da estatística do teste (amostra)
- Se o valor calculado no passo 5 pertence à RC, rejeita-se H0, se não pertencer, não se rejeita H0
- Conclusão do teste
Teste de hipóteses
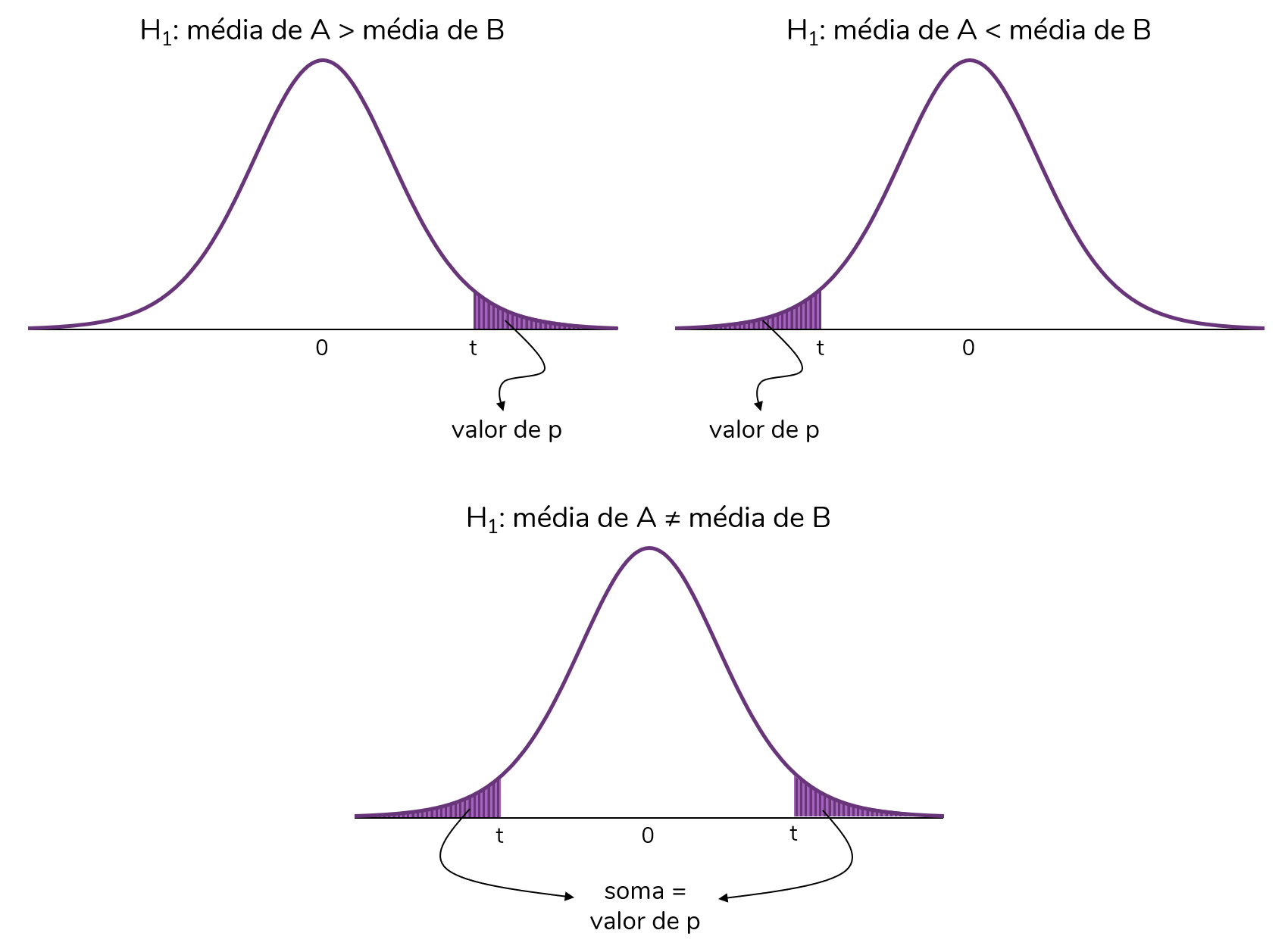
Detalhamento
1 Definição das hipóteses
H0: µ = µ0
H1: µ ≠ µ0
H0: µ = µ0
H1: µ < µ0
H0: µ = µ0
H1: µ > µ0
Fixar o nível de significância α (5% ou 0.05)
Definir a estatística de teste (t)
\[t = \frac{\bar{x} - \mu}{s / \sqrt{n}} \sim t_{n - 1}\]
Teste de hipóteses
Detalhamento
4 Definir a região crítica (RC)
H0: µ = µ0
H1: µ ≠ µ0
H0: µ = µ0
H1: µ < µ0
H0: µ = µ0
H1: µ > µ0
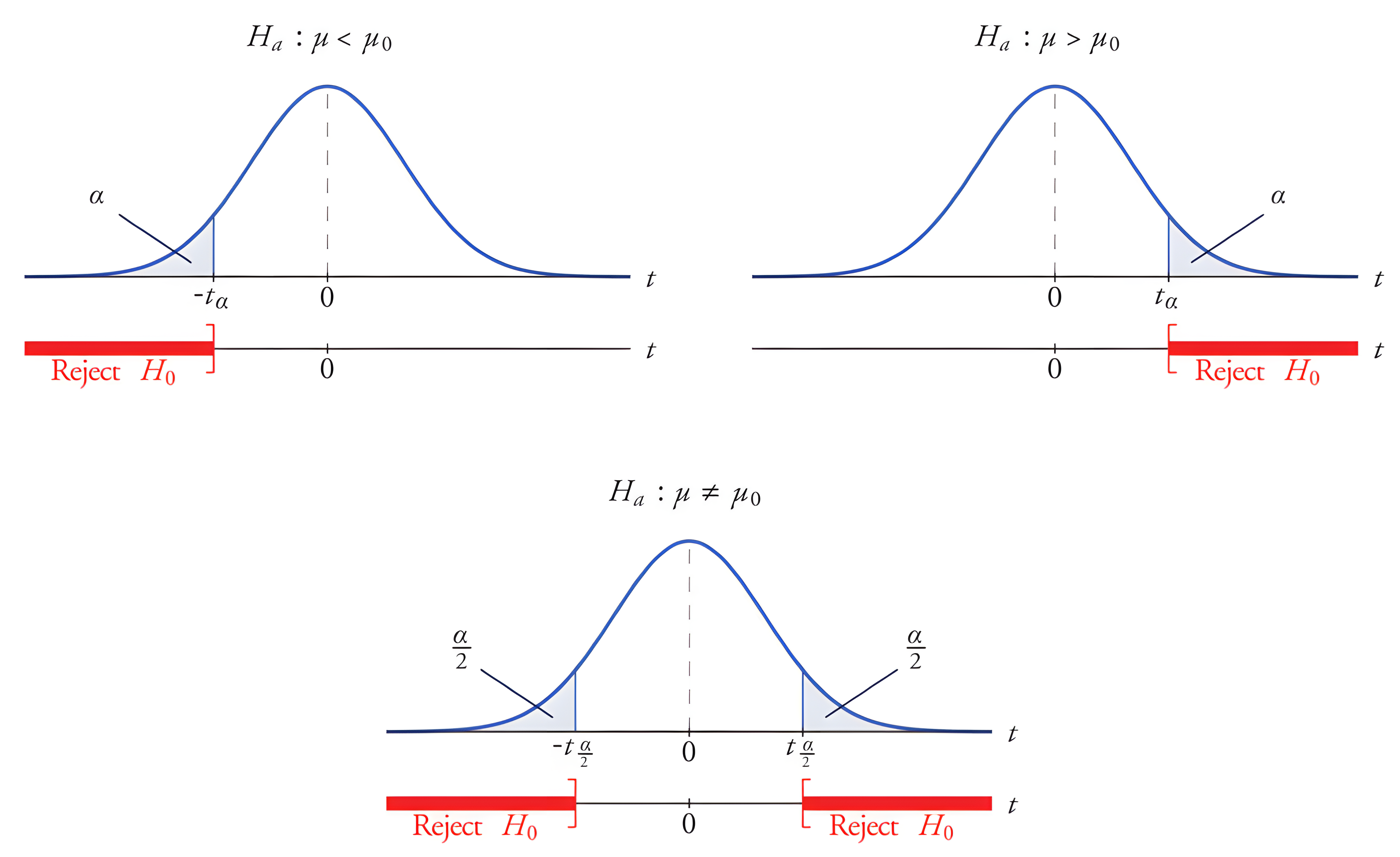
Teste de hipóteses
Detalhamento
- Com base nos valores observados da amostra, calcular o valor da estatística do teste t
\[t_c = \frac{\bar{x} - \mu_0}{s/\sqrt{n}}\]
Se tc ∈ RC ⇒ rejeitar H0 (corrobora H1)
Se tc ∉ RC ⇒ não rejeitar H0 (não corrobora H1)Concluir sobre a decisão tomada no passo 6
Teste de hipóteses
Exemplo
Medimos a altura de 25 bananeiras adultas num SAF. Sabemos que num cultivo convencional, as bananeiras adultas chegam a 3 metros. Considerando um nível de significância de 5%, podemos dizer que, em média, as bananeiras crescem mais no SAF?
altura = [4.3, 2.6, 4.4, 3.0, 2.7, 2.9, 3.3, 2.7, 4.3, 3.8, 3.1, 2.5, 4.3, 4.2, 4.0, 4.2, 3.3, 3.2, 3.4, 4.5, 3.5, 2.6, 2.5, 2.5, 4.2]
Teste de hipóteses
Exemplo
- As hipóteses que queremos testar são:
H0: µ = 3 contra H1: µ > 3
Fixamos α = 0.05
A estatística de teste é a t
\[t = \frac{\bar{x} - \mu}{s / \sqrt{n}} \sim t_{n - 1}\]
Teste de hipóteses
Exemplo
- A região crítica é do tipo (teste unicaldal à direita)
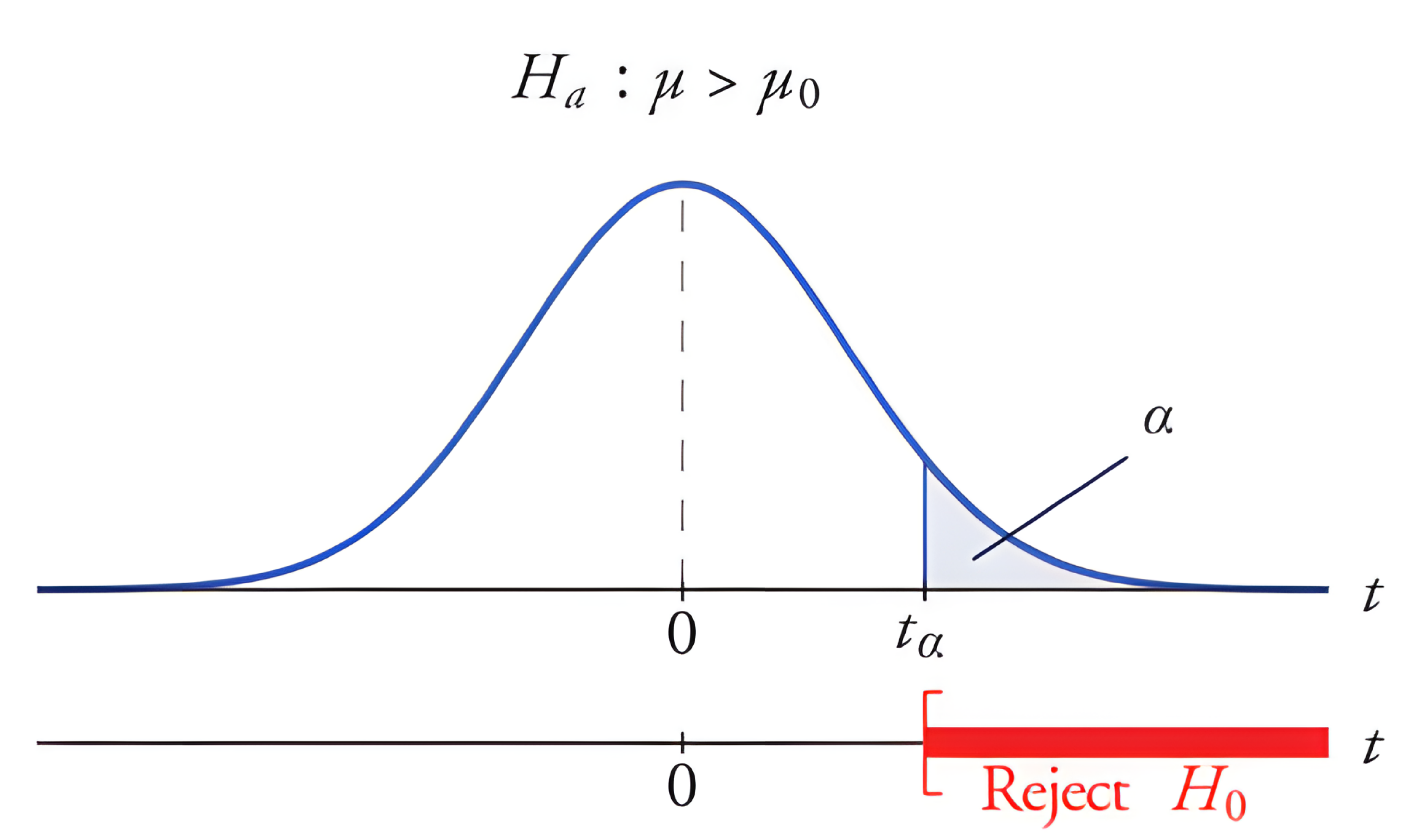
onde t = t(n-1, α) = t(24,0.05) = 1.71 (tabela da distribuição t)
Teste de hipóteses
Exemplo
- A partir dos dados amostrais temos que
\[t_c = \frac{\bar{x} - \mu_0}{s/\sqrt{n}} = \frac{3.44 - 3}{0.72/\sqrt{25}}=3.0731\]
Temos que tc ∈ RC pois 3.0731 > 1.71, portanto, rejeitamos a hipótese nula
Com base nos dados amostrais, podemos concluir, ao nível de 5% de significância, que a altura média das bananeiras é maior no SAF do que na agricultura convencional
Teste de hipóteses
Exemplo
Teste de hipóteses
Exemplo
# amostra
amostra <- c(4.3, 2.6, 4.4, 3.0, 2.7, 2.9, 3.3, 2.7, 4.3, 3.8, 3.1, 2.5, 4.3, 4.2, 4.0, 4.2, 3.3, 3.2, 3.4, 4.5, 3.5, 2.6, 2.5, 2.5, 4.2)
# teste t
t.test(x = amostra, mu = 3, alternative = "greater")
One Sample t-test
data: amostra
t = 3.0731, df = 24, p-value = 0.002607
alternative hypothesis: true mean is greater than 3
95 percent confidence interval:
3.195039 Inf
sample estimates:
mean of x
3.44 Valor de p
Probabilidade de termos obtido aqueles resultados (ou mais extremos), dado que a H0 é verdadeira
P(resultados | H0 é verdadeira)
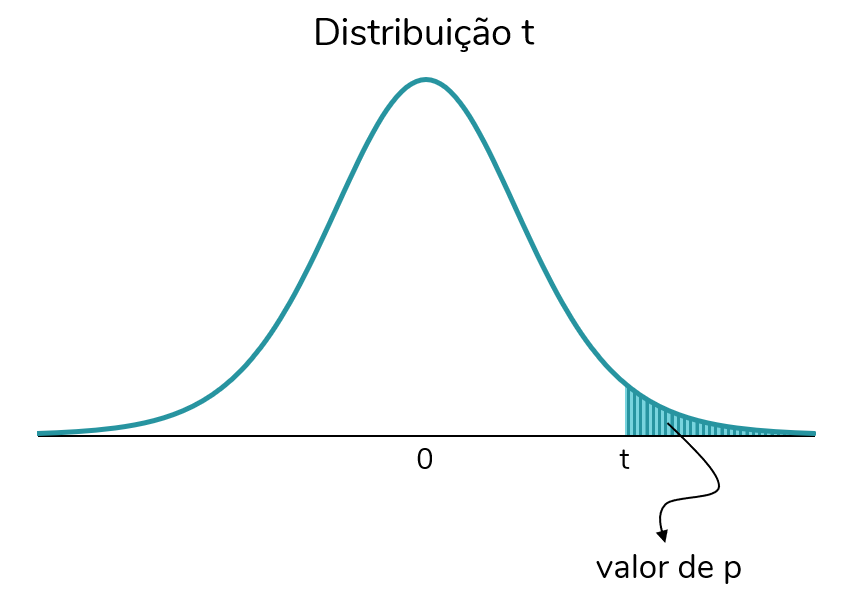
Valor de p
Probabilidade de termos obtido aqueles resultados (ou mais extremos), dado que a H0 é verdadeira
P(resultados | H0 é verdadeira)

Teste t
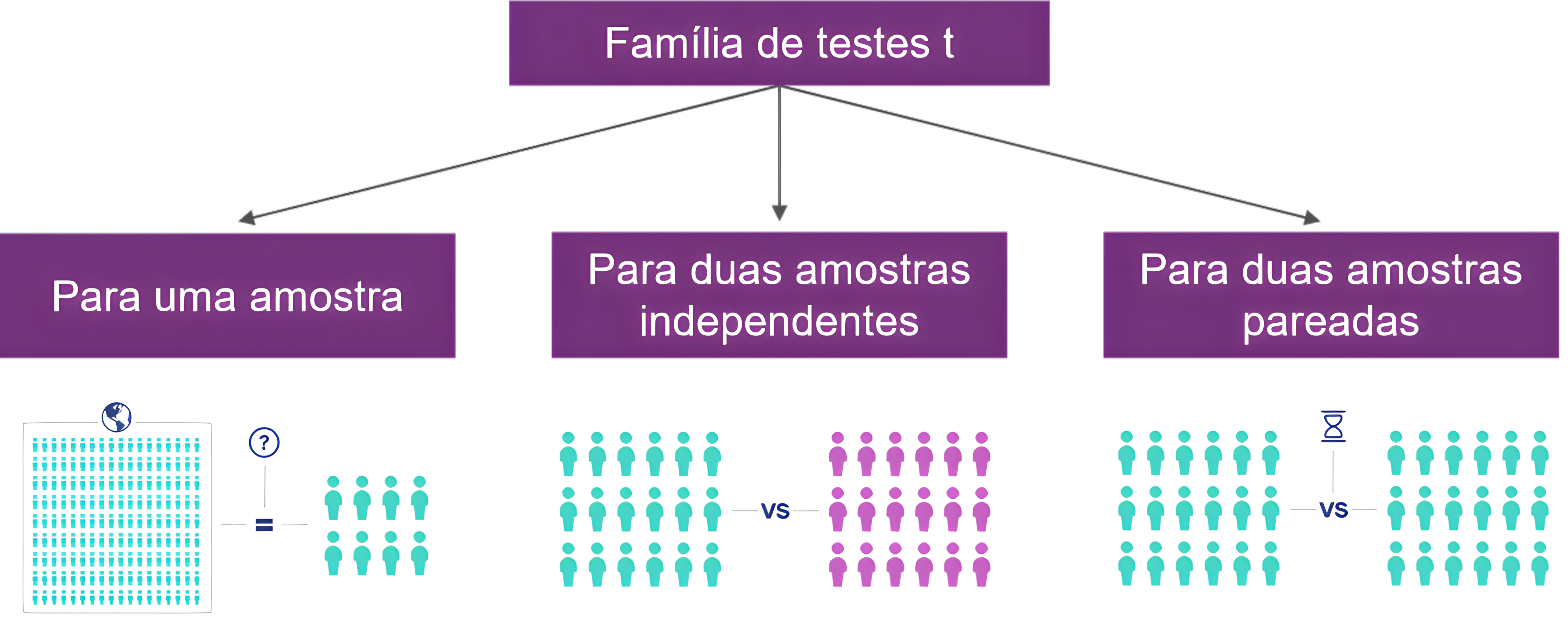
Teste t para uma amostra
One sample t test
Teste t para uma amostra
One sample t test
## maior
# amostra
amostra <- c(4.3, 2.6, 4.4, 3.0, 2.7, 2.9, 3.3, 2.7, 4.3, 3.8, 3.1, 2.5, 4.3, 4.2, 4.0, 4.2, 3.3, 3.2, 3.4, 4.5, 3.5, 2.6, 2.5, 2.5, 4.2)
# teste t
t.test(x = amostra, mu = 3, alternative = "greater")
One Sample t-test
data: amostra
t = 3.0731, df = 24, p-value = 0.002607
alternative hypothesis: true mean is greater than 3
95 percent confidence interval:
3.195039 Inf
sample estimates:
mean of x
3.44 Teste t para uma amostra
One sample t test
## menor
# amostra
amostra <- c(4.3, 2.6, 4.4, 3.0, 2.7, 2.9, 3.3, 2.7, 4.3, 3.8, 3.1, 2.5, 4.3, 4.2, 4.0, 4.2, 3.3, 3.2, 3.4, 4.5, 3.5, 2.6, 2.5, 2.5, 4.2)
# teste t
t.test(x = amostra, mu = 3, alternative = "less")
One Sample t-test
data: amostra
t = 3.0731, df = 24, p-value = 0.9974
alternative hypothesis: true mean is less than 3
95 percent confidence interval:
-Inf 3.684961
sample estimates:
mean of x
3.44 Teste t para uma amostra
One sample t test
## diferente
# amostra
amostra <- c(4.3, 2.6, 4.4, 3.0, 2.7, 2.9, 3.3, 2.7, 4.3, 3.8, 3.1, 2.5, 4.3, 4.2, 4.0, 4.2, 3.3, 3.2, 3.4, 4.5, 3.5, 2.6, 2.5, 2.5, 4.2)
# teste t
t.test(x = amostra, mu = 3, alternative = "two.sided")
One Sample t-test
data: amostra
t = 3.0731, df = 24, p-value = 0.005213
alternative hypothesis: true mean is not equal to 3
95 percent confidence interval:
3.144495 3.735505
sample estimates:
mean of x
3.44 Teste t

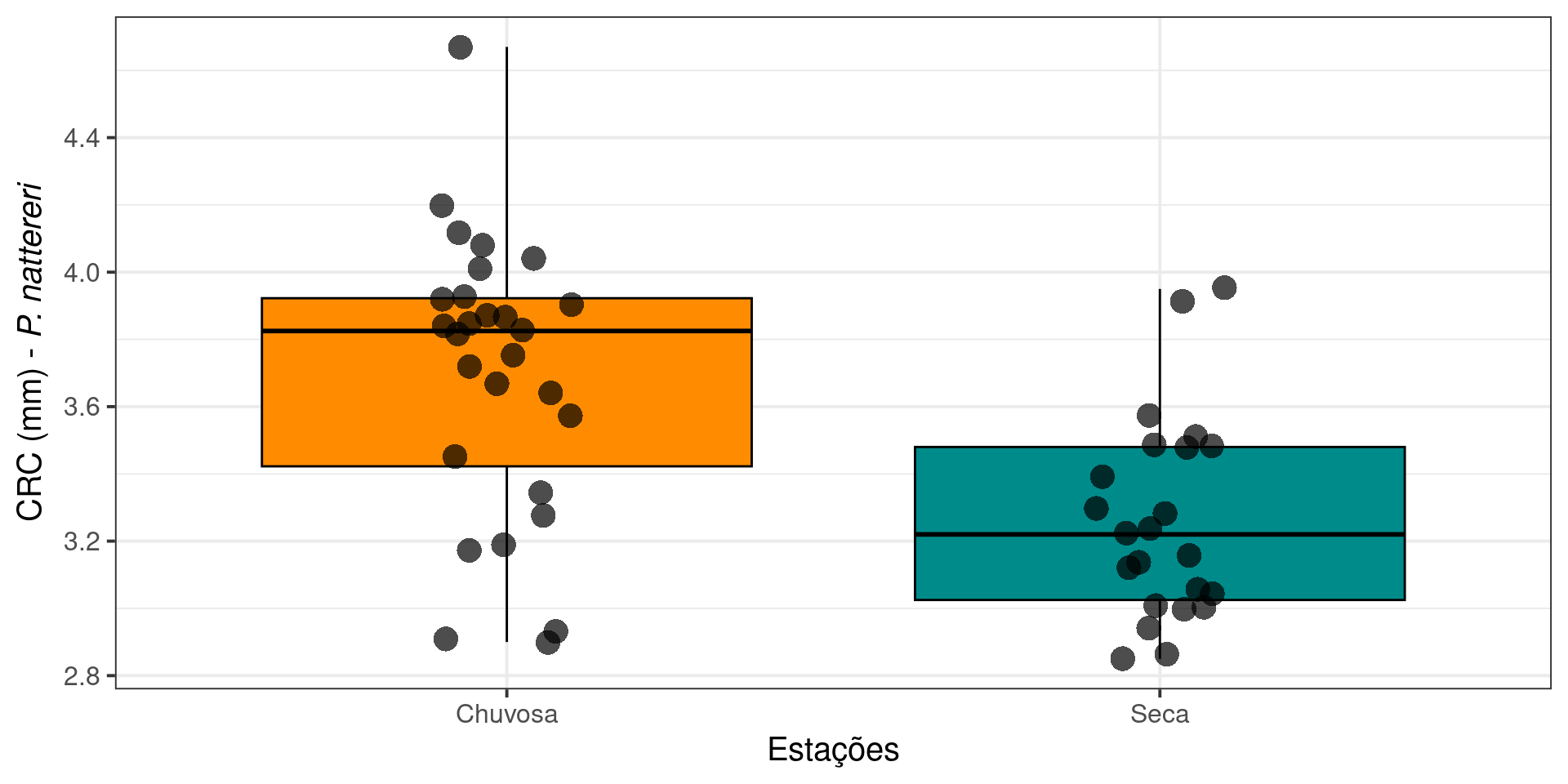
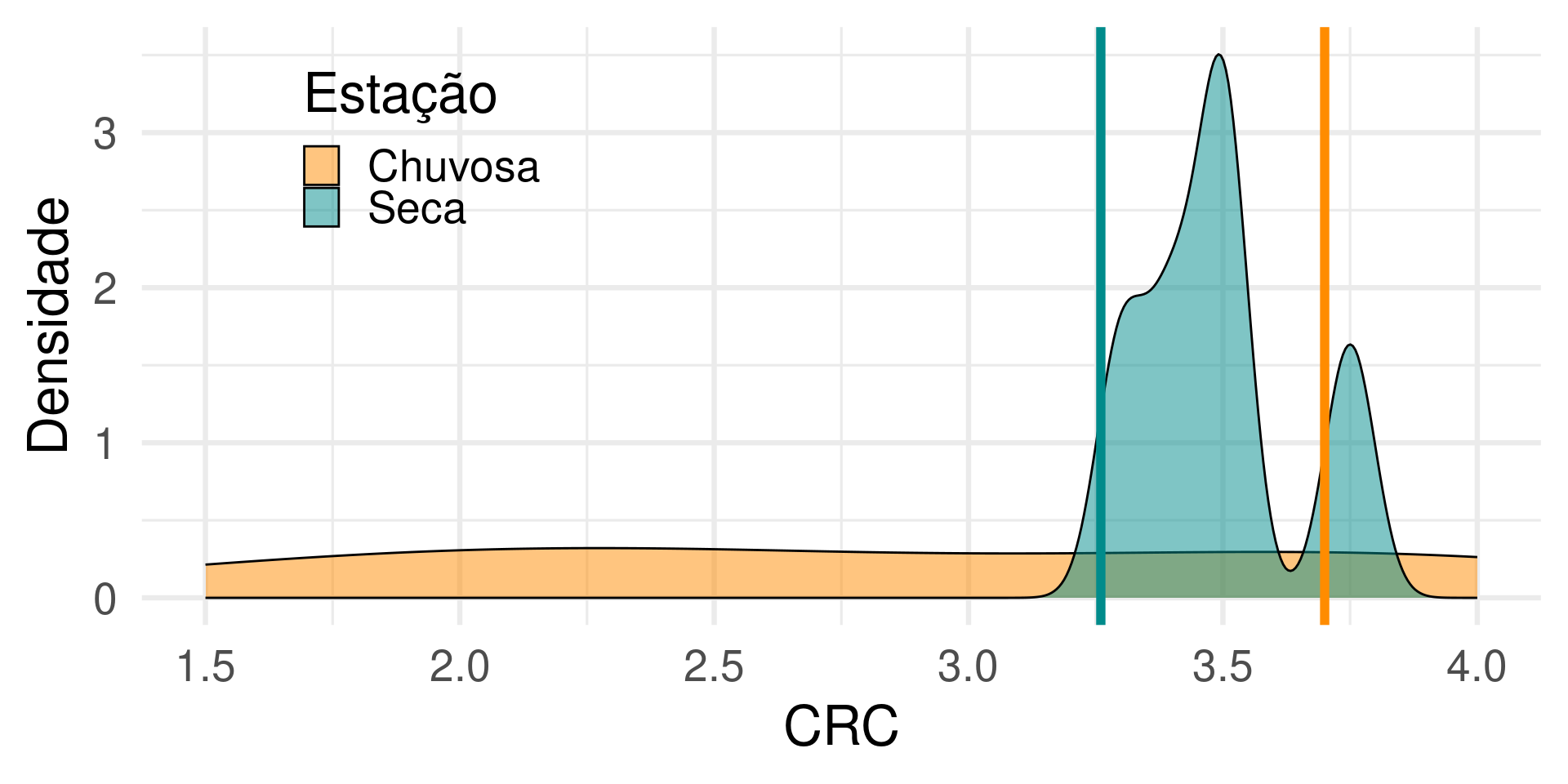
Physalaemus nattereri
Comprimento rostro-cloacal (CRC)

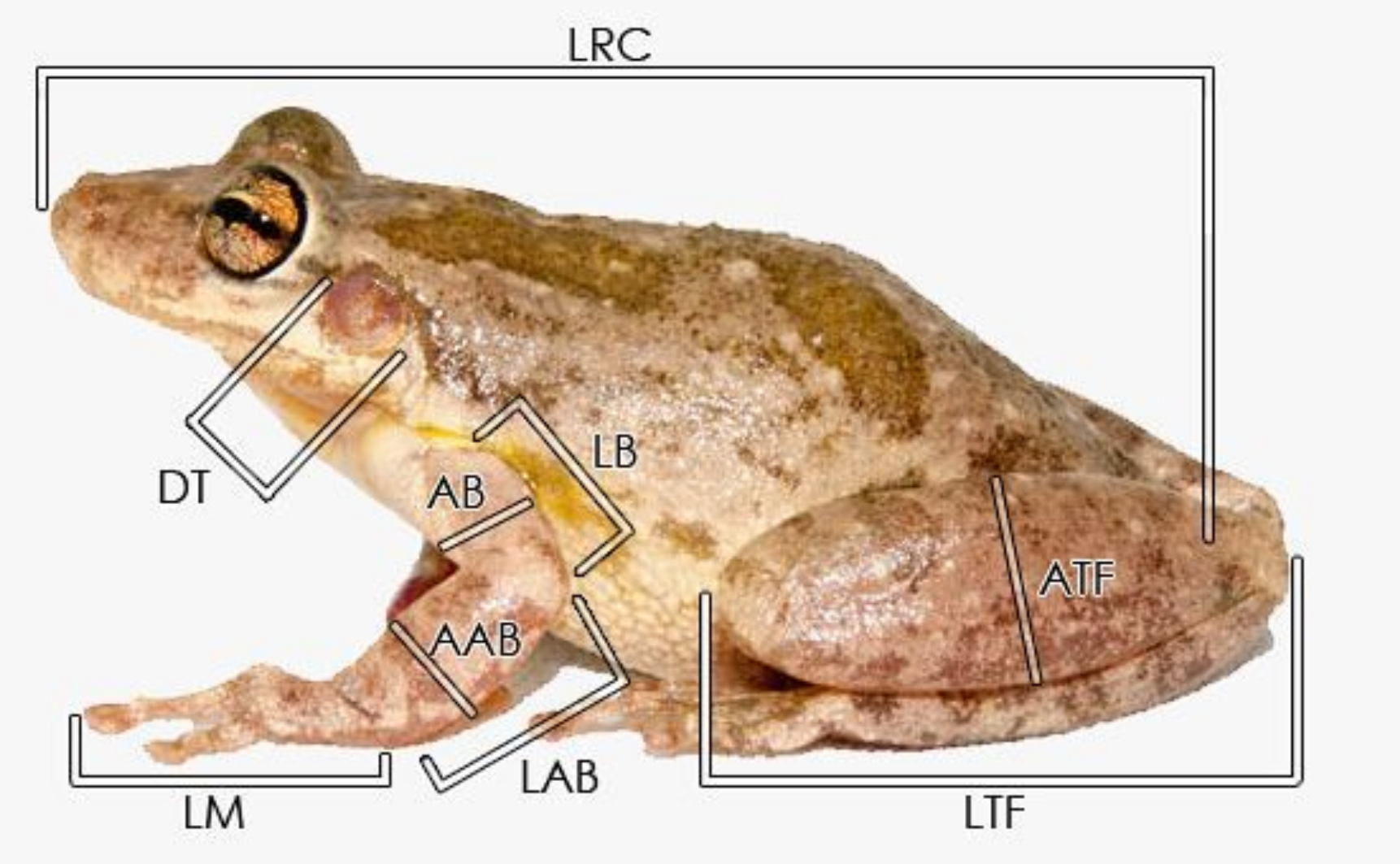
Teste t para duas amostras indep.
Independent-samples t test
| Estacao | mean | sd |
|---|---|---|
| Chuvosa | 3.70 | 0.42 |
| Seca | 3.26 | 0.30 |
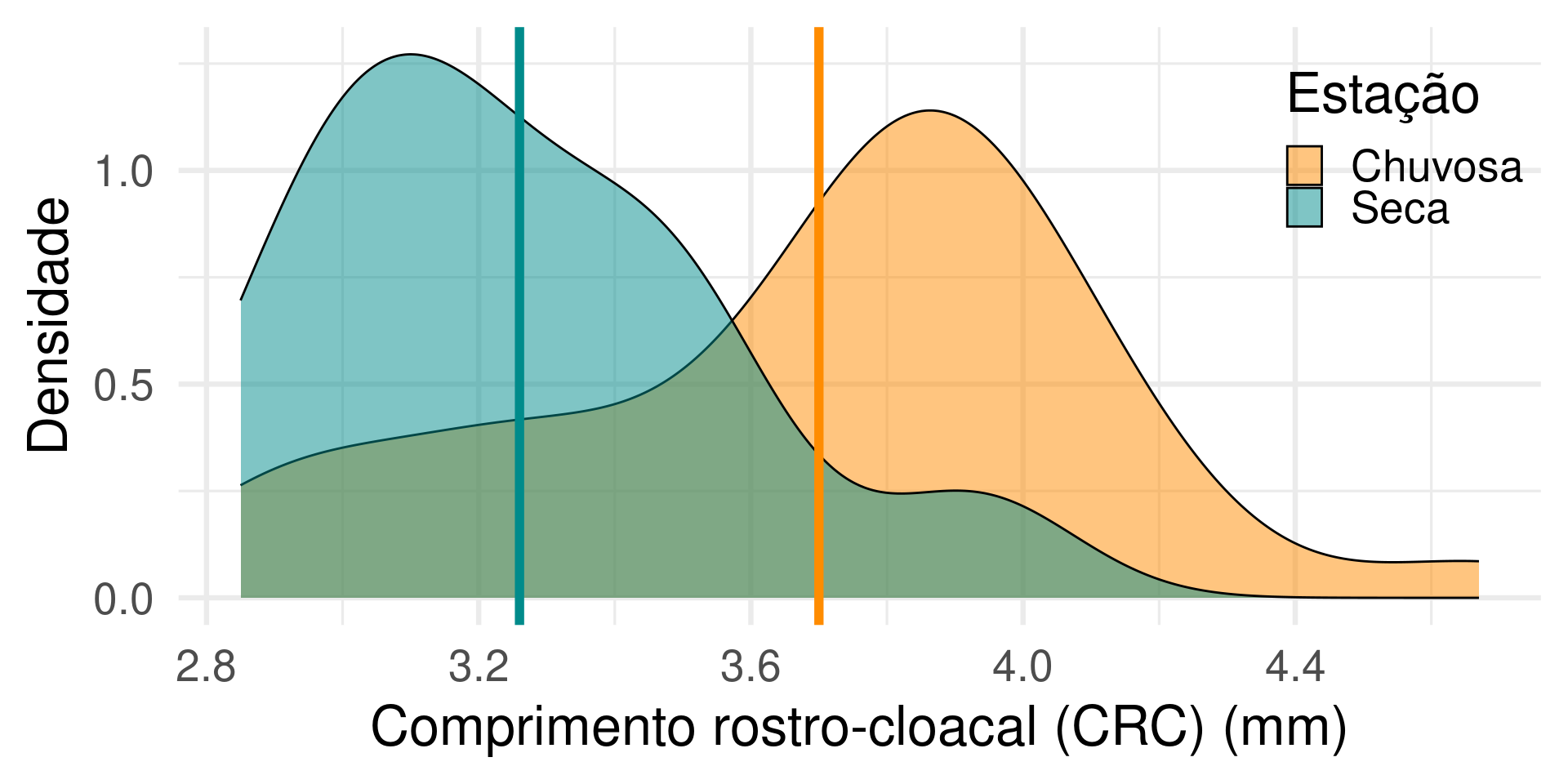
Teste t para duas amostras indep.
Independent-samples t test
## Teste de normalidade dos residuos - H0: distribuicao dos residuos é normal
residuos <- residuals(lm(CRC ~ Estacao, data = teste_t_var_igual))
shapiro.test(residuos)
Shapiro-Wilk normality test
data: residuos
W = 0.98307, p-value = 0.6746
Teste t para duas amostras indep.
Independent-samples t test
## Teste de homogeneidade de variancia - H0: variancia e homogenea
car::leveneTest(CRC ~ as.factor(Estacao), data = teste_t_var_igual)Levene's Test for Homogeneity of Variance (center = median)
Df F value Pr(>F)
group 1 1.1677 0.2852
49 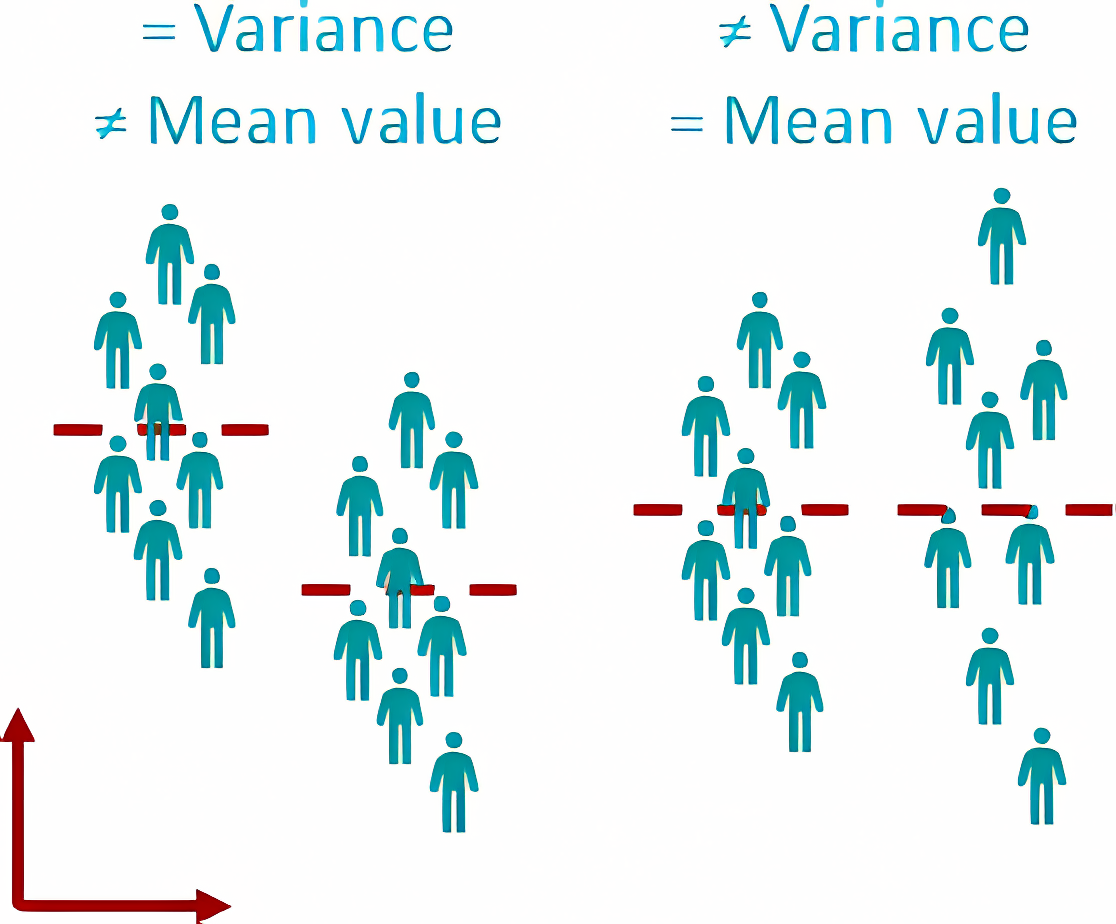
Teste t para duas amostras indep.
Independent-samples t test
Two Sample t-test
data: CRC by Estacao
t = 4.1524, df = 49, p-value = 0.000131
alternative hypothesis: true difference in means between group Chuvosa and group Seca is not equal to 0
95 percent confidence interval:
0.2242132 0.6447619
sample estimates:
mean in group Chuvosa mean in group Seca
3.695357 3.260870 Teste t para duas amostras indep.
Independent-samples t test
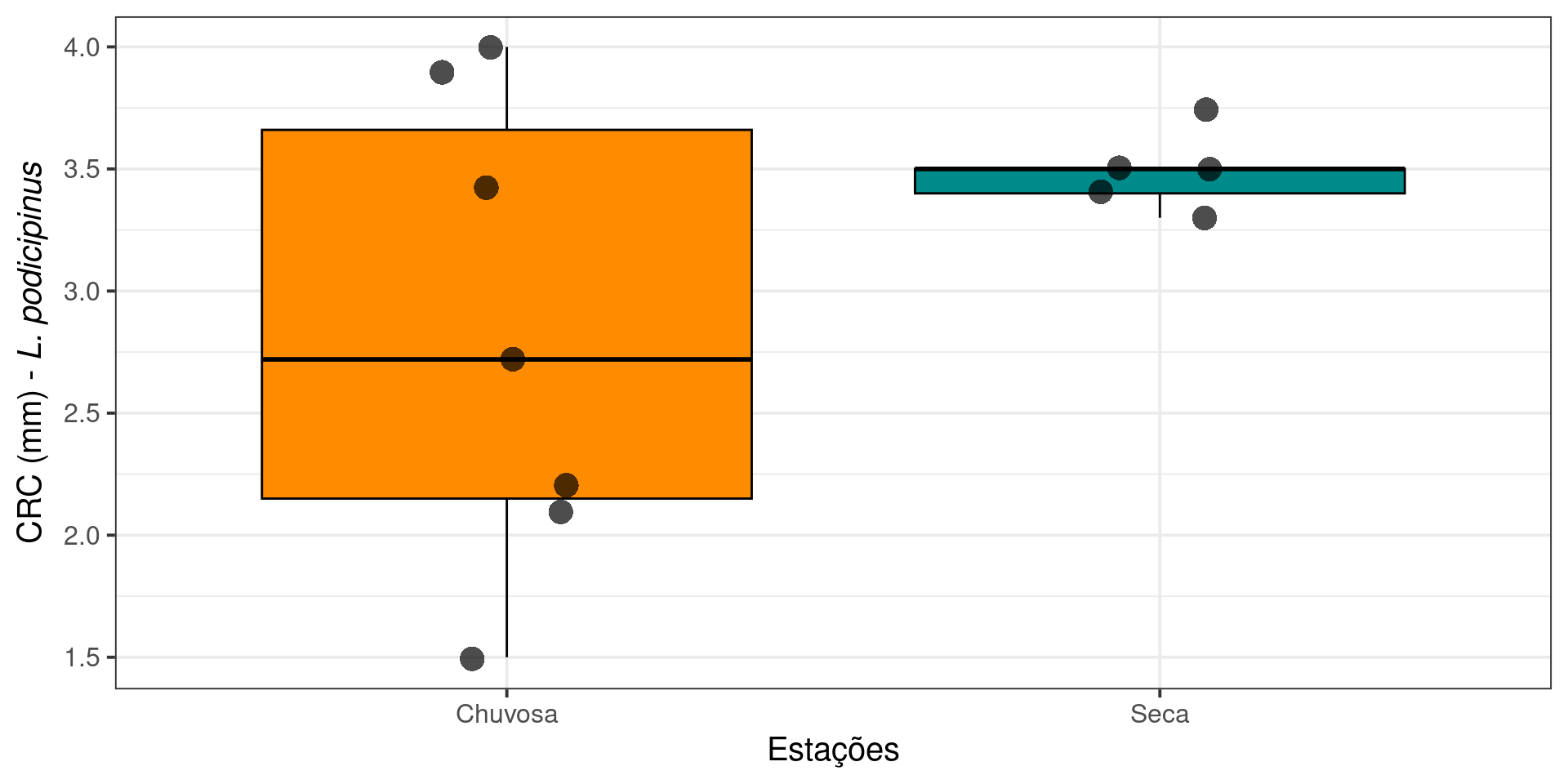
Leptodactylus podicipinus
Comprimento rostro-cloacal (CRC)


Teste t para duas amostras indep.
Independent-samples t test
| Estacao | mean | sd |
|---|---|---|
| Chuvosa | 2.83 | 0.96 |
| Seca | 3.49 | 0.17 |
Teste t para duas amostras indep.
Independent-samples t test
## Teste de normalidade dos residuos - H0: distribuicao dos residuos e normal
residuos <- residuals(lm(CRC ~ Estacao, data = teste_t_var_diferente))
shapiro.test(residuos)
Shapiro-Wilk normality test
data: residuos
W = 0.96272, p-value = 0.8219
Teste t para duas amostras indep.
Independent-samples t test
## Teste de homogeneidade de variancia - H0: variancia e homogenea
car::leveneTest(CRC ~ as.factor(Estacao), data = teste_t_var_diferente)Levene's Test for Homogeneity of Variance (center = median)
Df F value Pr(>F)
group 1 9.8527 0.01053 *
10
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Teste t para duas amostras indep.
Independent-samples t test
Two Sample t-test
data: CRC by Estacao
t = -1.4853, df = 10, p-value = 0.1683
alternative hypothesis: true difference in means between group Chuvosa and group Seca is not equal to 0
95 percent confidence interval:
-1.6393804 0.3279518
sample estimates:
mean in group Chuvosa mean in group Seca
2.834286 3.490000 Teste t para duas amostras indep.
Independent-samples t test
Welch Two Sample t-test
data: CRC by Estacao
t = -1.7633, df = 6.4998, p-value = 0.1245
alternative hypothesis: true difference in means between group Chuvosa and group Seca is not equal to 0
95 percent confidence interval:
-1.5489301 0.2375016
sample estimates:
mean in group Chuvosa mean in group Seca
2.834286 3.490000 Teste t para duas amostras indep.
Independent-samples t test
Teste t para duas amostras indep.
Conclusão
Os machos de P. nattereri coletados na estação chuvosa foram em média 0,43 mm maiores do que os machos coletados na estação seca (t49 = 4,15, P < 0,001)
As fêmeas de L. podicipinus coletadas na estação chuvosa não são maiores do que as fêmeas coletadas na estação seca, apesar de possuírem maior variância, o que pode ser biologicamente interessante
Teste t

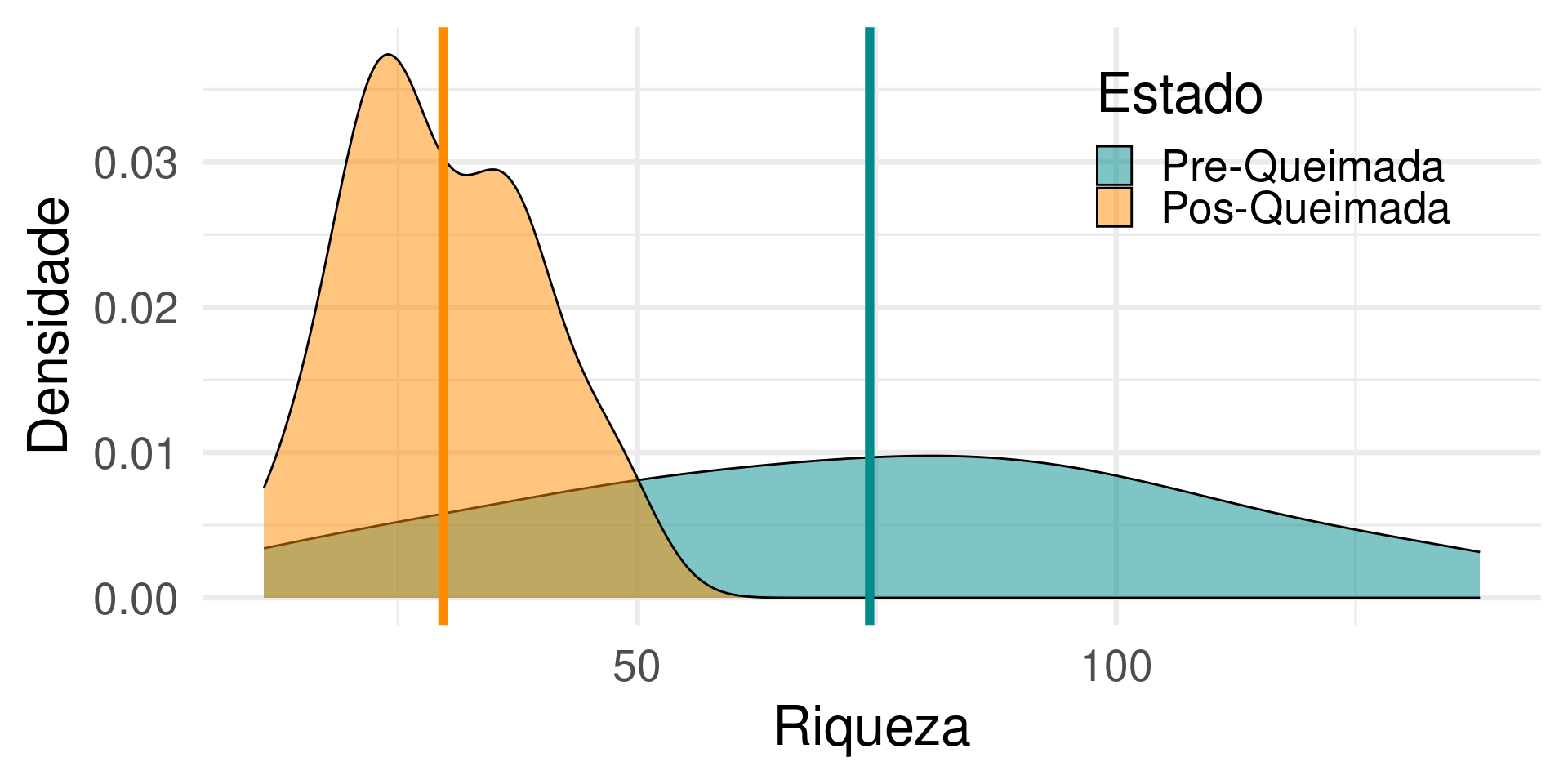
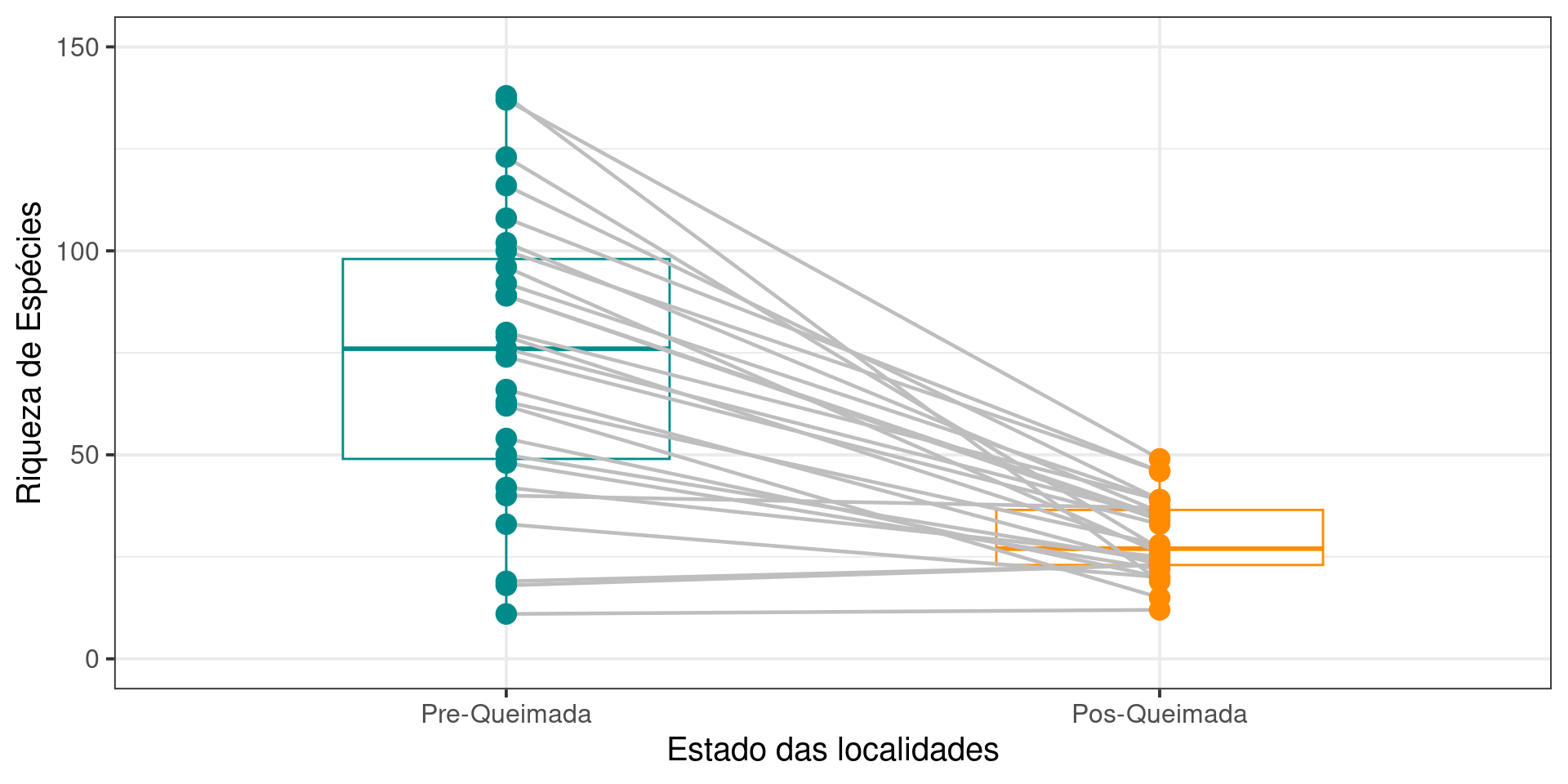
Área queimada
Número de espécies de artrópodes


Teste t para duas amostras pareadas
Paired-samples t test
| Estado | mean | sd |
|---|---|---|
| Pre-Queimada | 74.26 | 35.16 |
| Pos-Queimada | 29.70 | 9.71 |
Teste t para duas amostras indep.
Paired-samples t test
## Teste de normalidade dos residuos - H0: é normal
residuos <- residuals(lm(Riqueza ~ Estado, data = teste_t_pareado))
shapiro.test(residuos)
Shapiro-Wilk normality test
data: residuos
W = 0.96471, p-value = 0.1121
Teste t para duas amostras indep.
Paired-samples t test
## Teste de homogeneidade de variancia - H0: tem homogeneidade
car::leveneTest(Riqueza ~ as.factor(Estado), data = teste_t_pareado)Levene's Test for Homogeneity of Variance (center = median)
Df F value Pr(>F)
group 1 26.138 4.64e-06 ***
52
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Teste t para duas amostras indep.
Paired-samples t test
Paired t-test
data: Riqueza by Estado
t = -7.5788, df = 26, p-value = 4.803e-08
alternative hypothesis: true mean difference is not equal to 0
95 percent confidence interval:
-56.63994 -32.47117
sample estimates:
mean difference
-44.55556 Teste t para duas amostras indep.
Paired-samples t test
Teste t para duas amostras indep.
Conclusão
Os resultados mostram que as localidades após as queimadas apresentam em média 44,5 espécies de artrópodes a menos do que antes das queimadas
Dúvidas?
Resumindo
Resumindo
Linguagem R
Análise Exploratória de Dados (AED): medidas-resumo, tabelas e gráficos
Análise de dados univariados
Pergunta e hipótese
Hipótese ecológica e hipótese estatística
População e amostra
Parâmetro e estatística (estimador)
Média, variância, desvio padrão, erro padrão
Inferência estatística: intervalo de confiança e teste de hipóteses
Distribuição z e t e valor de p
Teste t para uma amostra
Teste t para duas amostras indep.
Teste t para duas amostras pareadas
Material de estudo
Material de estudo
Muito obrigado!
Slides por Maurício Vancine, feitos com Quarto. Código disponível no GitHub.