Modelagem de Distribuição de Espécies e algumas aplicações
Novembro de 2022
Licença: CC BY-NC-SA
Tópicos
- Apresentações
- Visão geral
- Conceitualização
- Preparação dos dados
- Ajuste dos modelos
- Avaliação dos modelos
- Predição e consenso dos modelos
- Aplicações
Maurício Vancine
Análises Ecológicas no R

15 capítulos: linguagem R, tidyverse, perguntas em ecologia, análises univariadas, multivariadas e geoespaciais
Visão geral
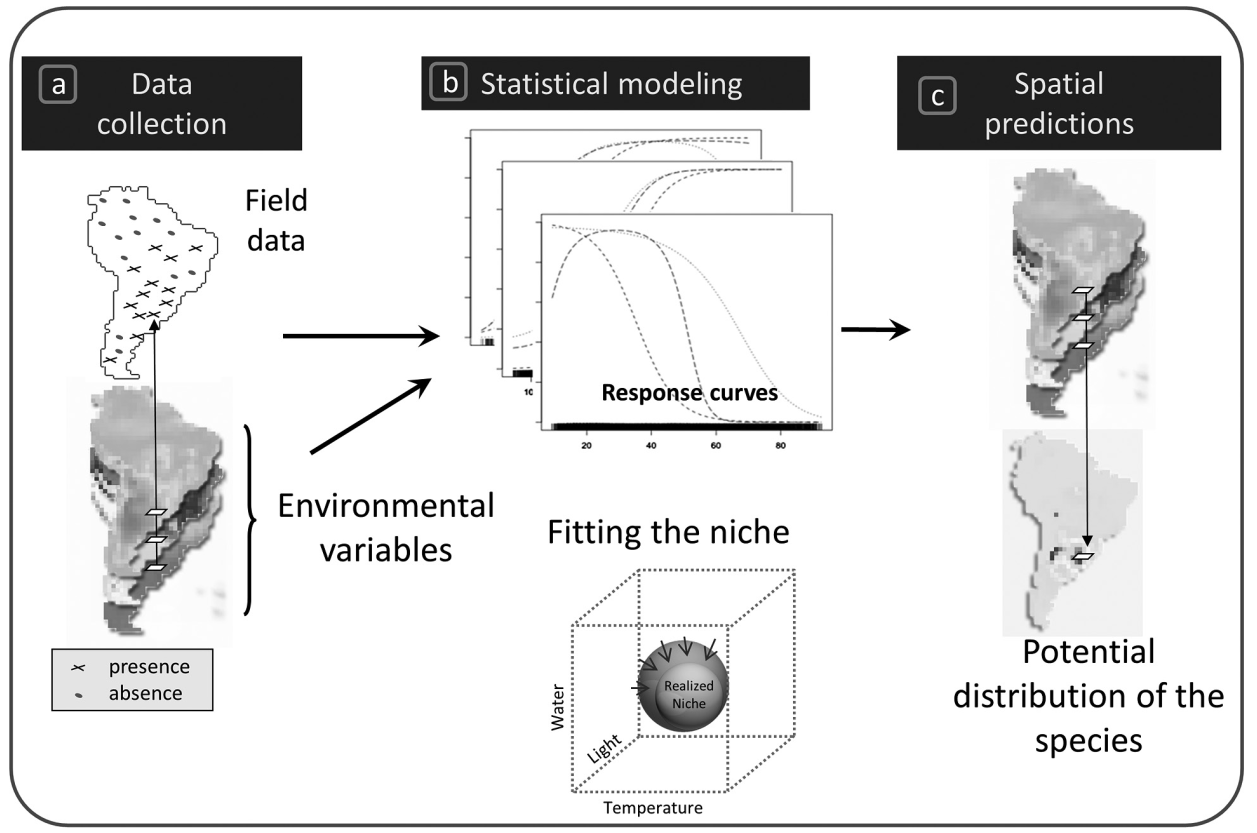
Visão geral
Protocolo padrão para modelos de distribuição de espécies
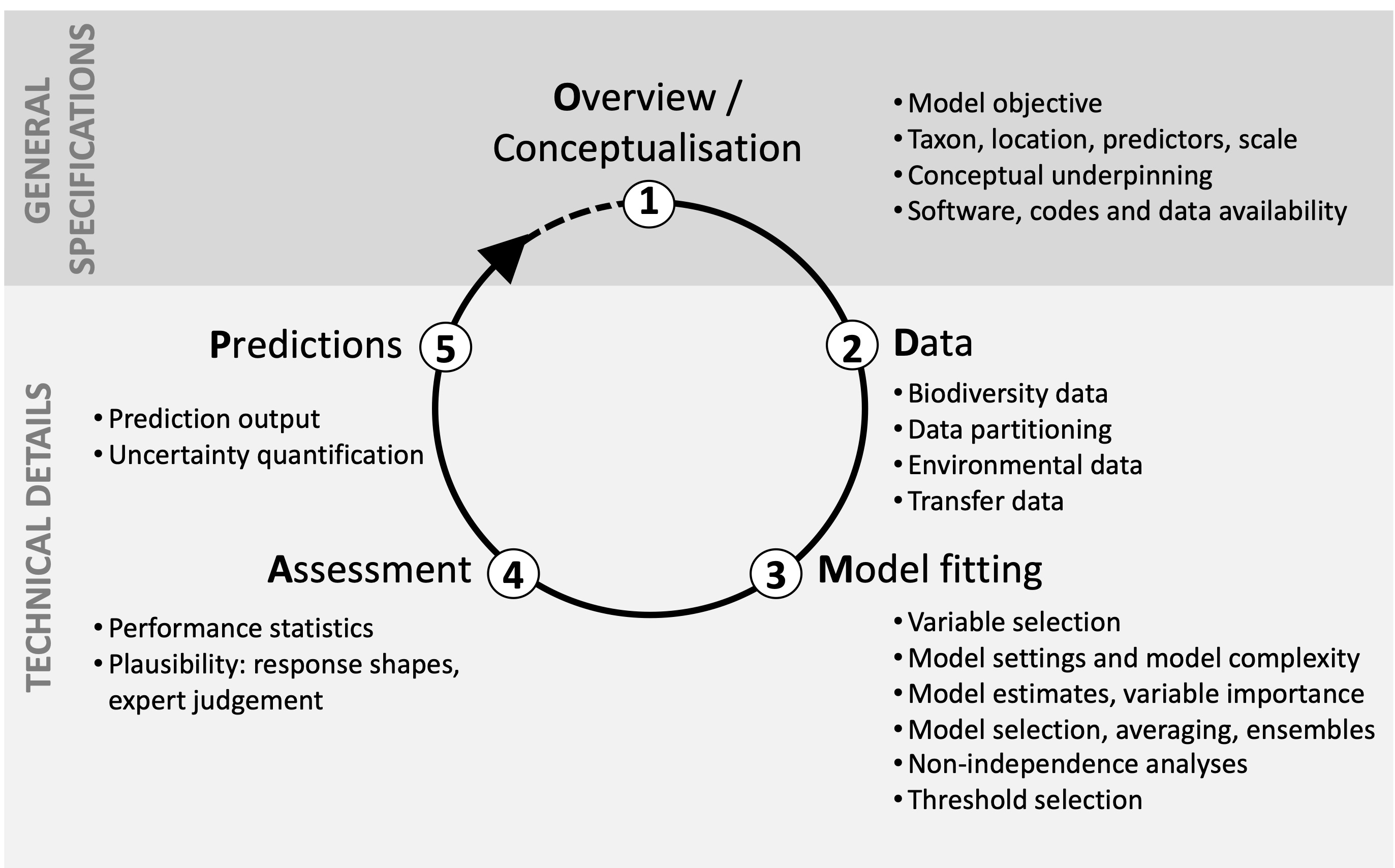
Visão geral
Protocolo padrão para modelos de distribuição de espécies
1. Conceitualização
Terminologia
1. Modelos de Distribuição de Espécies (Species Distribution Models - SDMs)
- Distribuição da espécie sem transferir para outras condições no espaço ou tempo
- Distribuição com maior precisão incorporando elementos de dispersão e colonização
2. Modelos de Nicho Ecológico (Ecological Niche Models - ENMs)
- Estima as preferências ambientais da espécie para poder transferir para outras condições no espaço ou tempo
3. Modelos de Adequabilidade de Habitat (Habitat Suitability Models - HSMs)
- Reflete melhor a base dos modelos: quantificar a relação espécie-ambiente (habitat)
- Aplicável a entidades que não sejam espécies (e.g. genes ou comunidades)
- Aplicável a casos em que um nicho é parcialmente capturado (área menor que a distribuição total)
Em comum: todos são modelos. Mas o que são modelos?
Modelos [científicos]
Idealizações da realidade com o objetivo de entendê-la

(…) essentially, all models are wrong, but some are useful. George E. P. Box (1919-2013)
Objetivo
Predizer a distribuição de espécies (ou entidades) a partir do nicho ecológico
Entretanto…
Estimar/analisar o nicho das espécies sem inferir sua distribuição geográfica
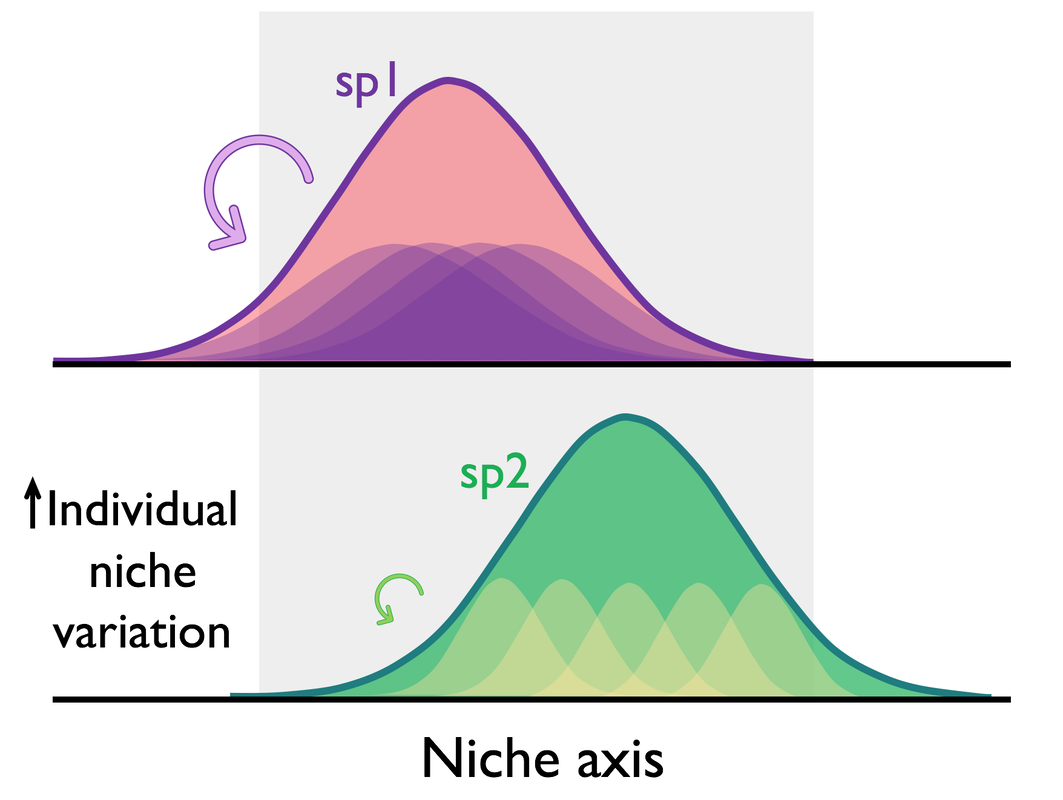
- Sobreposição de nicho
- Amplitude do nicho
- Montagem de comunidades
- Respostas ambientais
- Diversidade funcional
- …
Entretanto…
Inferir a distribuição das espécies sem estimar o nicho ecológico
- Buffers
- Densidade de Kernel
- Mínimo Polígono Convexo (MPC)
- Interpolações (IDW, Spline, Krigagem)
- Opinião de especialistas (IUCN)
- …
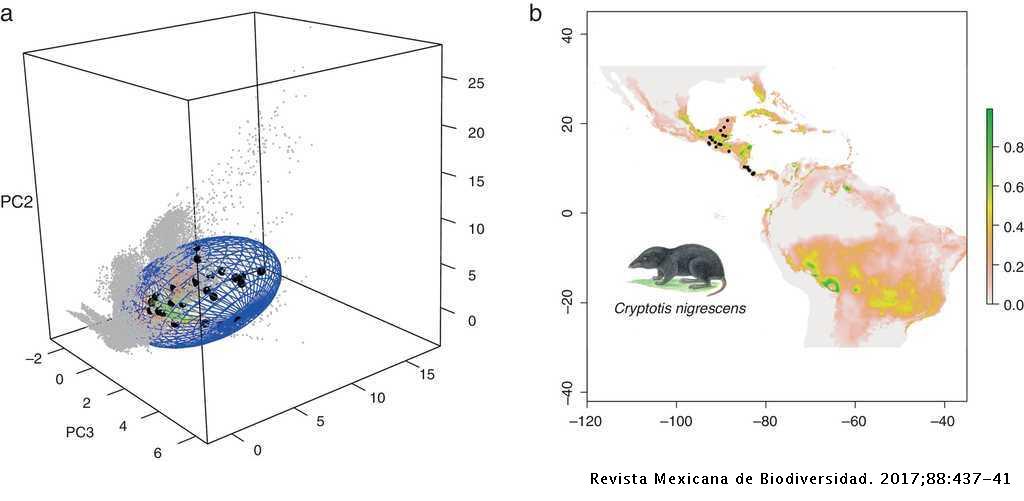
Nicho ecológico
- Modelagem de nicho ecológico
- Coexistência entre espécies
- Metacomunidades
Modelagem de nicho ecológico
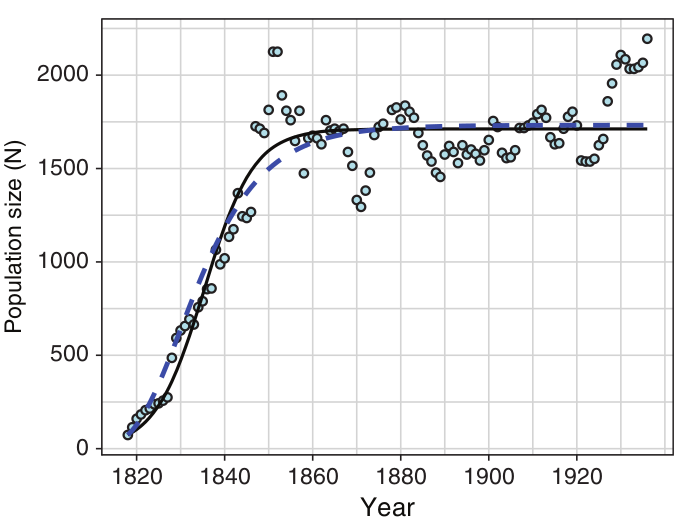
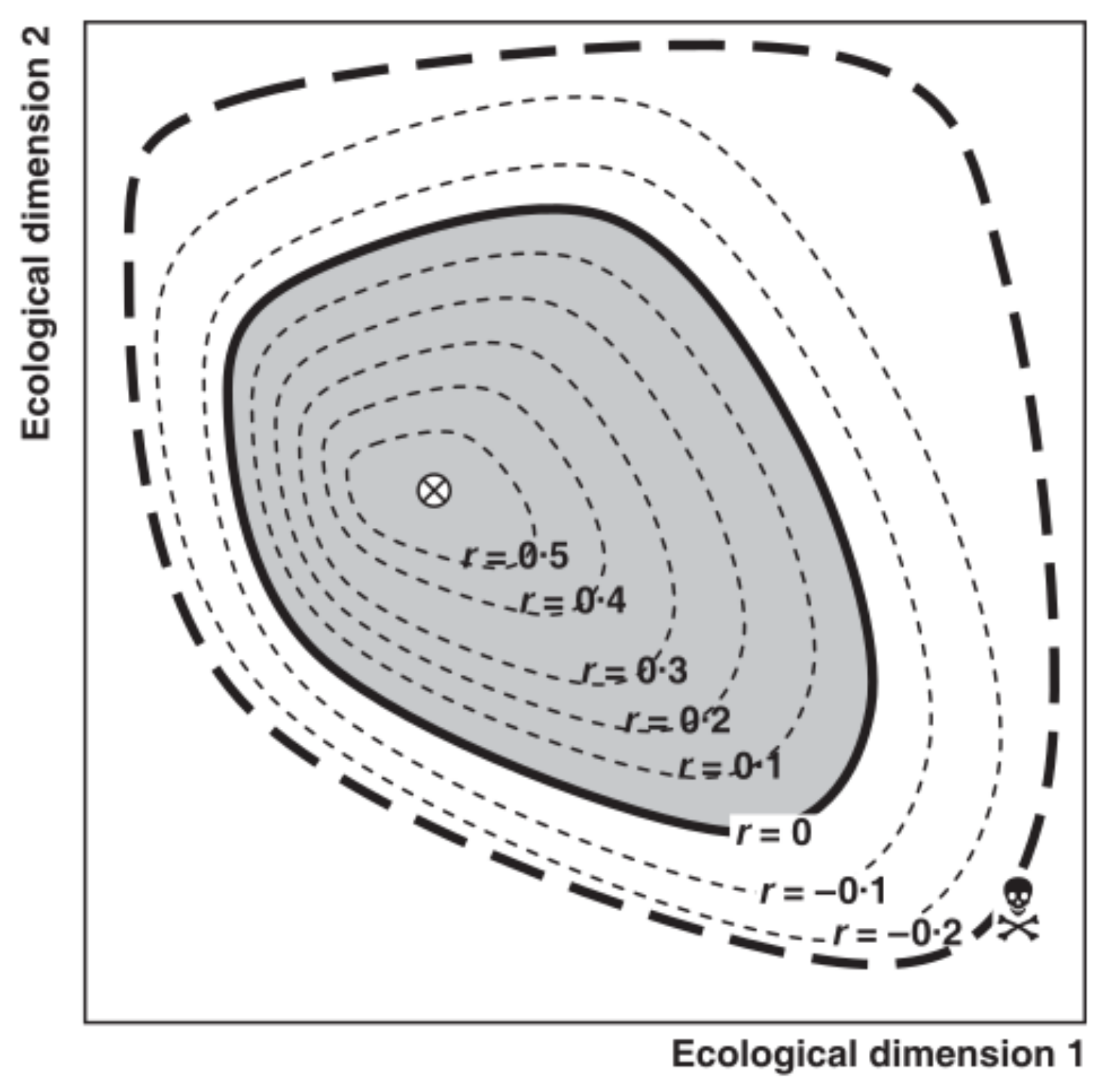
Combinações de condições que permitem a uma população sobreviver e crescer (r >= 0) em um determinado lugar
Modelagem de nicho ecológico
- Podemos associar a taxa de crescimento intrínseca (r) com as condições favoráveis
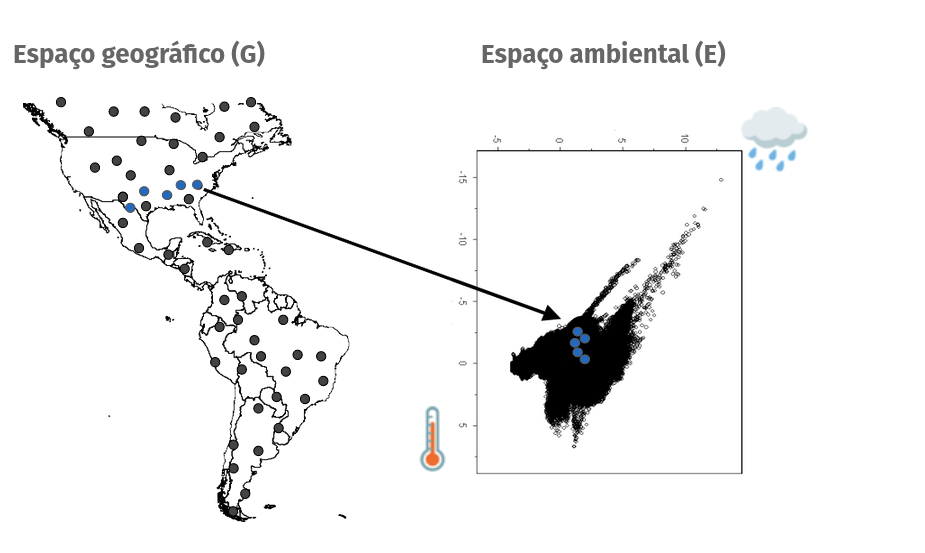
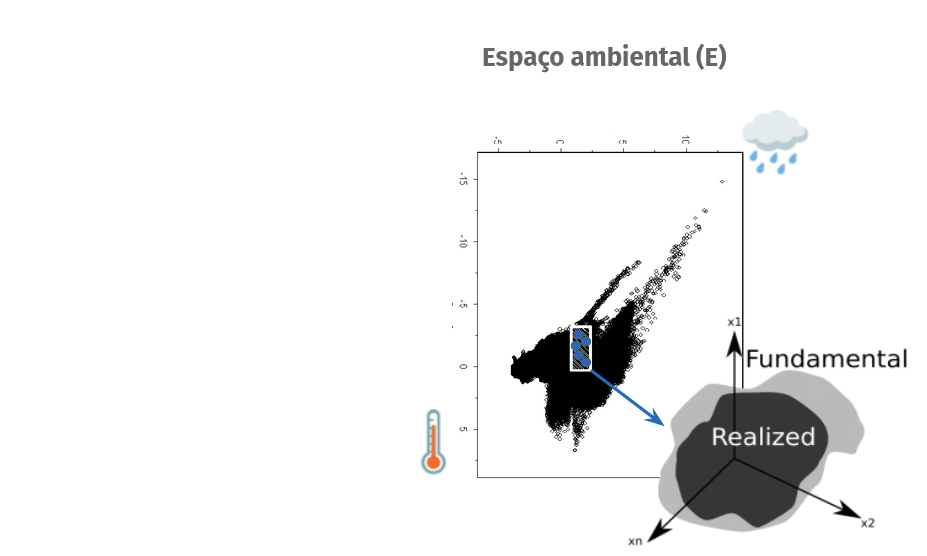
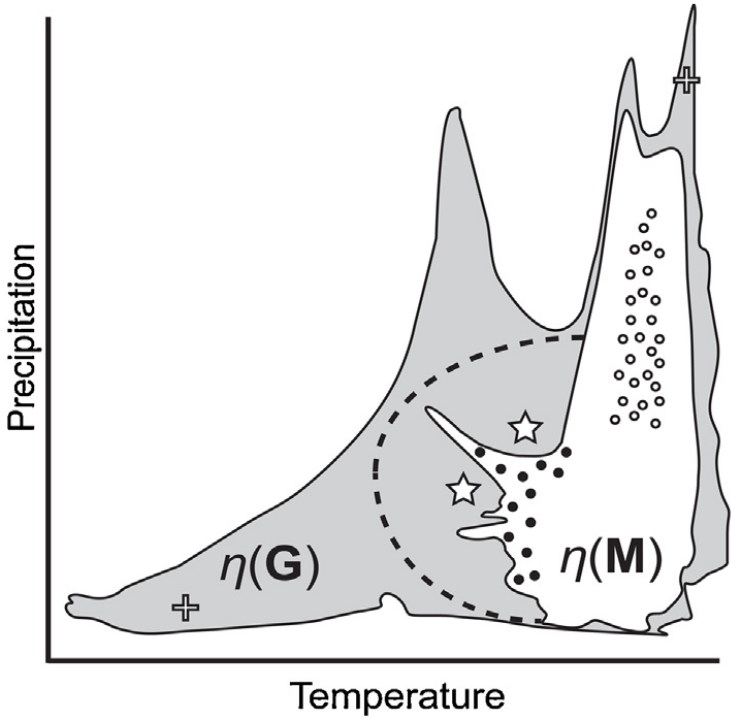
- O modelo então estima as condições favoráveis (nicho ecológico) no Espaço Ambiental (E) e prediz a distribuição da espécie Espaço Geográfico (G)
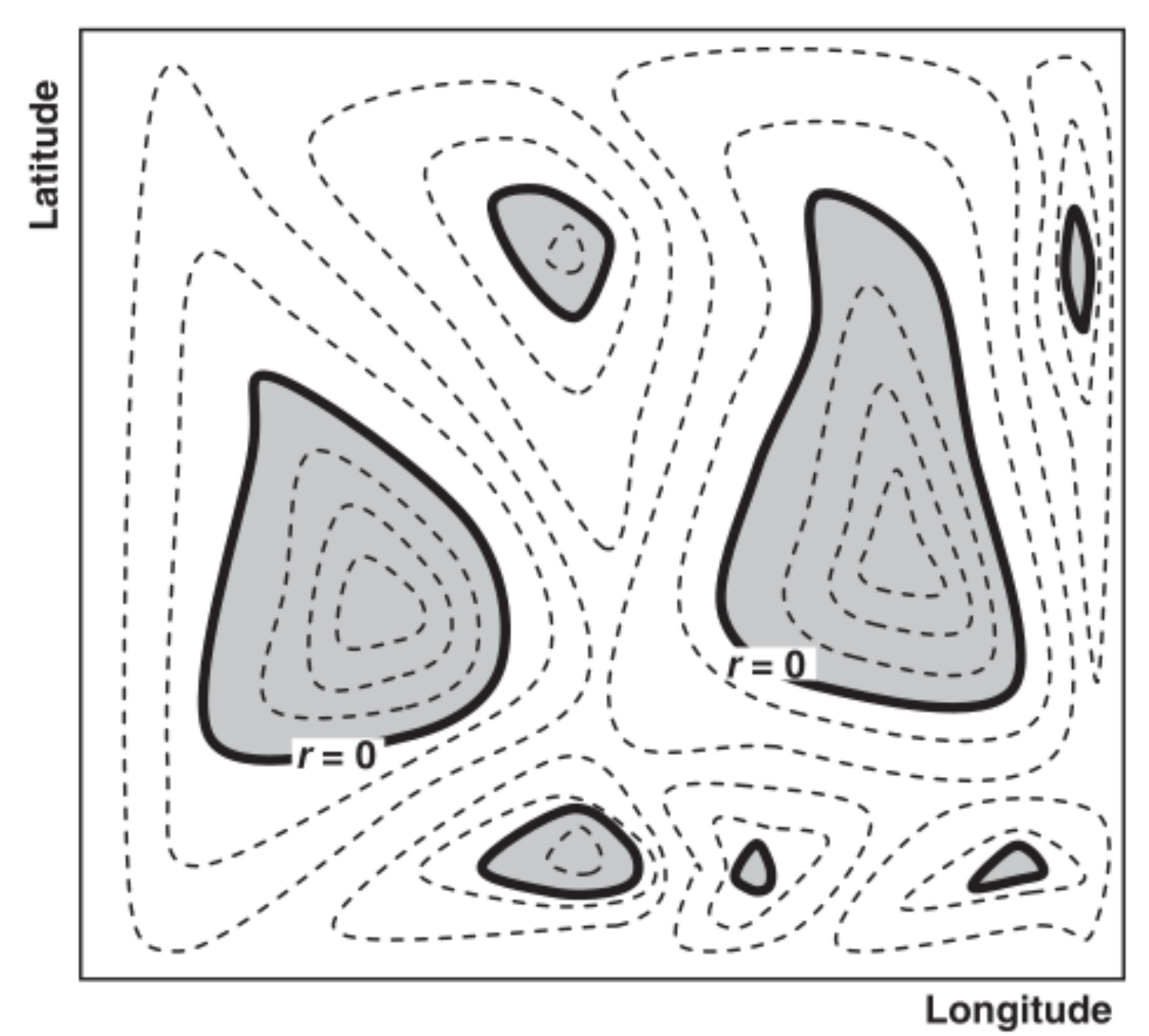
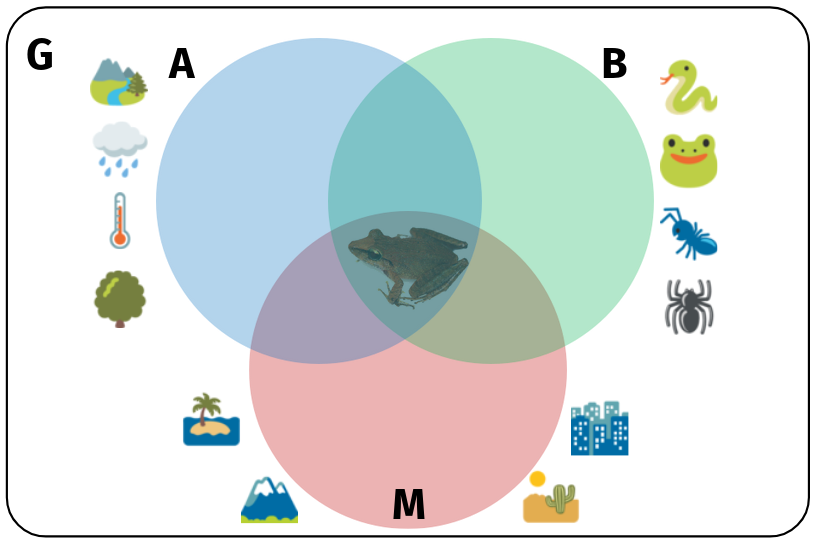
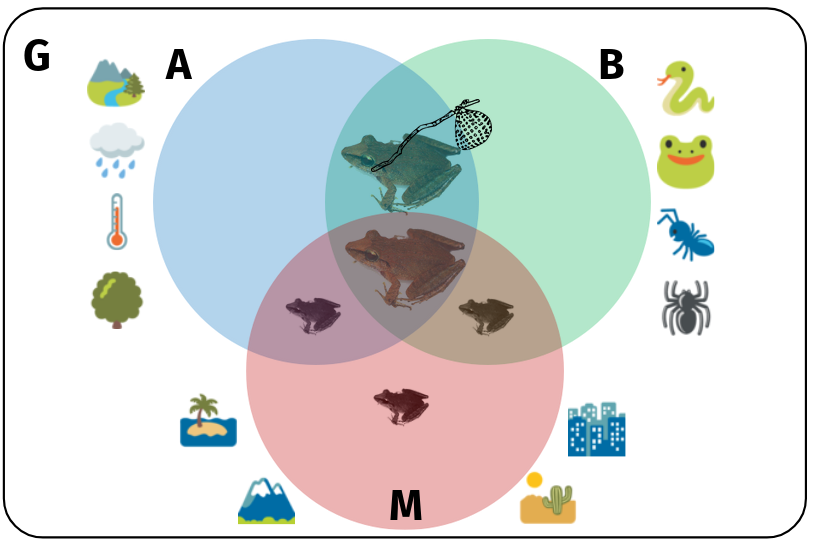
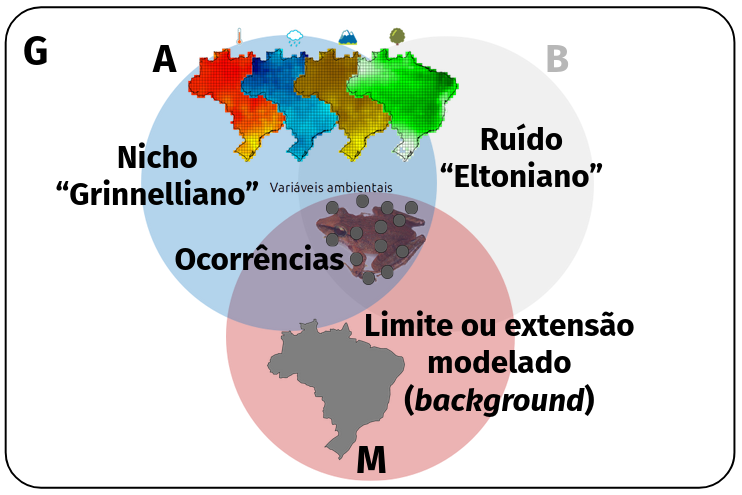
Espaço geográfico (G)

Distribuição de espécies

Condições abióticas (A)
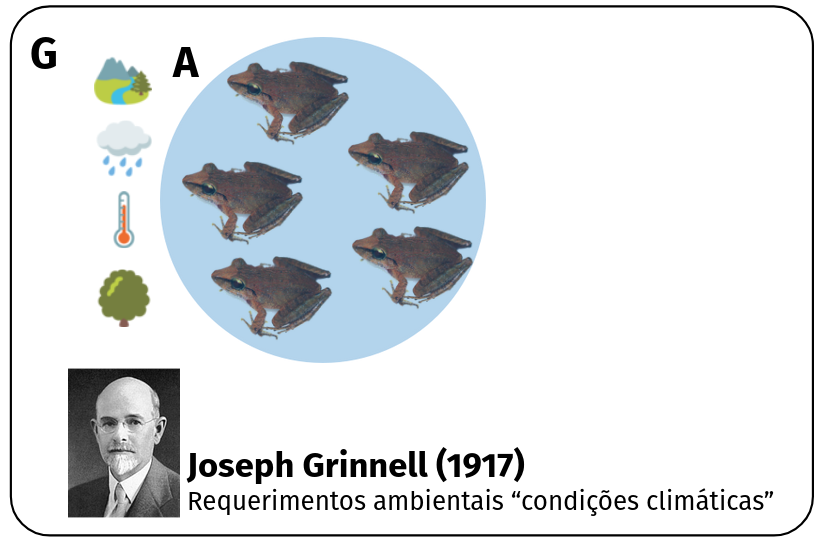
Condições bióticas (B)
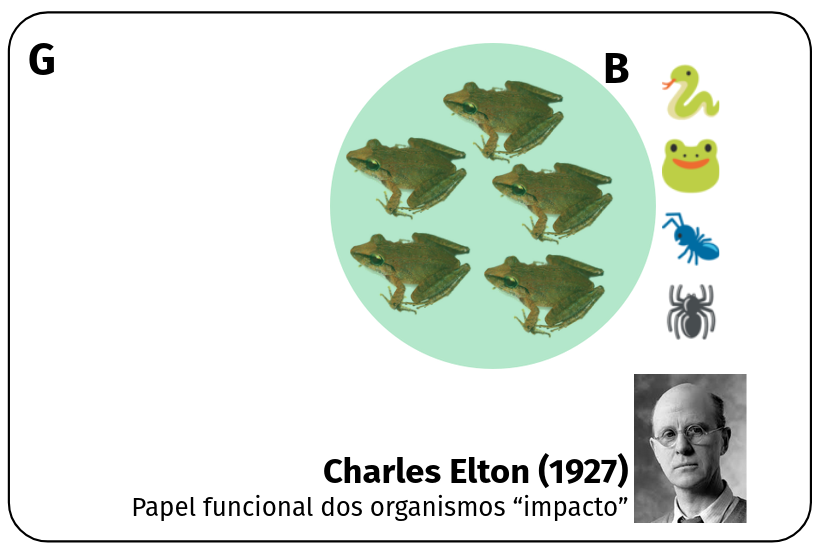
Condições abióticas (A) e bióticas (B)
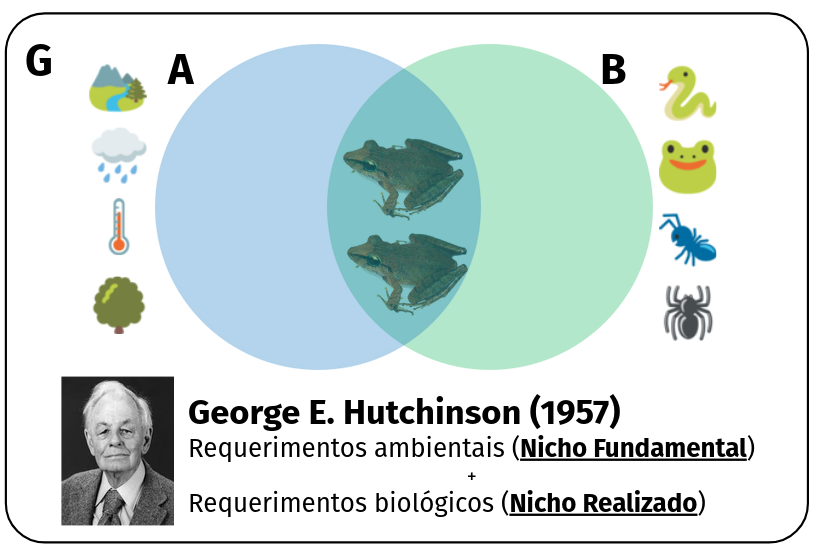
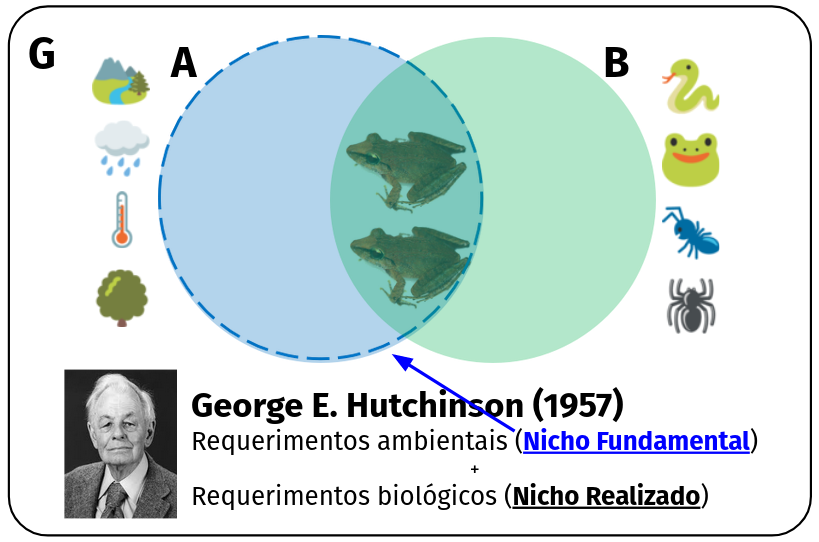
Nicho fundamental
Todas as condições ambientais favoráveis
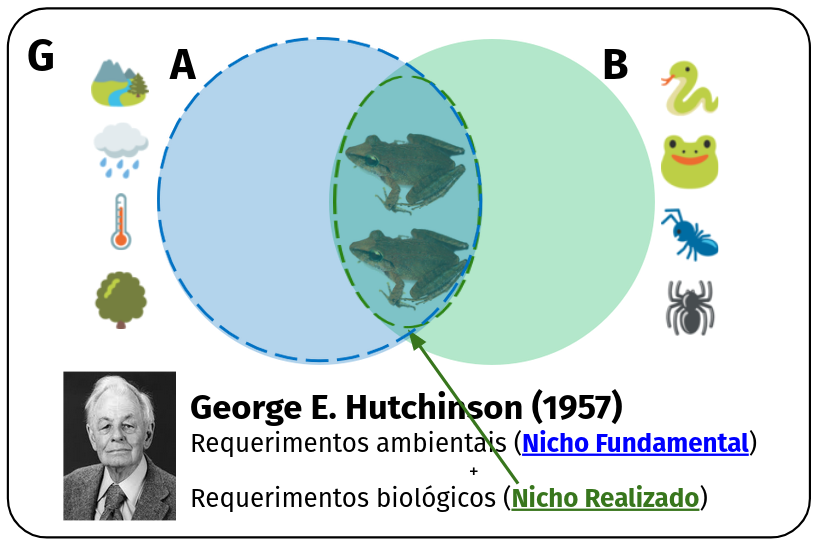
Nicho realizado
Condições ambientais favoráveis limitadas pela interação
Nicho ecológico
Hipervolume n-dimensional
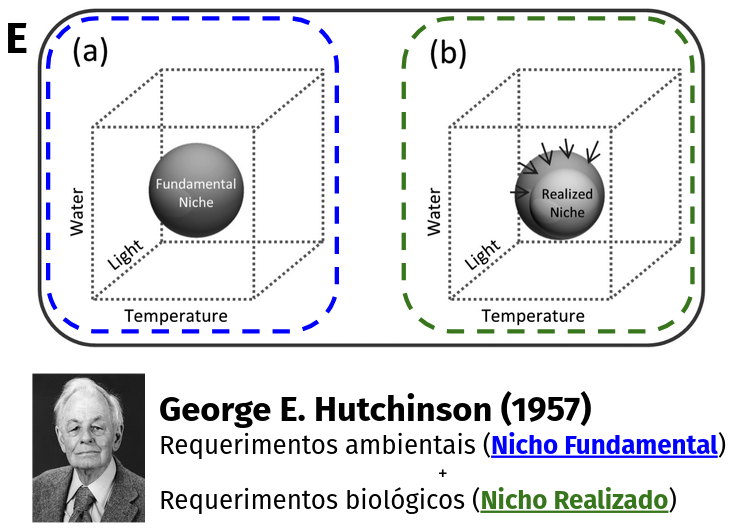
Dualidade Hutchinsoniana
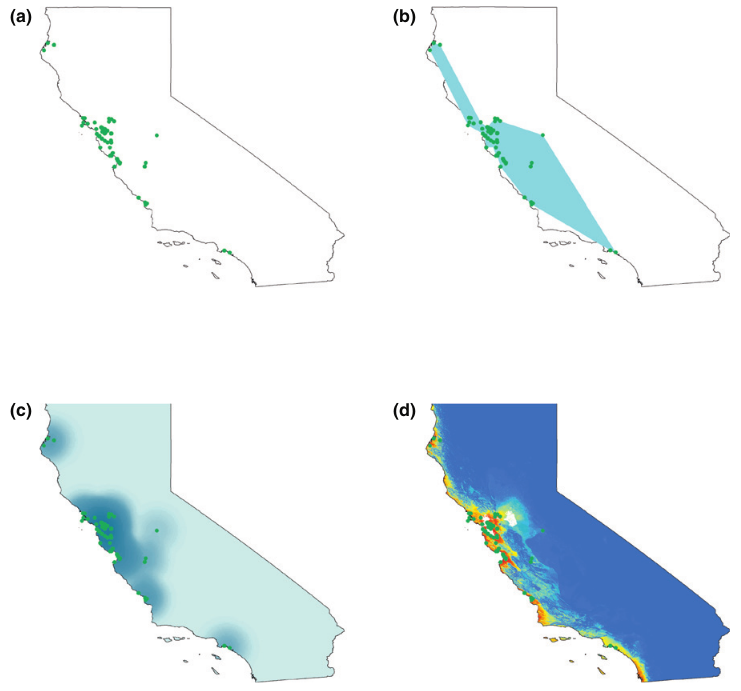
Inferir a distribuição geográfica de uma espécie a partir do nicho ecológico
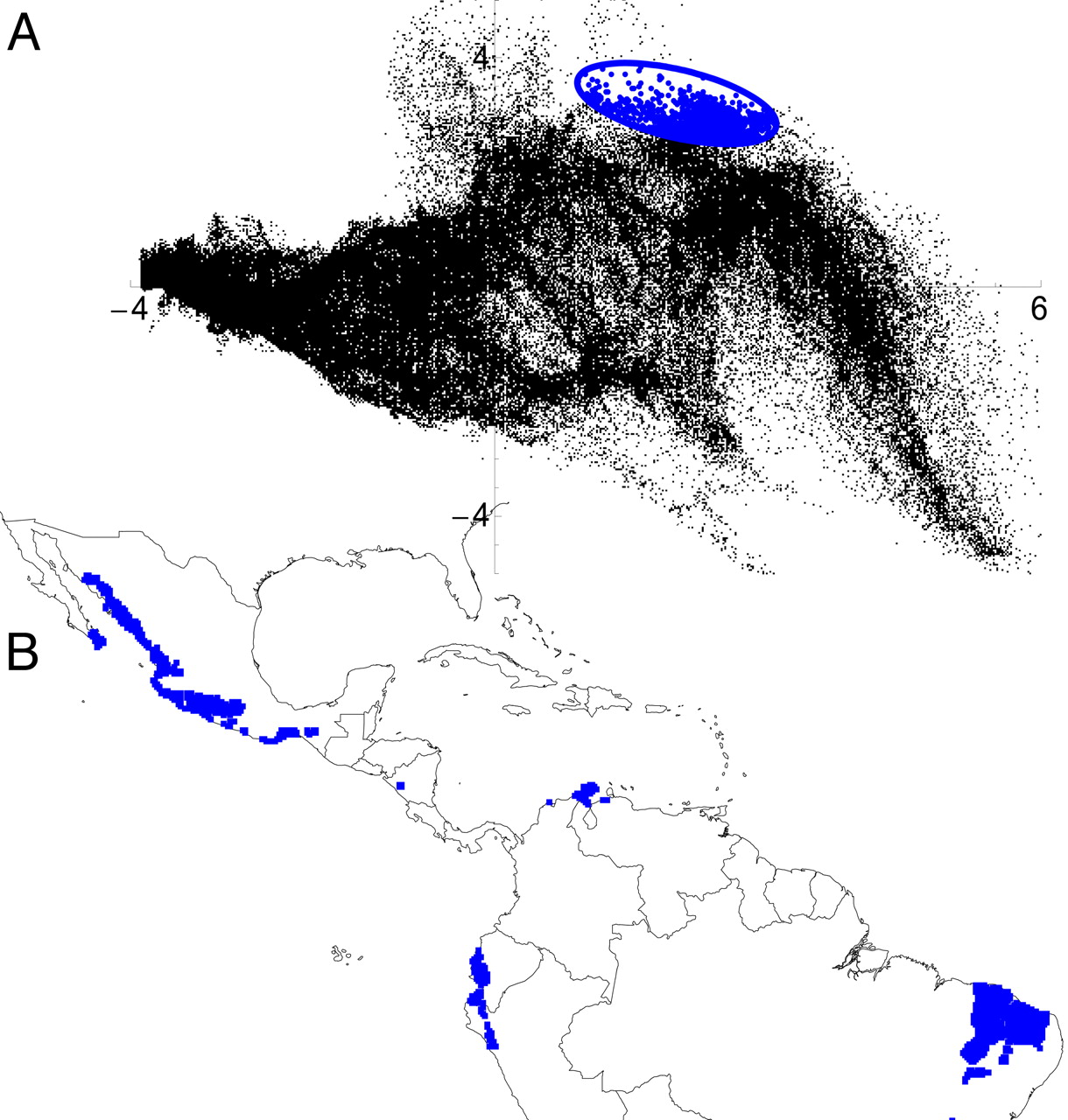
Ocorrências no espaço geográfico
Condições ambientais
Estimativa do nicho realizado
Predição geográfica
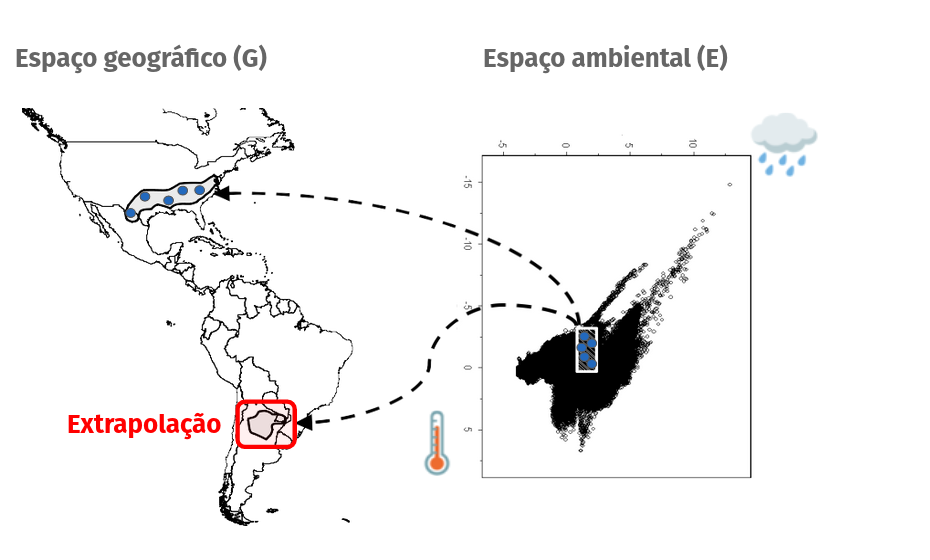
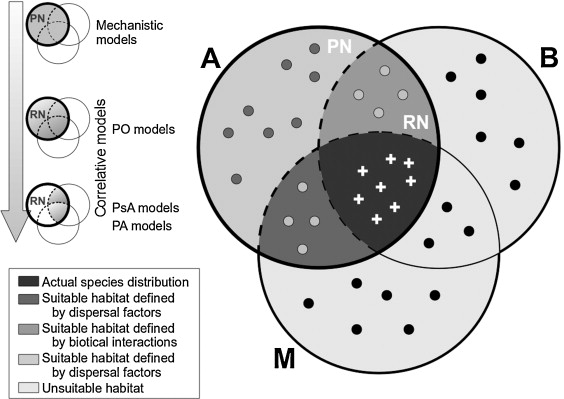
Como contornar essa extrapolação?
Área acessível (movimento) (M)
Área acessível (movimento) (M)
O limite deve considerar as condições de dispersão históricas das espécies
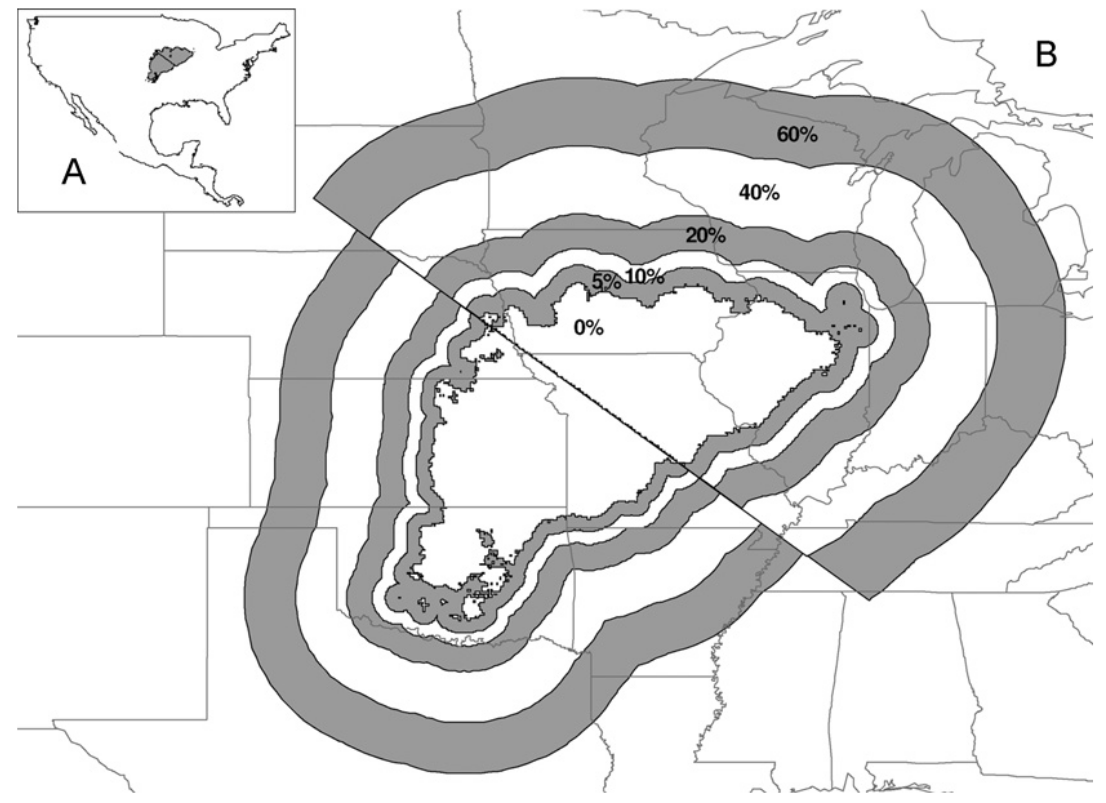
Populações fonte e ralo (source-sink)
Populações fonte e ralo (source-sink)
Populações fonte e ralo (source-sink)
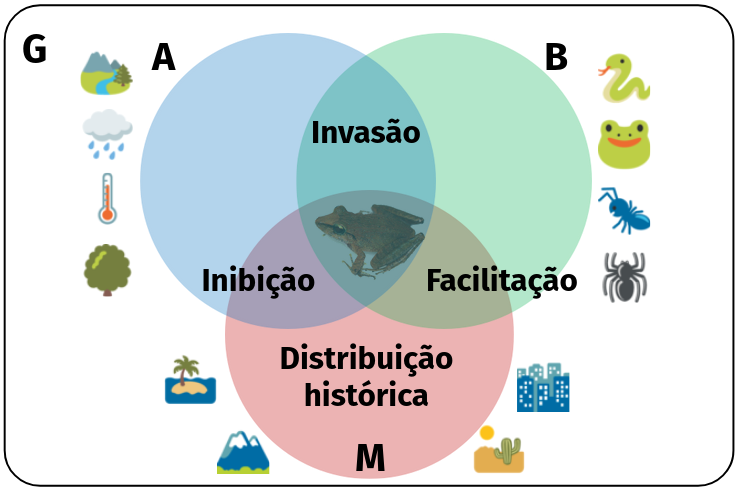
Invasão
- Espécies exóticas
- Ecossistemas
- Extinção
- Hibridização
Distribuição histórica
- Paleoclima
- Refúfios
- Conexões
- Dispersão
- Vicariância

Populações fonte e ralo (source-sink)
Inibição
- Competição
- Predação
- Parasitismo
- Amensalismo
Facilitação
- Mutualismo
- Cooperação
- Comensalismo
- Inquilinismo
- Epifitismo

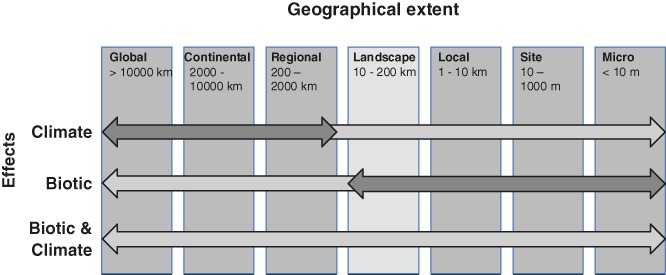
Estabilidade
Variações das condições abióticas e bióticas em função do tempo e do espaço
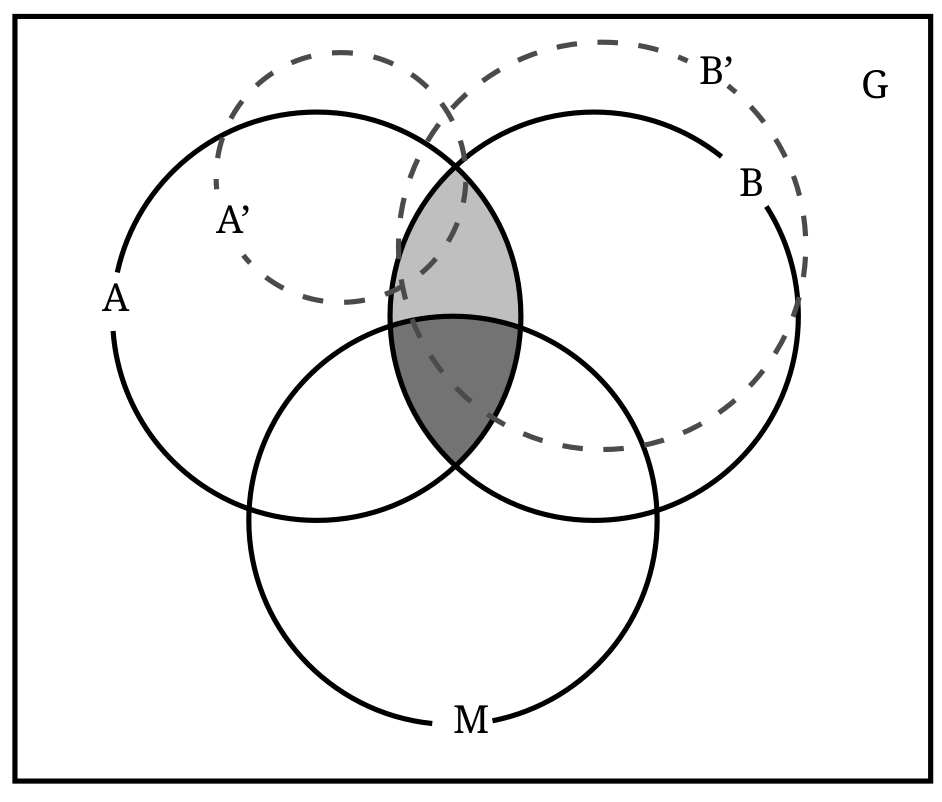
O que de fato modelamos?
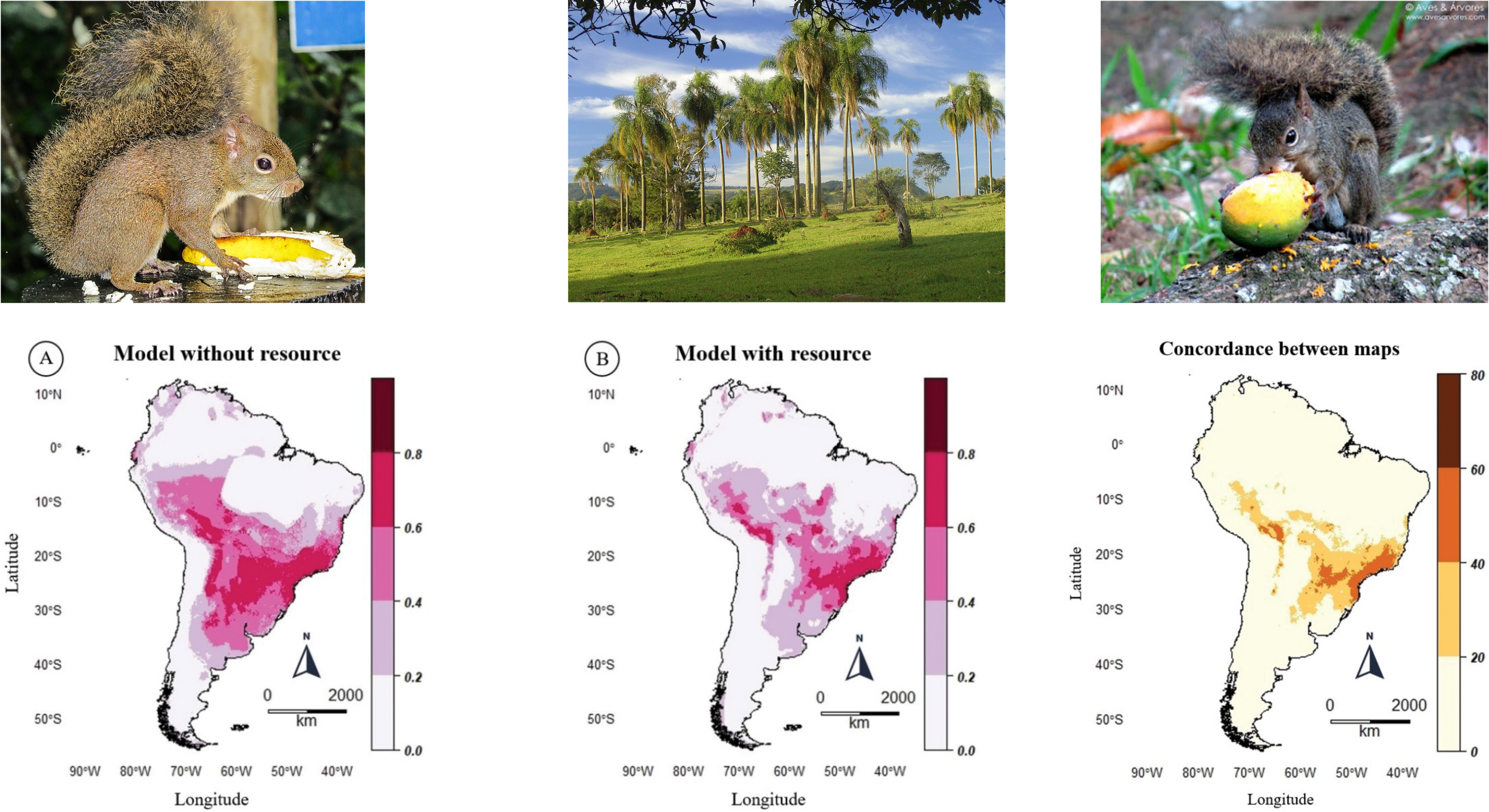
Ruído Eltoniano
Desconsideramos as condições bióticas
Entretanto…
Esquilo: Sciurus aestuans (catinguelê)
Palmeira: Syagrus romanzoffiana (jerivá)
Pressupostos
- Estabilidade do nicho ecológico: as espécies mantêm o mesmo nicho ecológico ao longo do tempo, ou, pelo menos, entre os períodos utilizados na modelagem
- Equilíbio espécie-clima: distribuição de uma espécie é considerada em equilíbrio com o clima quando ela está presente em todas as áreas climaticamente adequadas à sua ocorrência e não ocorre nas regiões inadequadas
- Controle taxonômico: certeza em relação à taxonomia da espécie para as ocorrências (discutível)
- “Ruído Eltoniano”: as interações bióticas não afetam a distribuição da espécie na macroescala
Temas
- Distribuição de espécies
- Padrões de diversidade
- Mudanças climáticas (passado e futuro)
- Invasão biológicas
- Transmissão de doenças
- Interações entre espécies
- Processos de extinção
- Conservação-evolução de nicho
- Estabelecer refúgios climáticos
- Estabelecimento e eficiência de áreas protegidas
Visão geral

Visão geral
Protocolo padrão para modelos de distribuição de espécies

2. Preparação dos dados
Dados de entrada
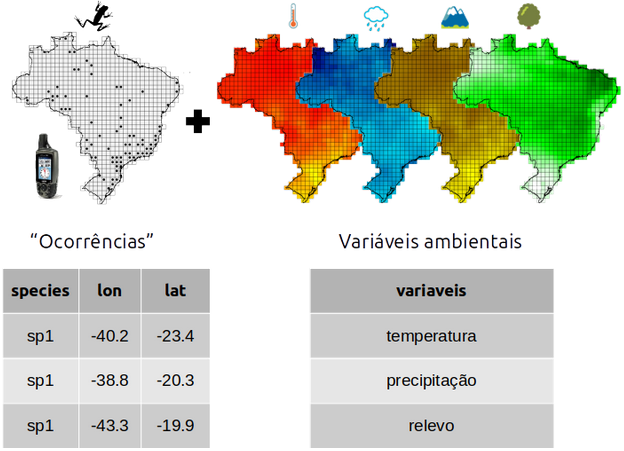
Ocorrências
Formato
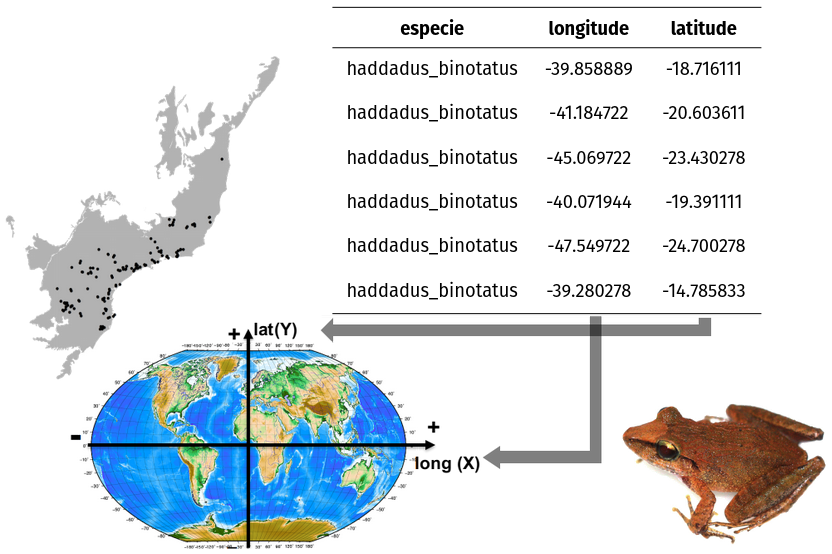
Ocorrências
Fontes
- Coletas em campo (amostragens em campo)
- Literatura (artigos, data papers, …)
- Ciência cidadã (e-Bird, iNaturalist, …)
- Coleções científicas e museus (Museu Nacional, …)
- Banco de dados (GBIF, SpeciesLink, …)



Ocorrências
Bancos de dados
- Global Biodiversity Information Facility (GBIF)
- iNaturalist
- VertNet
- eBird
- iDigBio
- Ocean Biogeographic Information System (OBIS)
- Botanical Information and Ecology Network (BIEN)
- speciesLink
- SIBBr

Ocorrências
Pacotes no R
Ocorrências
Viés de amostragem - Brasil
- 1.144.629 (total) e 882.468 (válidos) ocorrências para 4345 espécies
- Grupos: vertebrados , artropodes e angiospermas
- Todos as ocorrências < 1km de rotas de acesso (estradas e rios)
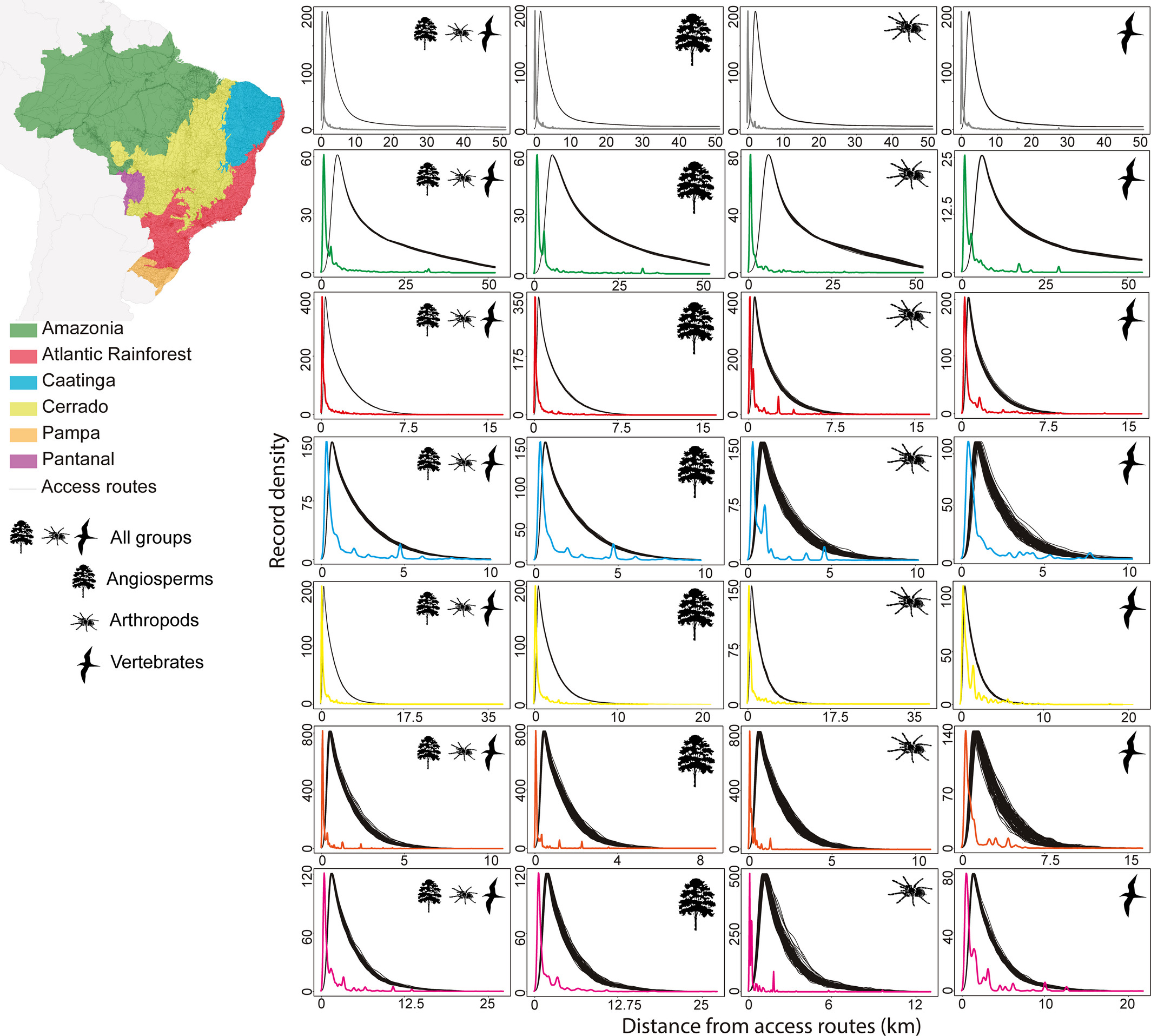
Ocorrências
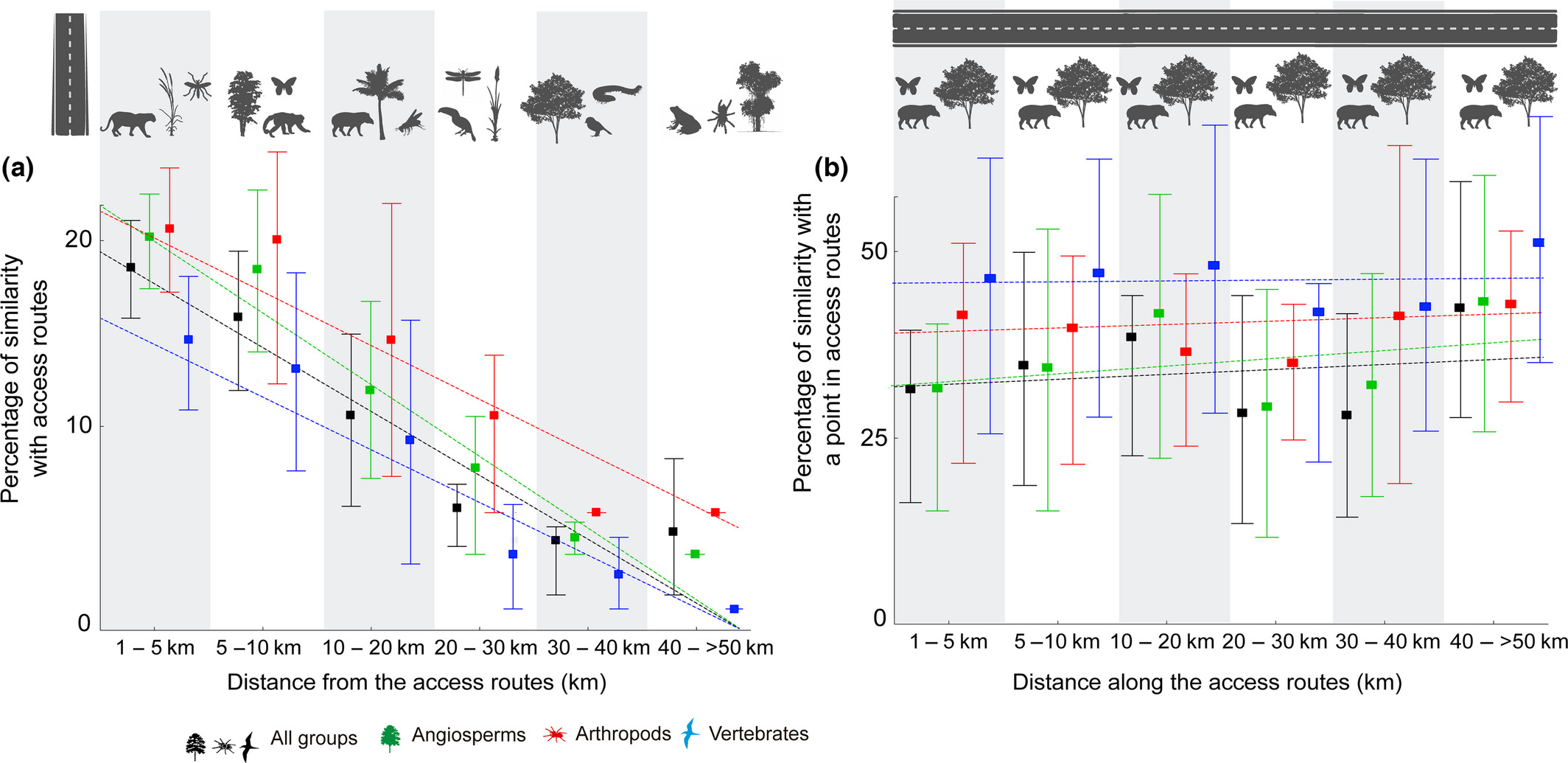
Viés de amostragem - Mundo
- 742 milhões de ocorrências de 374.900 espécies
- Representando 6,74% do globo amostrado
Variáveis ambientais
Raster - contínuos ou categóricos
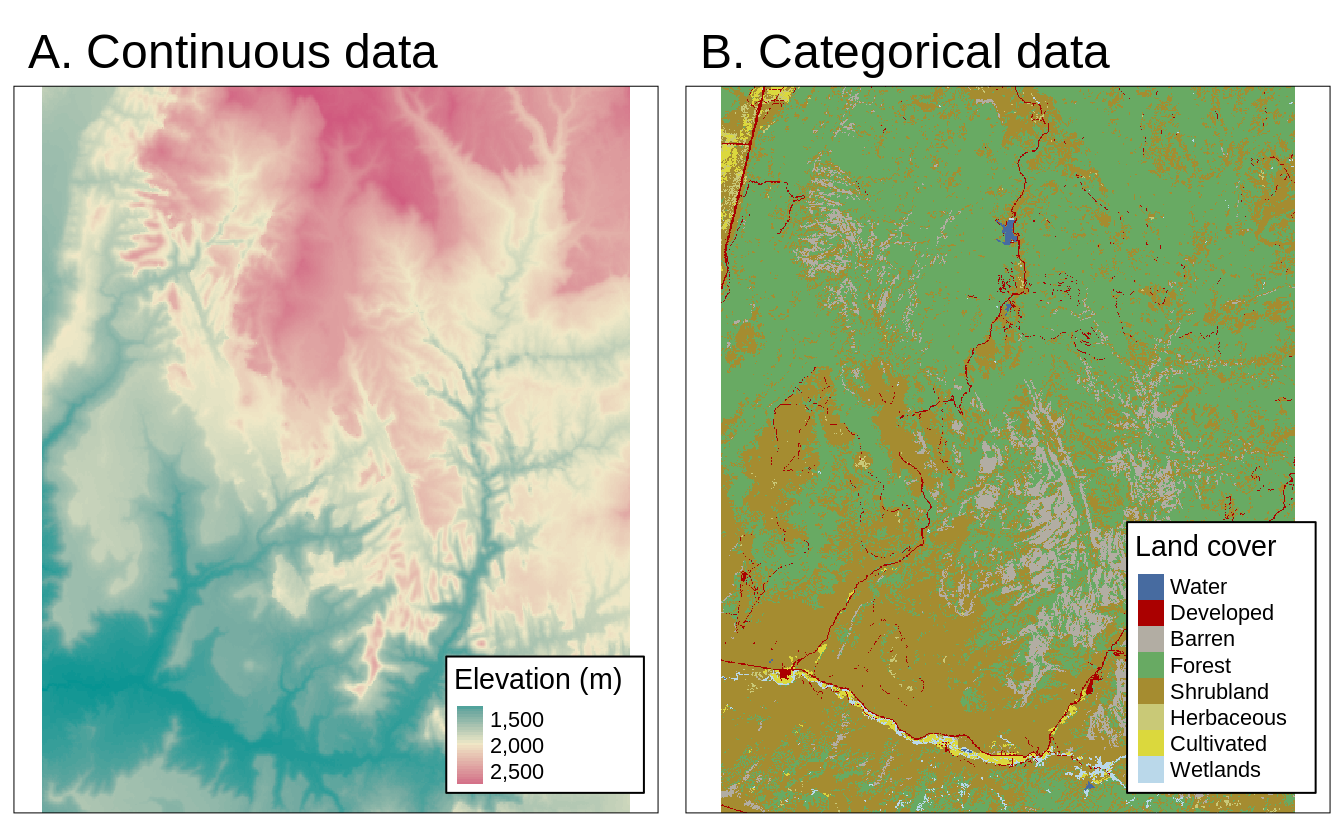
Variáveis ambientais
Bases de dados
- WorldClim
- CHELSA
- ENVIREM
- PaleoClim
- ecoClimate
- EarthEnv
- Land-Use Harmonization (LUH2)
- Global Human Footprint
- SoilGrids
- MARSPEC
- Bio-ORACLE



Variáveis ambientais
Interpolação
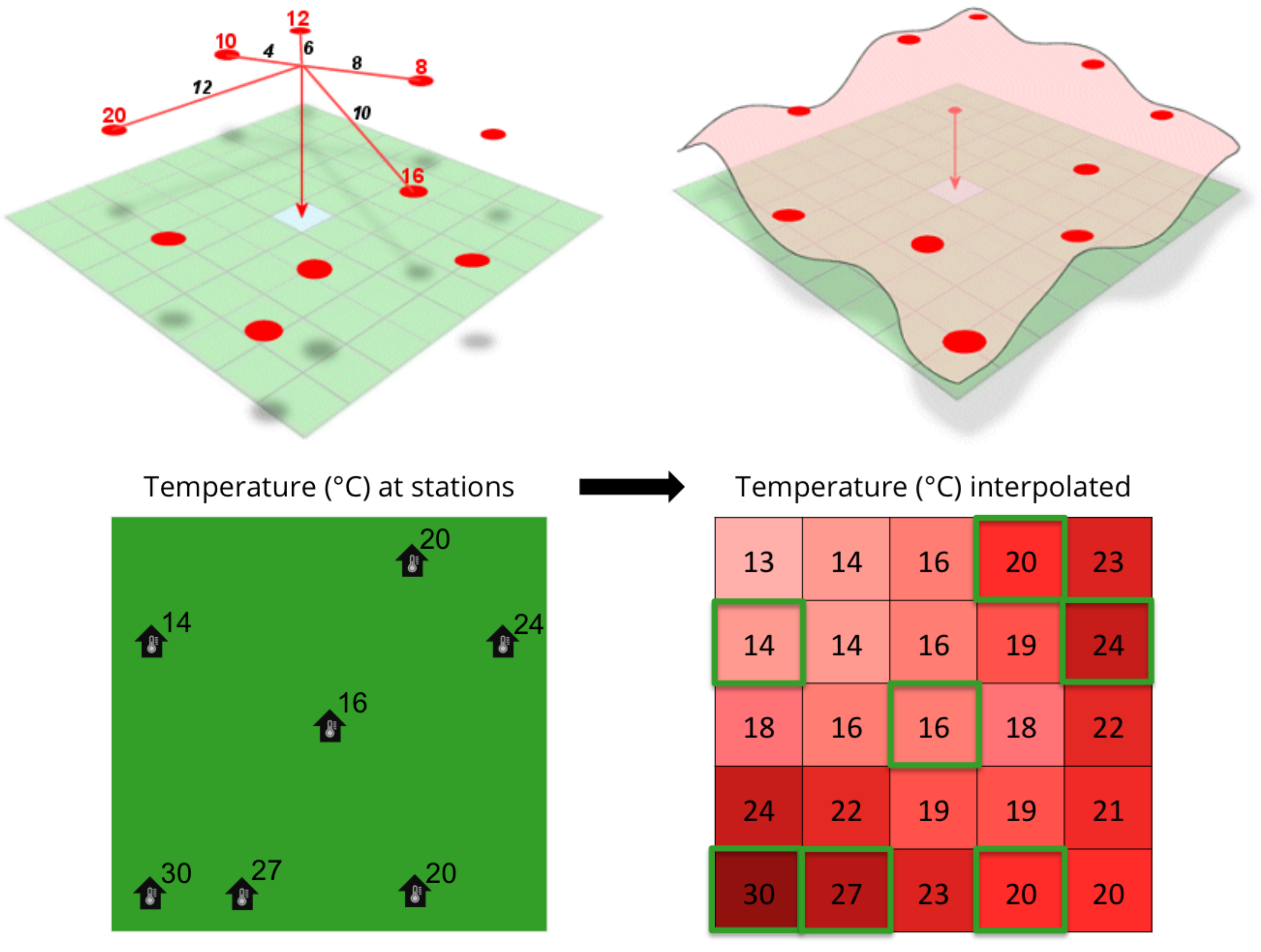
Variáveis ambientais
| Variável | Tipo | Descrição |
|---|---|---|
| BIO01 | Temperatura | Temp. média anual |
| BIO02 | Temperatura | Variação diurna média da temp. |
| BIO03 | Temperatura | Isotermalidade |
| BIO04 | Temperatura | Sazonalidade da temp. |
| BIO05 | Temperatura | Temp. máx. do mês mais quente |
| BIO06 | Temperatura | Temp. mín. do mês mais frio |
| BIO07 | Temperatura | Amplitude térmica anual |
| BIO08 | Temperatura | Temp. média do trim. mais úmido |
| BIO09 | Temperatura | Temp. média do trim. mais seco |
| BIO10 | Temperatura | Temp. média do trim. mais quente |
| BIO11 | Temperatura | Temp. média do trim. mais frio |
Bioclimáticas
- Temp. = Temperatura
- Prec. = Precipitação
- trim. = Trimestre
Variáveis ambientais
| Variável | Tipo | Descrição |
|---|---|---|
| BIO12 | Precipitação | Prec. anual |
| BIO13 | Precipitação | Prec. do mês mais chuvoso |
| BIO14 | Precipitação | Prec. do mês mais seco |
| BIO15 | Precipitação | Sazonalidade da prec. |
| BIO16 | Precipitação | Prec. do trim. mais chuvoso |
| BIO17 | Precipitação | Prec. do trim. mais seco |
| BIO18 | Precipitação | Prec. do trim. mais quente |
| BIO19 | Precipitação | Prec. do trim. mais frio |
Bioclimáticas
- Temp. = Temperatura
- Prec. = Precipitação
- trim. = Trimestre
Variáveis ambientais
Princípio da Parcimônia
- William of Ockham’s (1285–1348)
- “É fútil fazer com mais o que pode ser feito com menos”

Variáveis ambientais
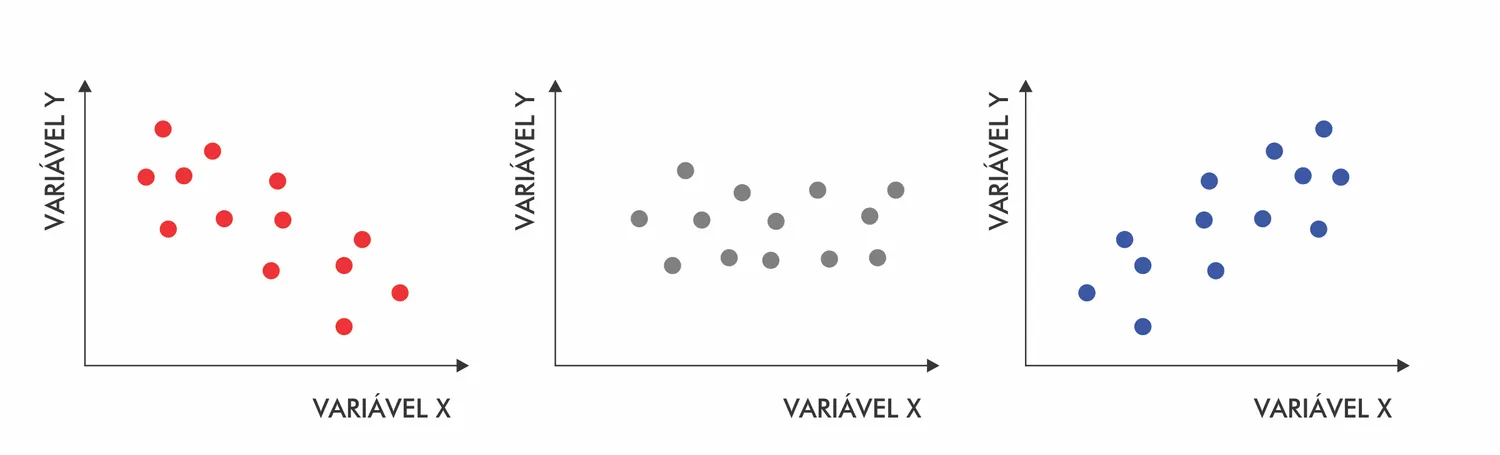
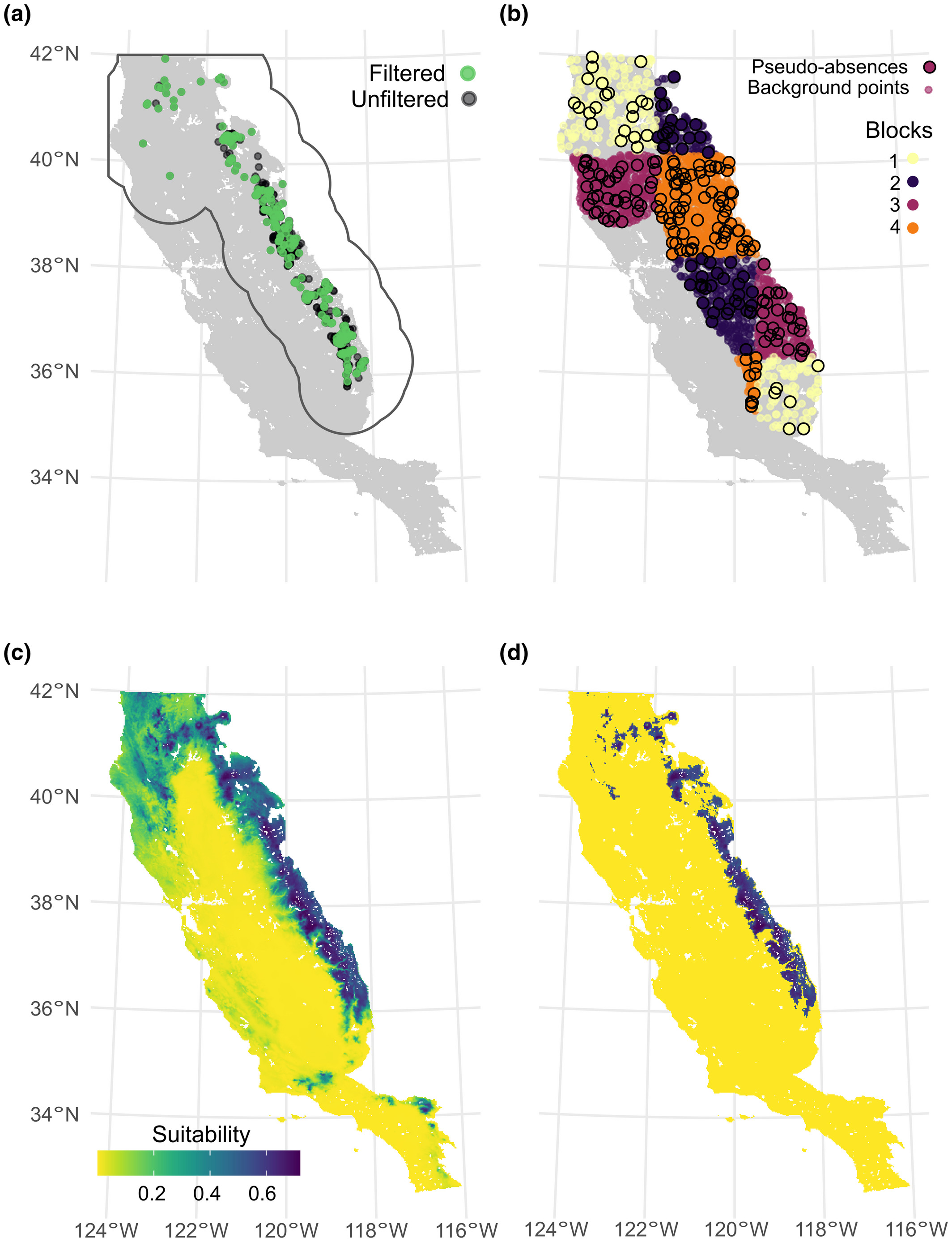
Colinearidade - Correlação
- Mede o grau de relação (positiva ou negativa) entre duas variáveis
- r > |0.7|: indica alta correlação
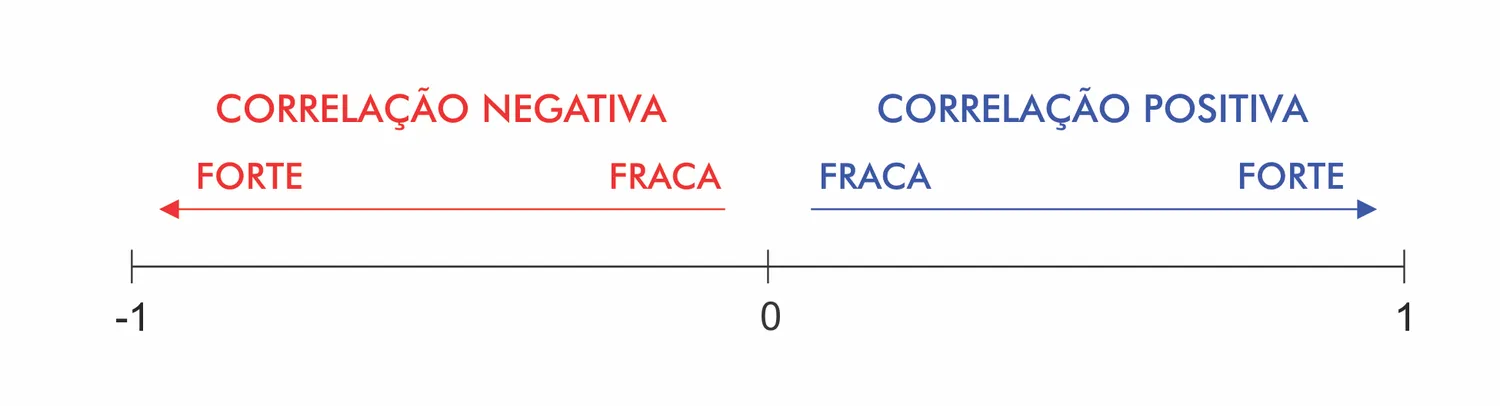
Variáveis ambientais
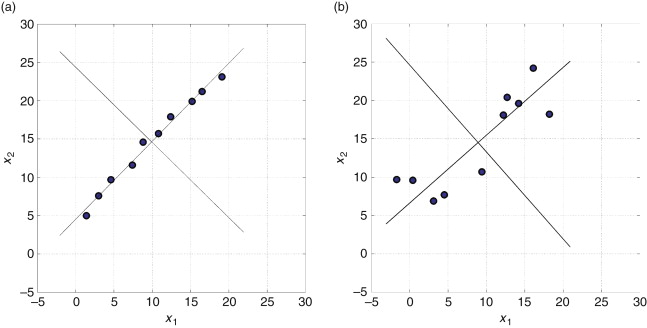
Colinearidade - Fator de Inflação de Variância (VIF)
- Medida da quantidade de multicolinearidade em um conjunto de múltiplas variáveis
- VIF = 1: não há multicolinearidade entre as variáveis
- VIF > 2 ou VIF > 5 ou VIF > 10: indica alta multicolinearidade (depende do autor)
- Na figura: a) r = 0.8946 e VIF = 5.007 e b) r = 0.7697 e VIF = 2.453
Visão geral

Visão geral
Protocolo padrão para modelos de distribuição de espécies

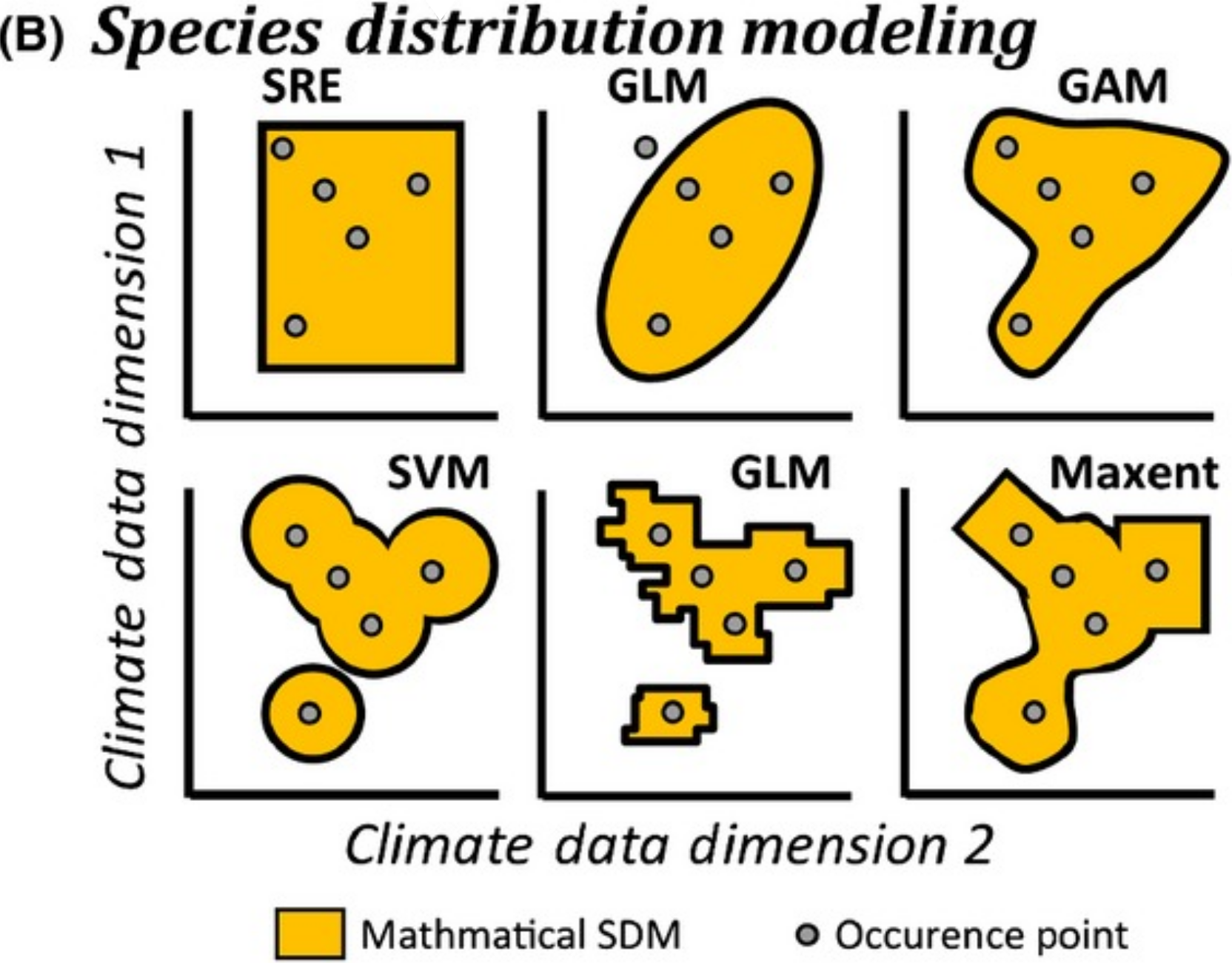
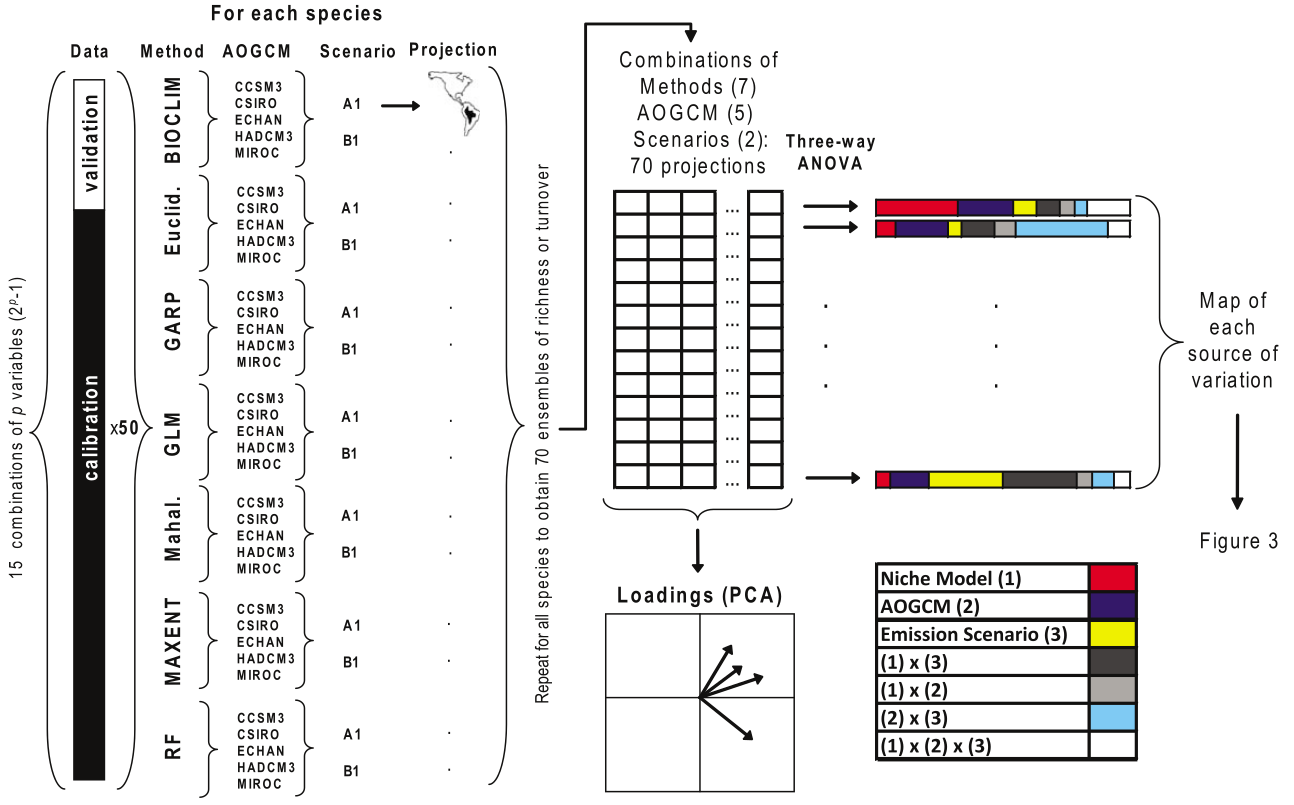
Métodos ou algoritmos
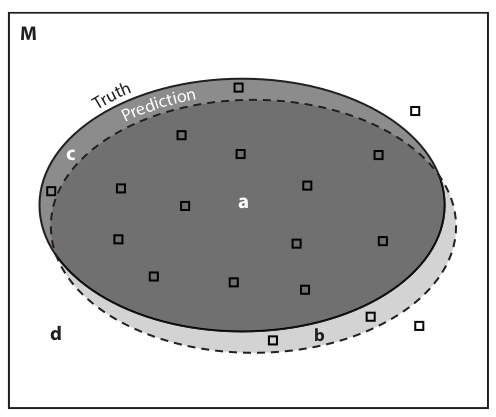
Métodos modelam diferentes áreas do Diagrama BAM
Métodos ou algoritmos
Depende dos dados de presença e ausência da espécie

Ausências em Ecologia
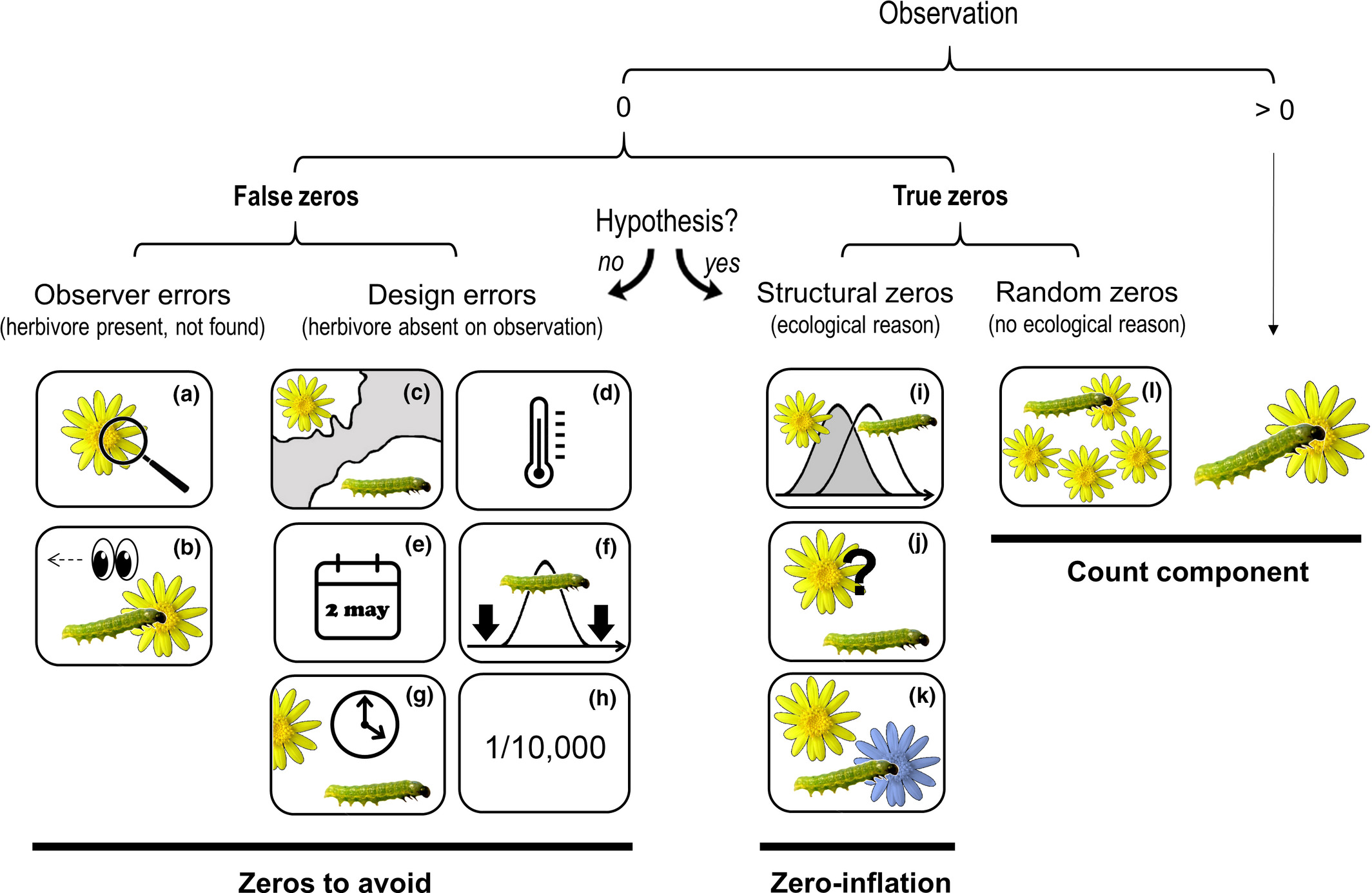
- Zeros falsos: experiência do observador (a – b) ou desenho experimental (c – h)
- Zeros verdadeiros: sistema ecológico estudado - zeros estruturais (i – k) ou variabilidade da amostragem - zeros aleatórios (l)
Métodos ou algoritmos
Depende dos dados de presença e ausência da espécie
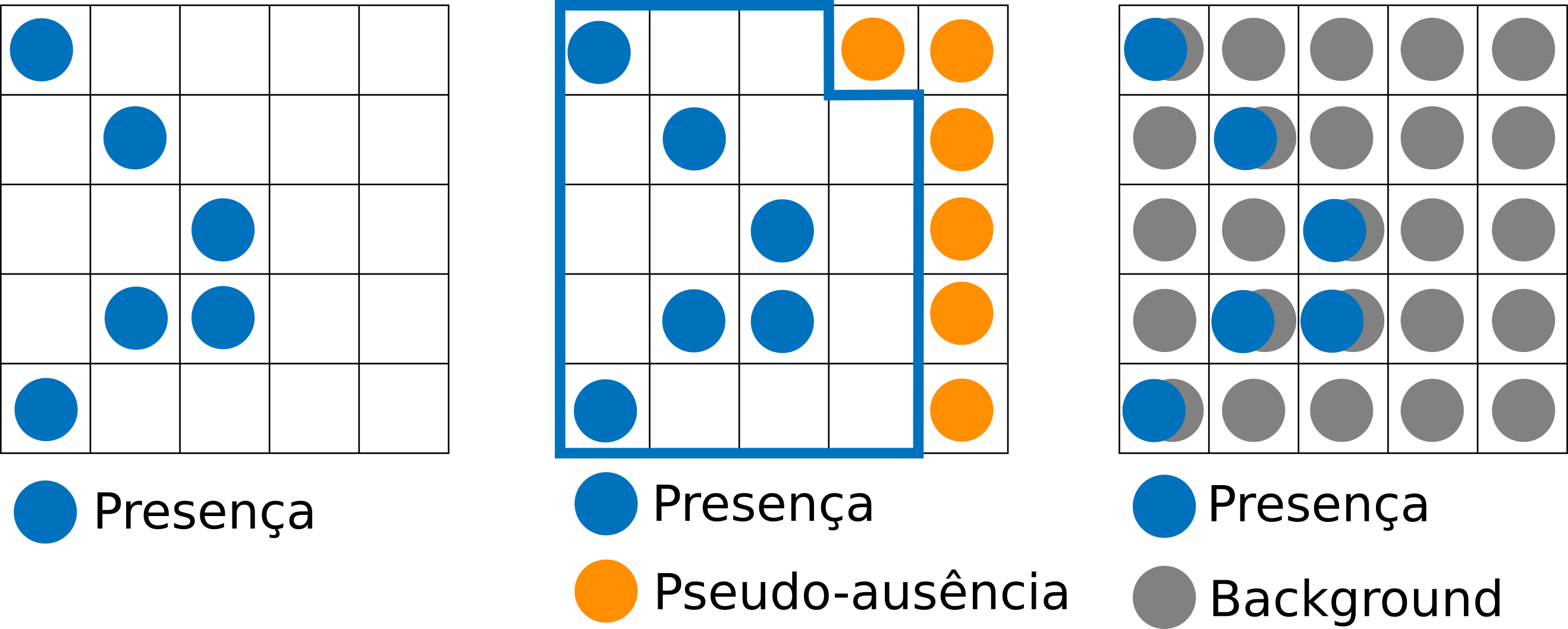
Somente presença
Bioclim (Envelope score)
Envelope climático com escores
Somente presença
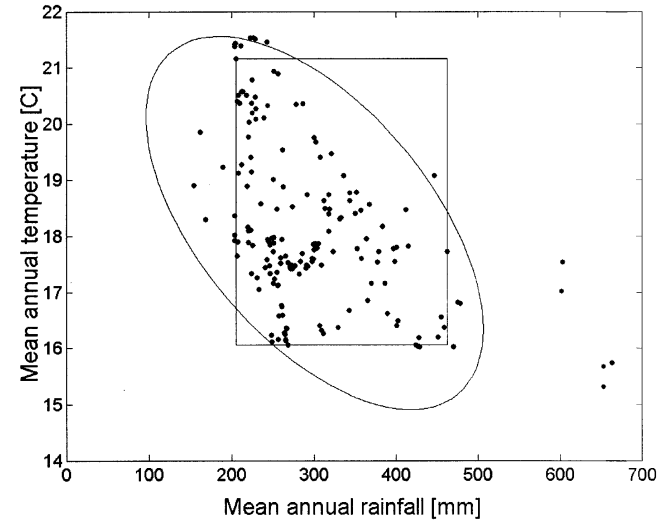
Domain (Distântica de Gower)
Distância ponto a ponto

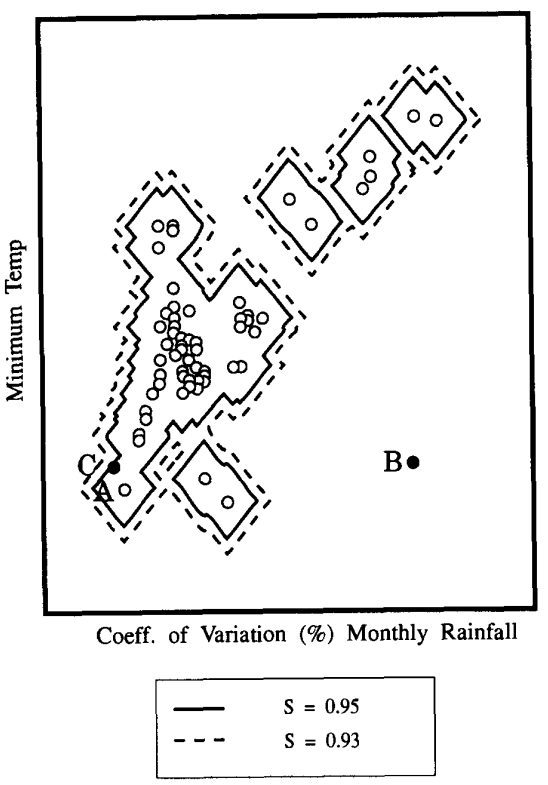
Somente presença
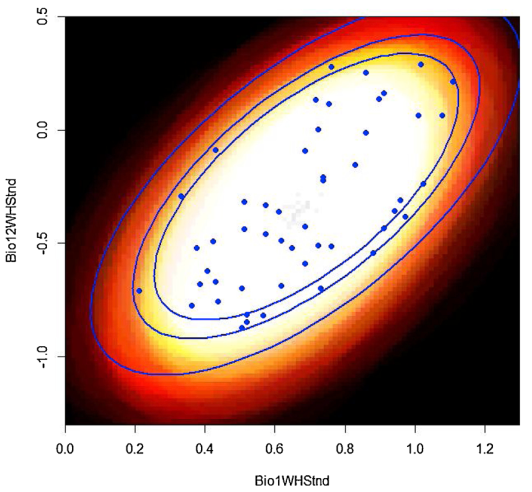
Mahalanobis (Distância de Mahalanobis)
Envelope elíptico reflete melhor a tendência central da teoria do nicho
Presença e pseudopresença
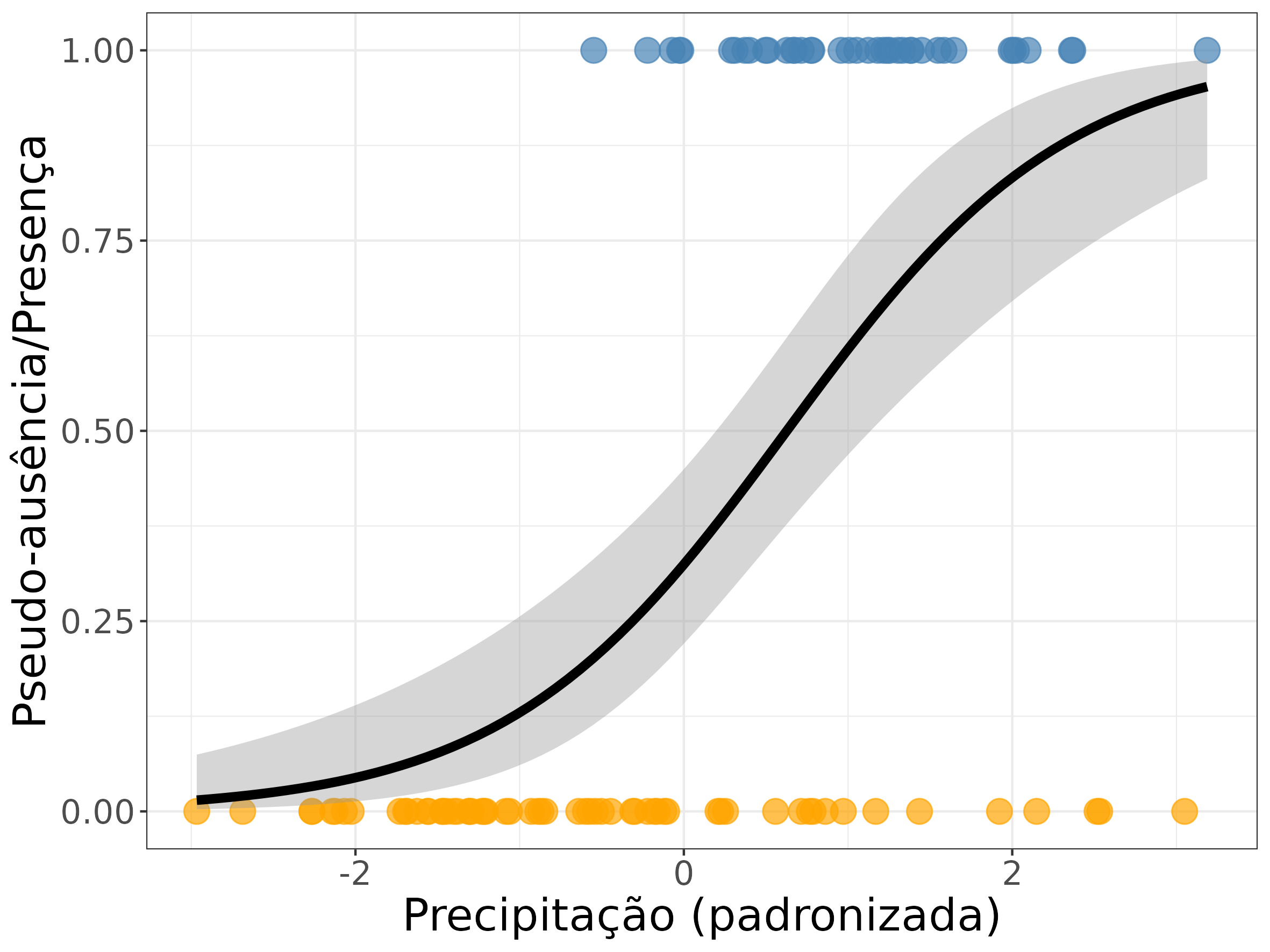
GLM (Modelos Lineares Generalizados - Generalized Linear Models)
- Distribuição de erros binomial
Presença e pseudopresença
GAM (Modelos Generalizados Aditivos - Generalized Additive Model)
- Distribuição de erros binomial com funções de suavização
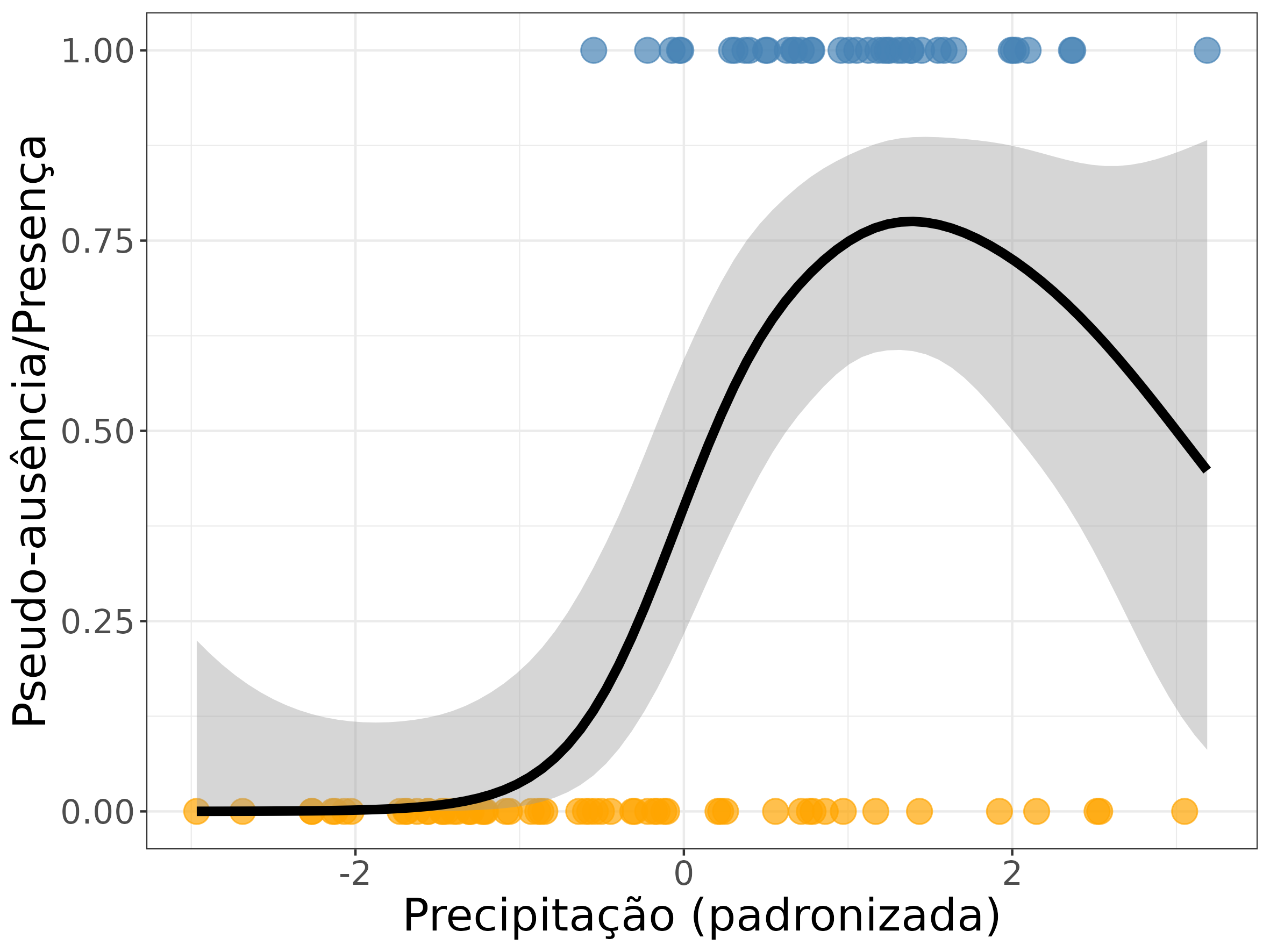
Presença e pseudopresença
Random Forest
- Cria diversas árvores de decisão (‘floresta’) por reamostragem (bootstrap)
- Combina os resultados de cada árvore em uma última classificação (categórico) ou regressão (contínuo)
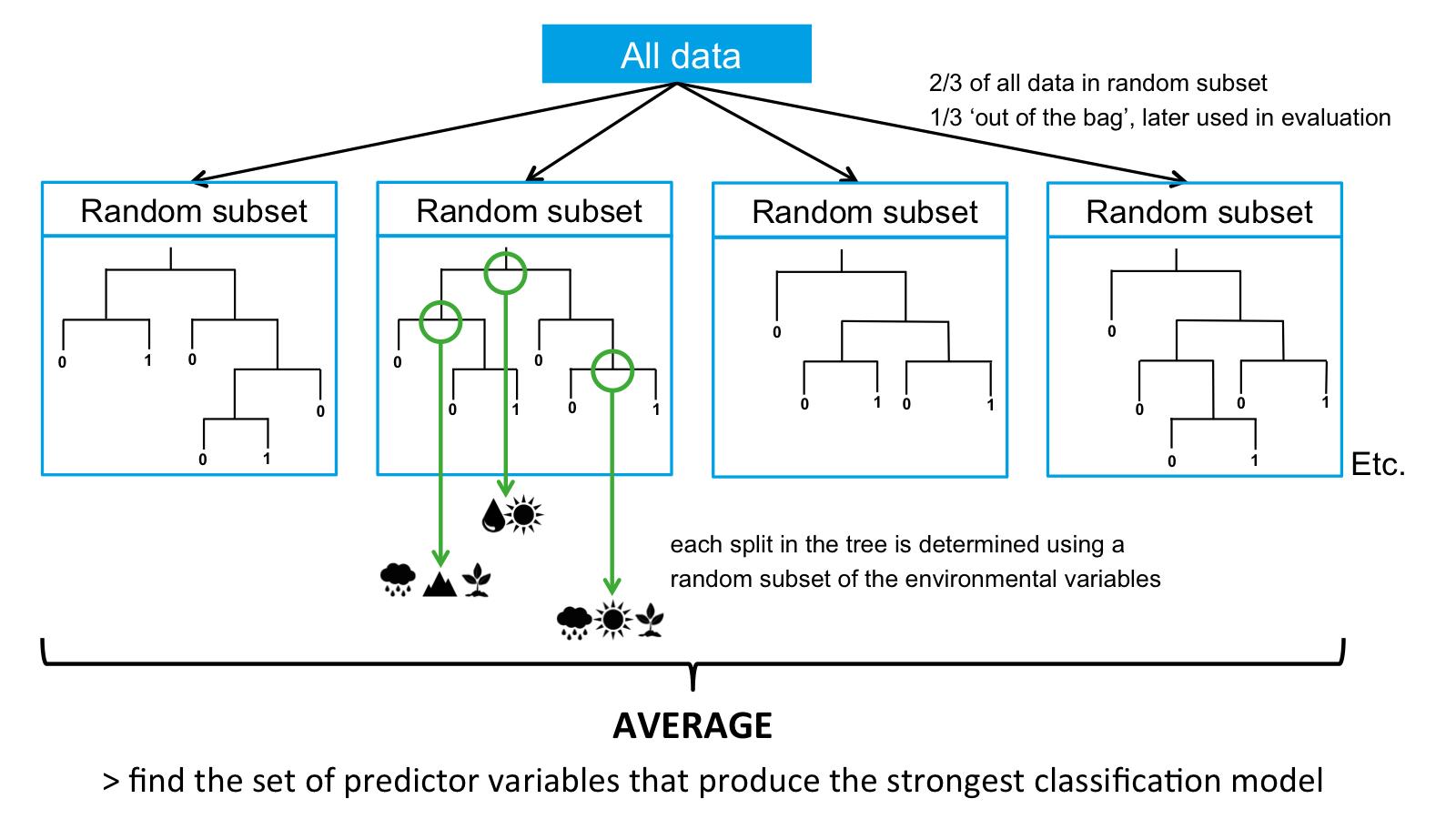
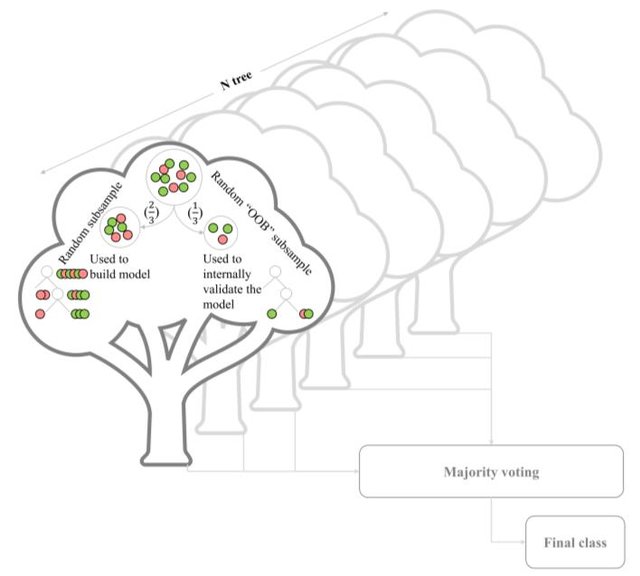
Presença e pseudopresença
SVM (Support Vector Machine)
- Cria hiperplanos através de vetores de suporte
- Faz a classificação (categórico) ou regressão (contínuo) para separar os dados em duas ou mais partes
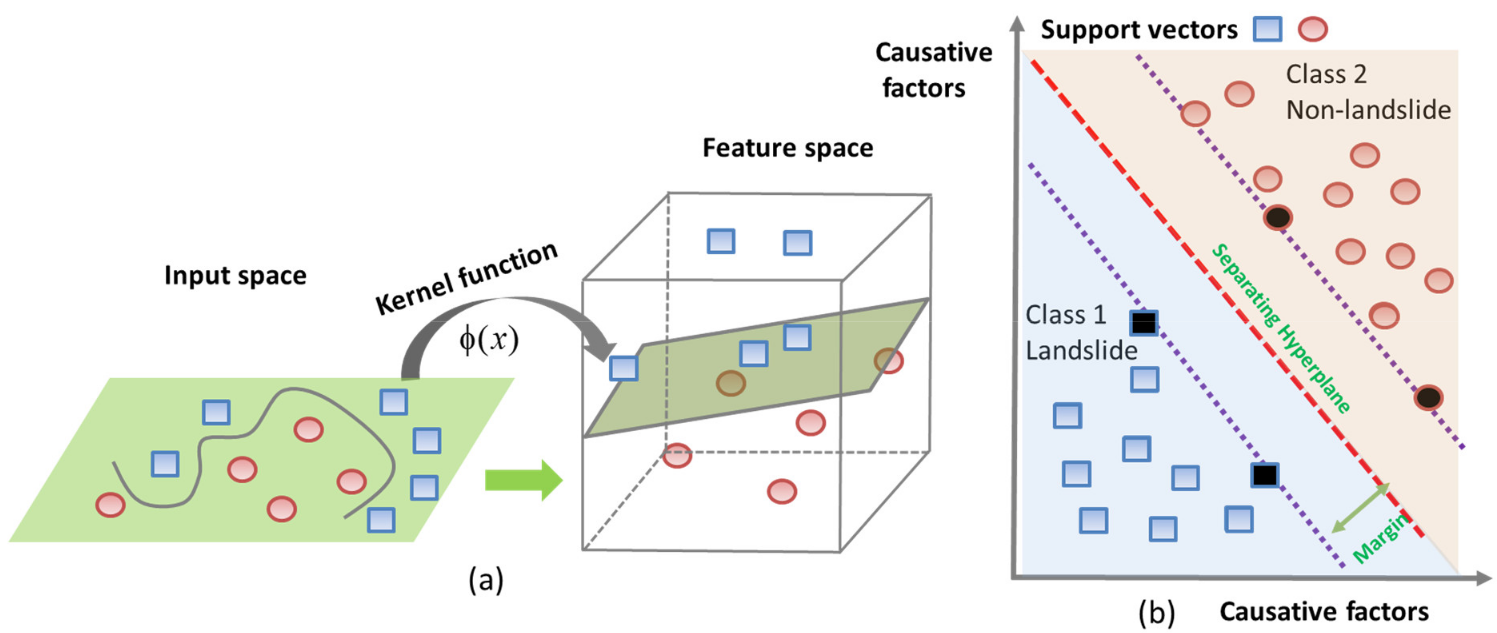
Presença e background
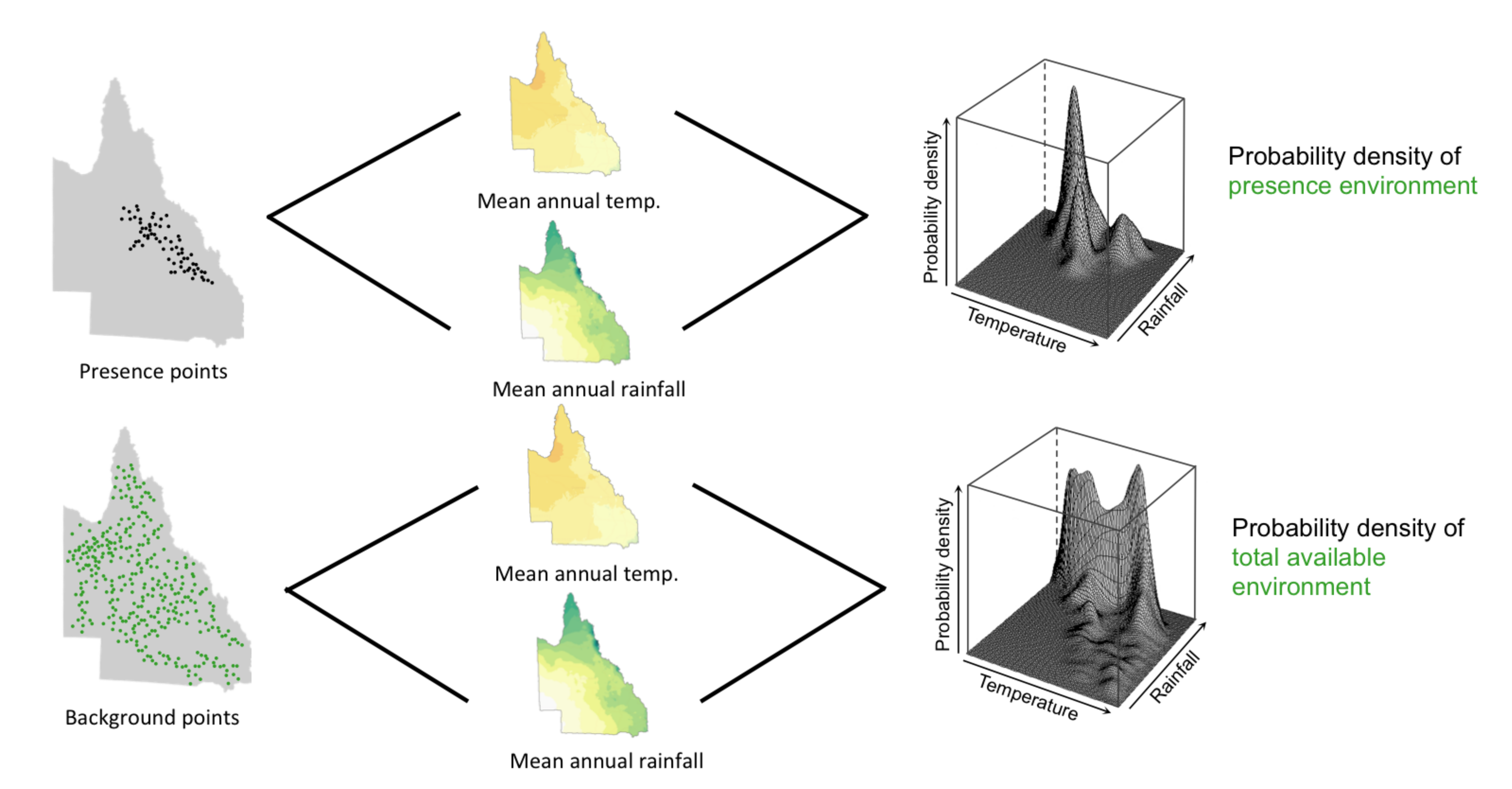
MaxEnt (Máxima Entropia)
- Compreender as características gerais de um sistema de informação parcial e incompleto
- Aproxima o formato da distribuição dos valores do background aos valores das presenças, usando diferentes curvas (features) para achar os valores de adequabilidade
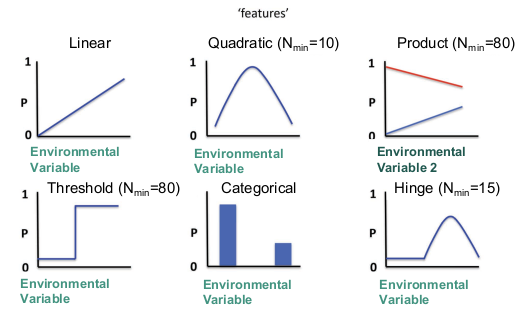
Outros métodos ou algoritmos
Presença-pseudoausência
- Classification and regression trees (CART)
- Multivariate adaptive regression spline (MARS)
- Mixture discriminant analysis (MAD)
- Artificial neural networks (ANN)
- Boosted Regression Trees (BRT)
- Bayesian Gaussian Process (GAU)
Presença-background
- Ecological Niche Factor Analysis (ENFA)
- Genetic Algorithm for Rule-set Production (GARP)
- Maxlike (MLK)
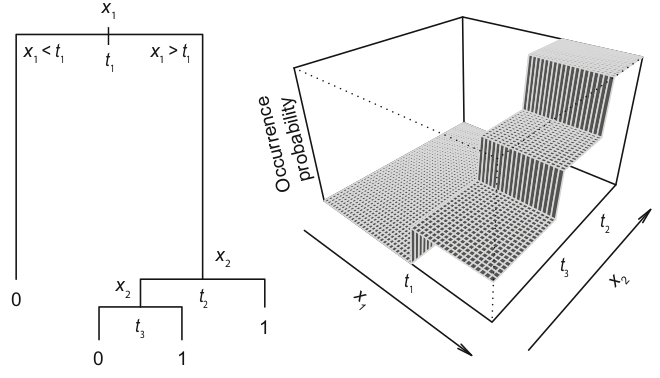
Métodos ou algoritmos
Tunando hiperparâmetros (Hyperparameter tuning)
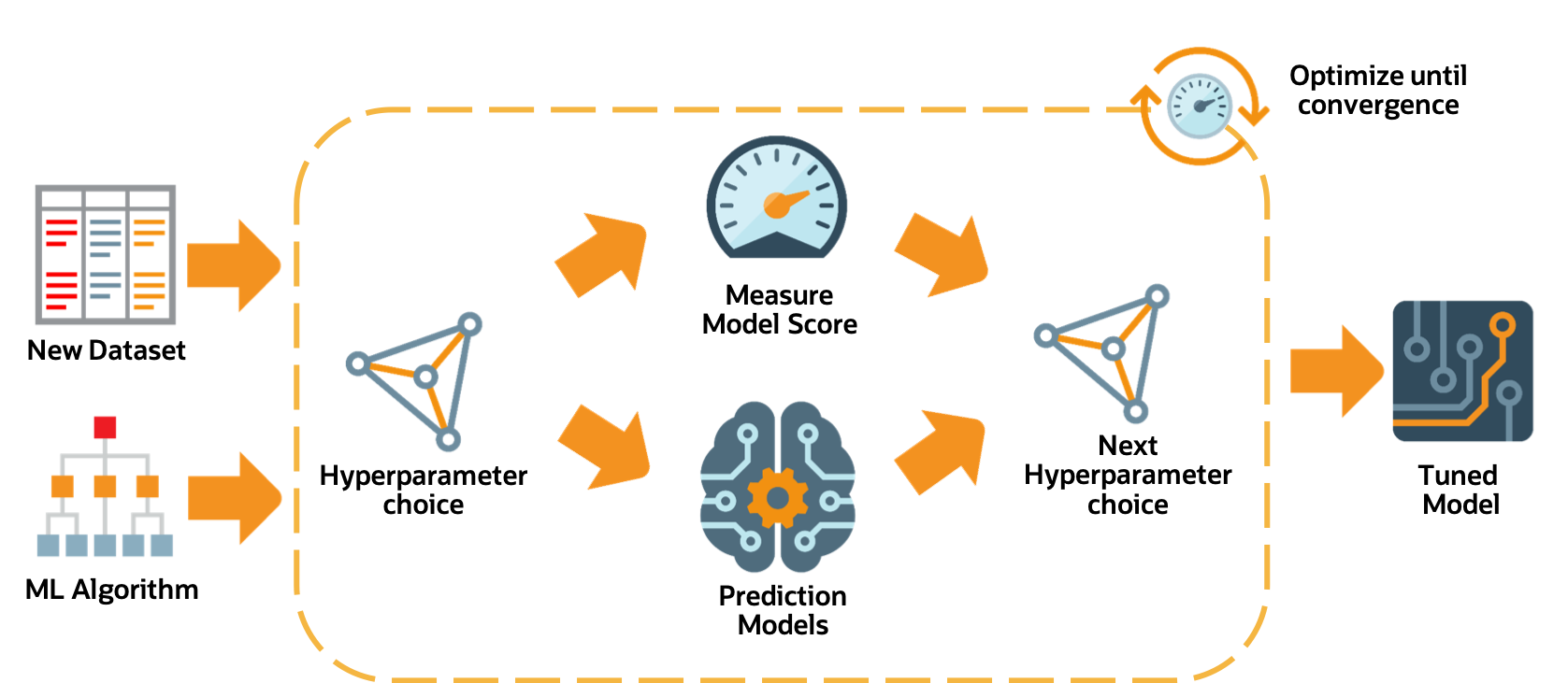
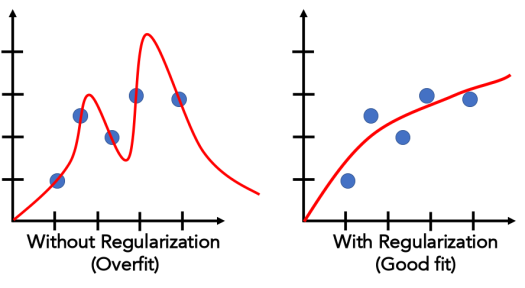
Métodos ou algoritmos
Tunando hiperparâmetros (Hyperparameter tuning)
Métodos ou algoritmos
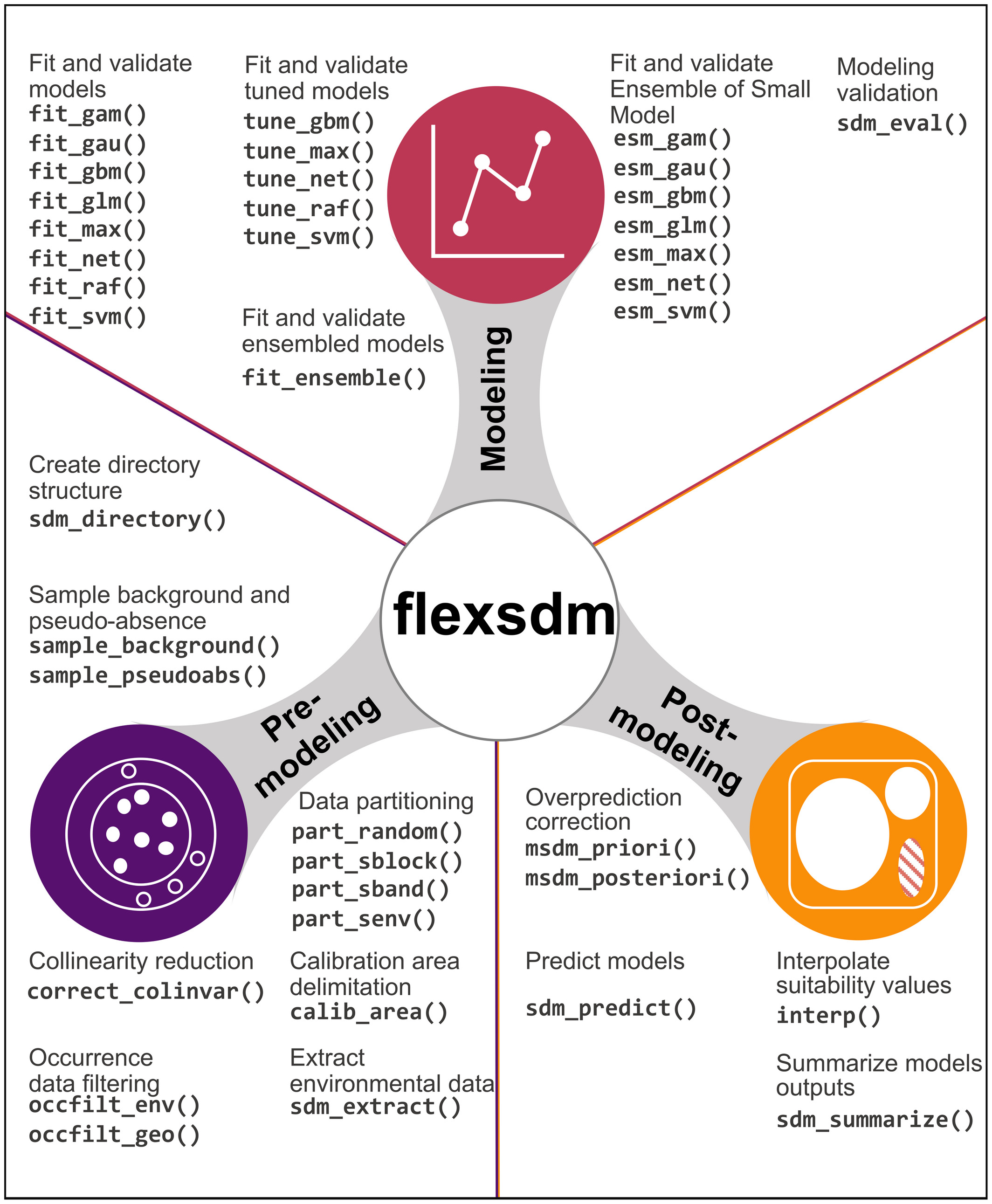
Pacotes no R

Visão geral

Visão geral
Protocolo padrão para modelos de distribuição de espécies

4. Avaliação (do ajuste) dos modelos
Avaliação
Partição dos dados (treino e teste)
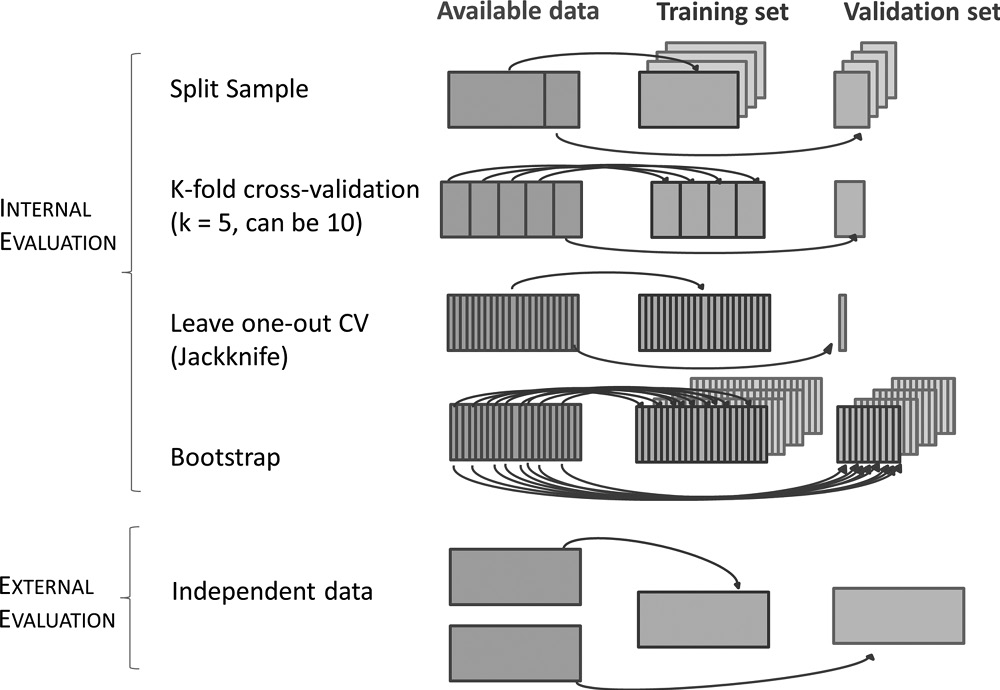
Avaliação
O modelo consegue prever nossos dados?
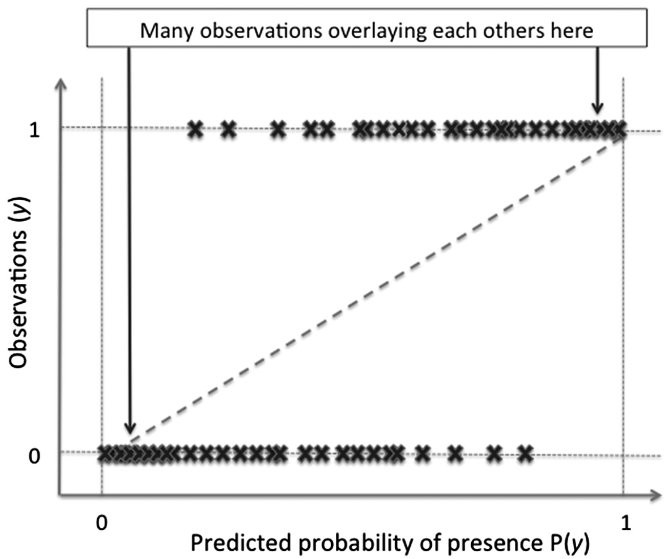
Avaliação
O modelo consegue prever nossos dados?
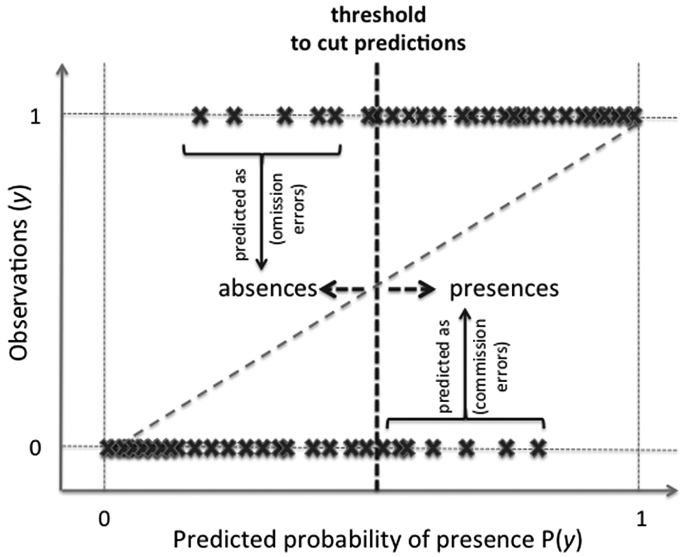
Avaliação
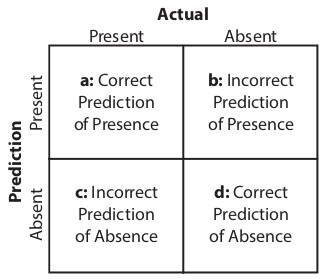
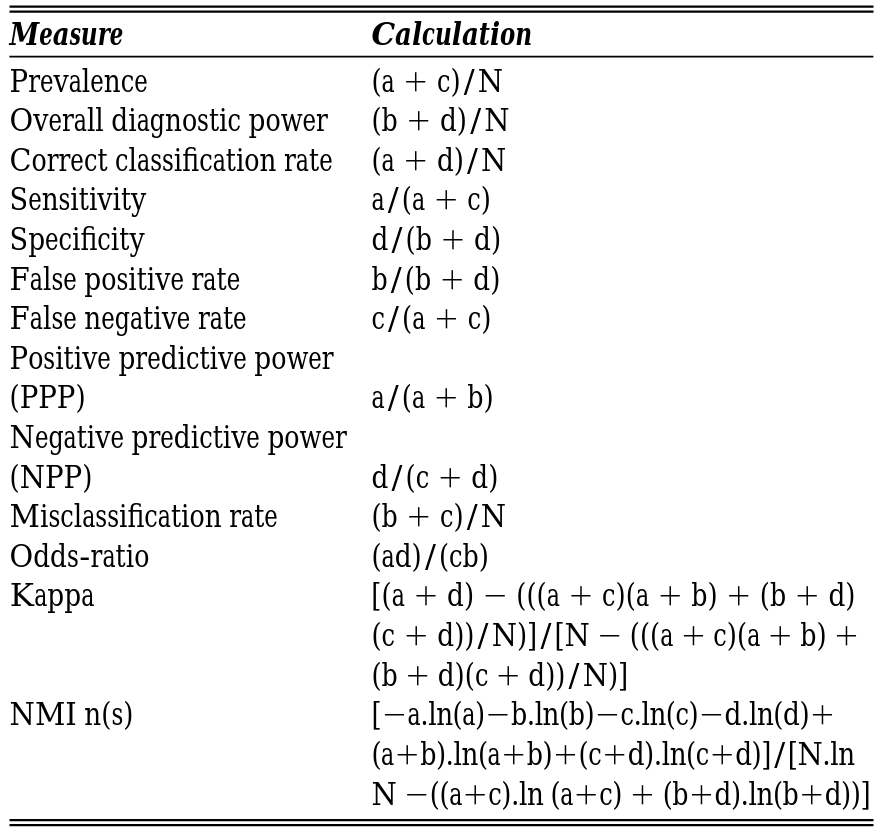
Métricas de avaliação
- Sensibilidade: proporção de presenças verdadeiras [0:1]
- Especificidade: proporção de ausências verdadeiras [0:1]
- AUC: probabilidade de classificação correta [0:1]
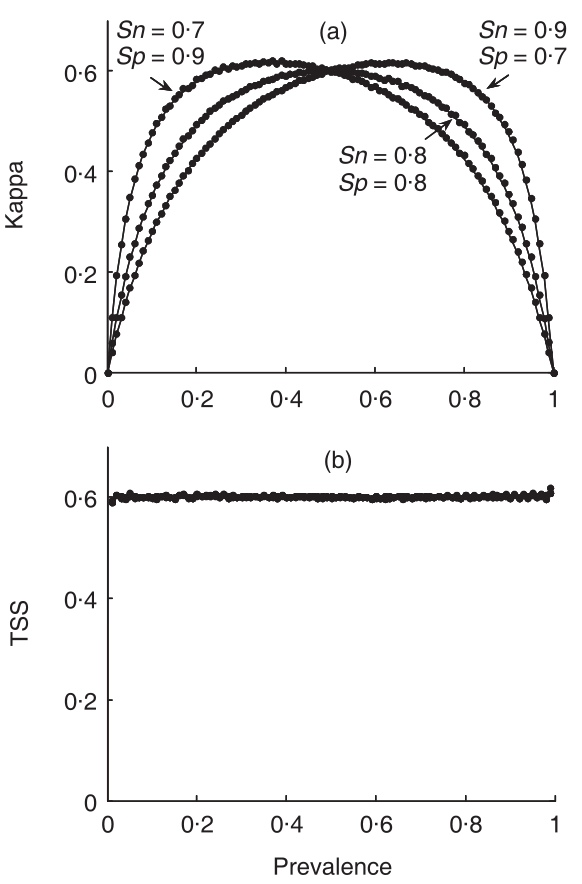
- Kappa: medida de concordância da classificação [-1:1]
- TSS: compara o número de classificações corretas, menos aquelas atribuídas aleatoriamente [-1:1]
Avaliação
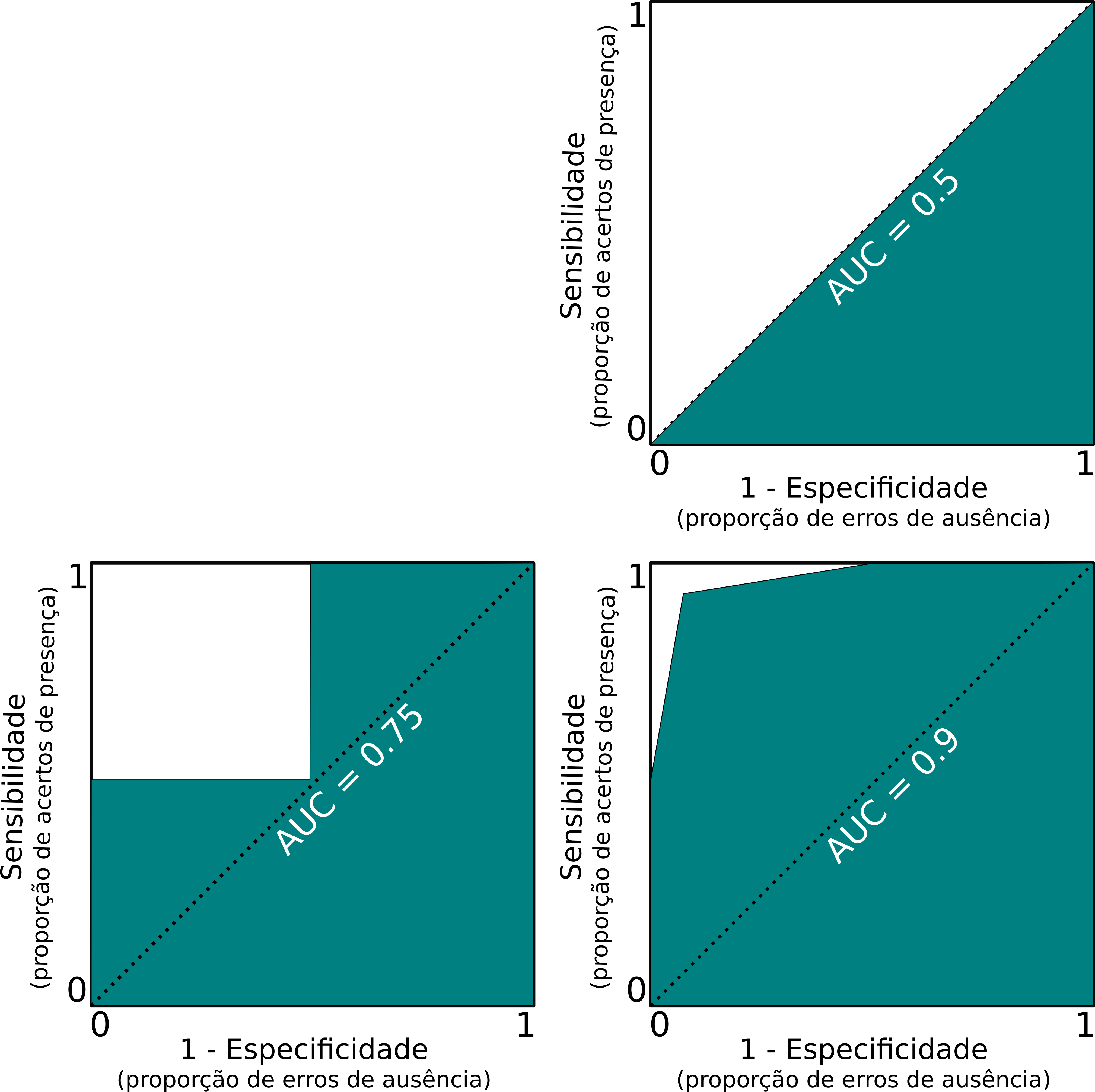
AUC (Área Abaixo da Curva - Area Under the Curve)
Avaliação
- Kappa (K) [-1 a +1]
\(K = \frac{\frac{a+d}{N}-\frac{(a+b)(a+c)+(c+d)(b+d)}{N^2}}{1-\frac{(a+b)(a+c)+(c+d)(d+b)}{N^2}} = \frac{P_o - P_e}{1 - P_e}\), onde P = acurácia
K > 0.75: Excelente
0.40 > K > 0.75: Bom
K < 0.40: pobre
- TSS (True Skill Statistic) [-1 a +1]
\(TSS = \frac{ad-bc}{(a+c)(b+d)} = Sensitividade + Especificidade - 1\)
TSS ≅ +1: concordância perfeita
TSS <= 0: desempenho não melhor do que aleatório
Indicativos: TSS > 0.5, TSS > 0.7, TSS > 0.8 e TSS > 0.9
Visão geral

Visão geral
Protocolo padrão para modelos de distribuição de espécies

5. Predição no espaço e no tempo
Predição
Estimar a distribuição para onde não temos dados
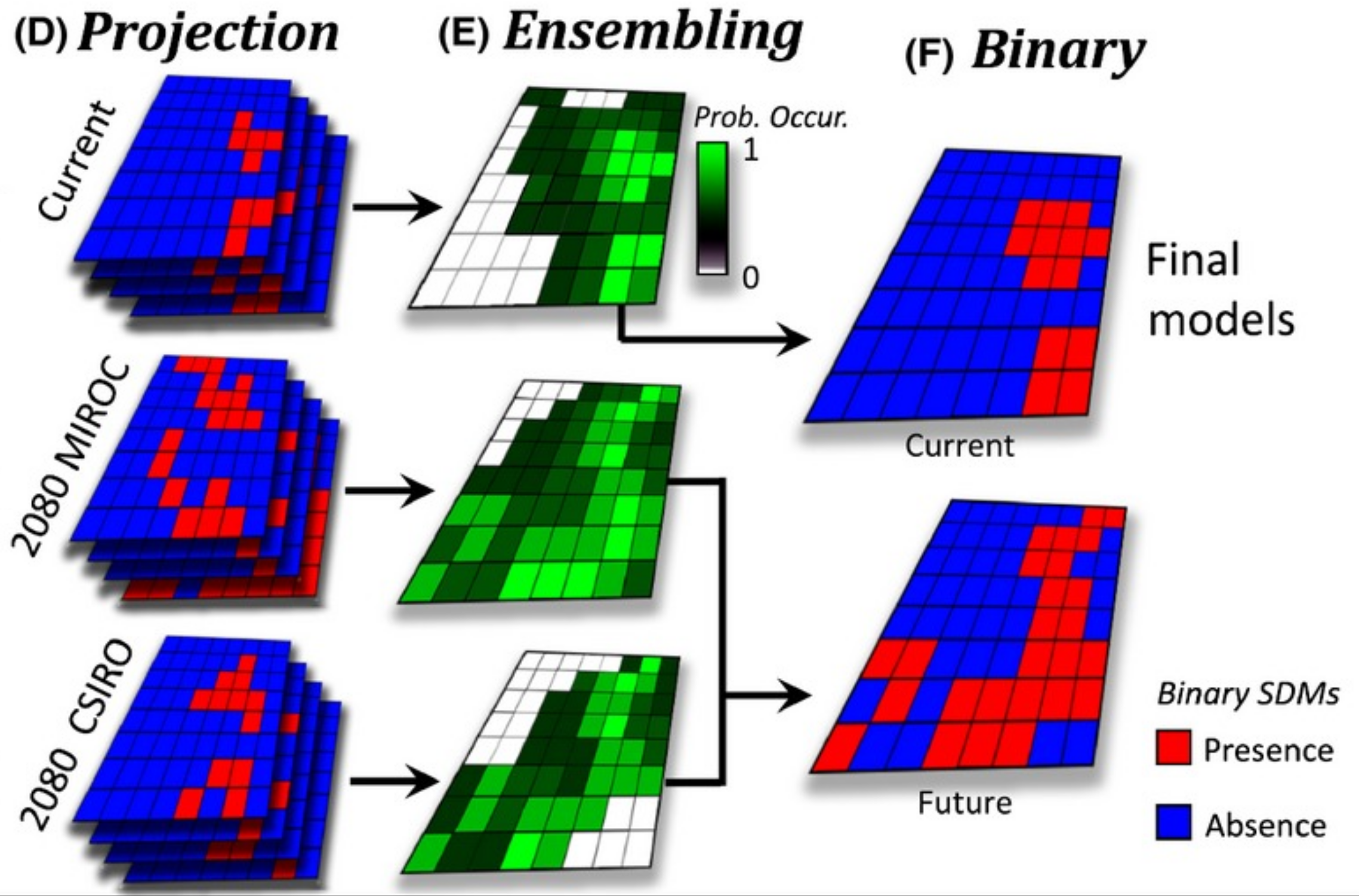
Predição
Diferentes estimativas do nicho geram diferentes predições geográficas
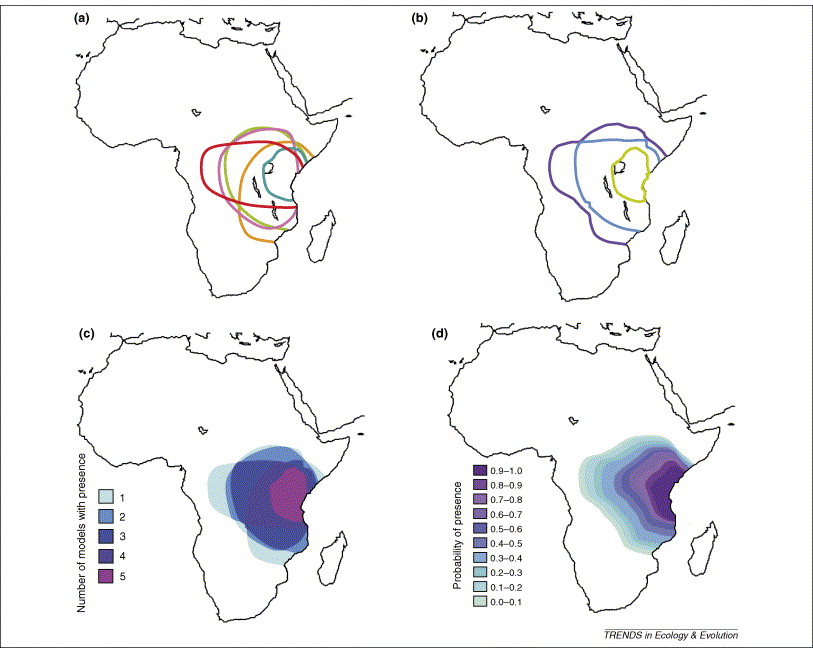
Predição
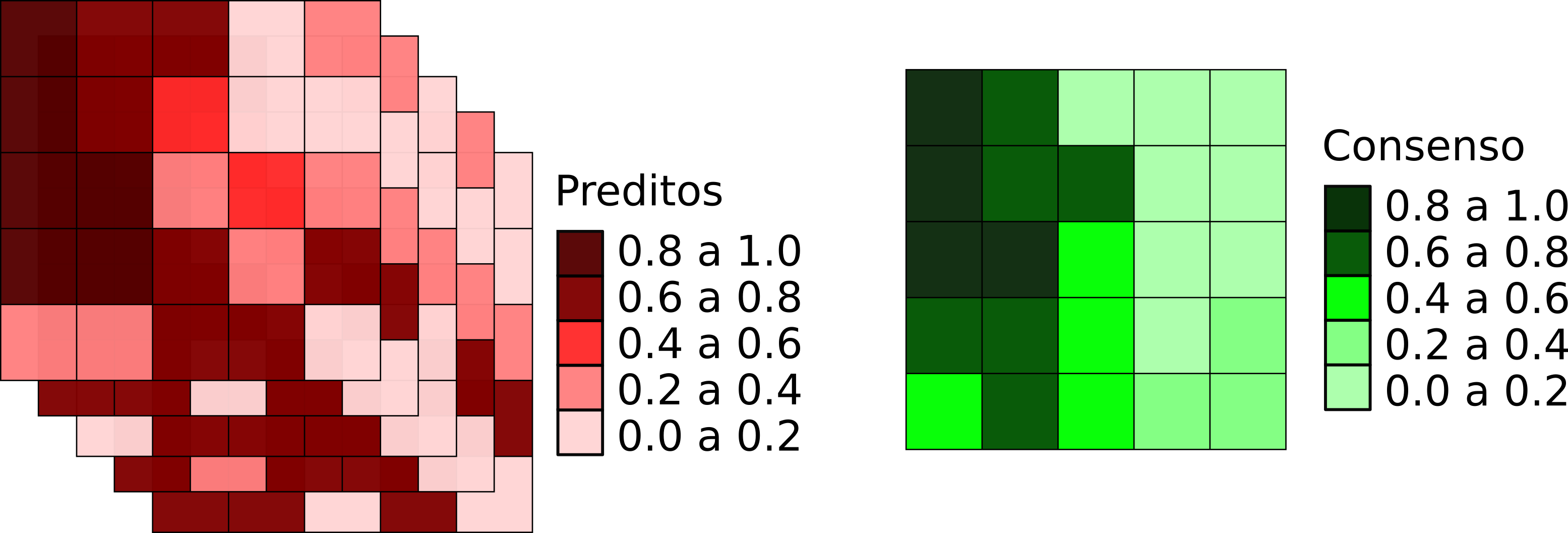
Consenso (ensemble)
- Frequência
- Média
- Média ponderada (AUC ou TSS)
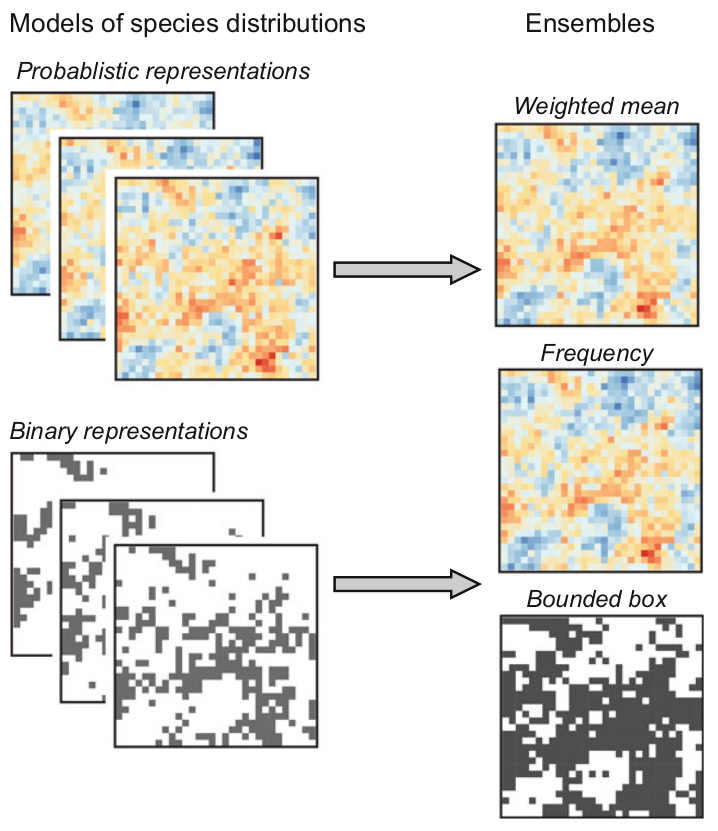
Consenso (ensemble)
Frequência: contabilizar pixels previstos como presença
- Uso de um limiar de corte para binarizar os modelos [0 e 1]
- Soma da quantidade de pixels e divisão pelo total de modelos [0 a 1]
\[ consenso = \sum_{i=1}^{N} (pixel_i \geq limiar_j) \]

Consenso (ensemble)
Média: calcular a média dos valores de adequabilidade para cada pixel
- Padronização dos valores de adequabilidade [0 a 1]
- Cálculo da média aritmética de cada pixel
\[ consenso = \frac{1}{N}\sum_{i=1}^{N} (pixel_i) \]
Consenso (ensemble)
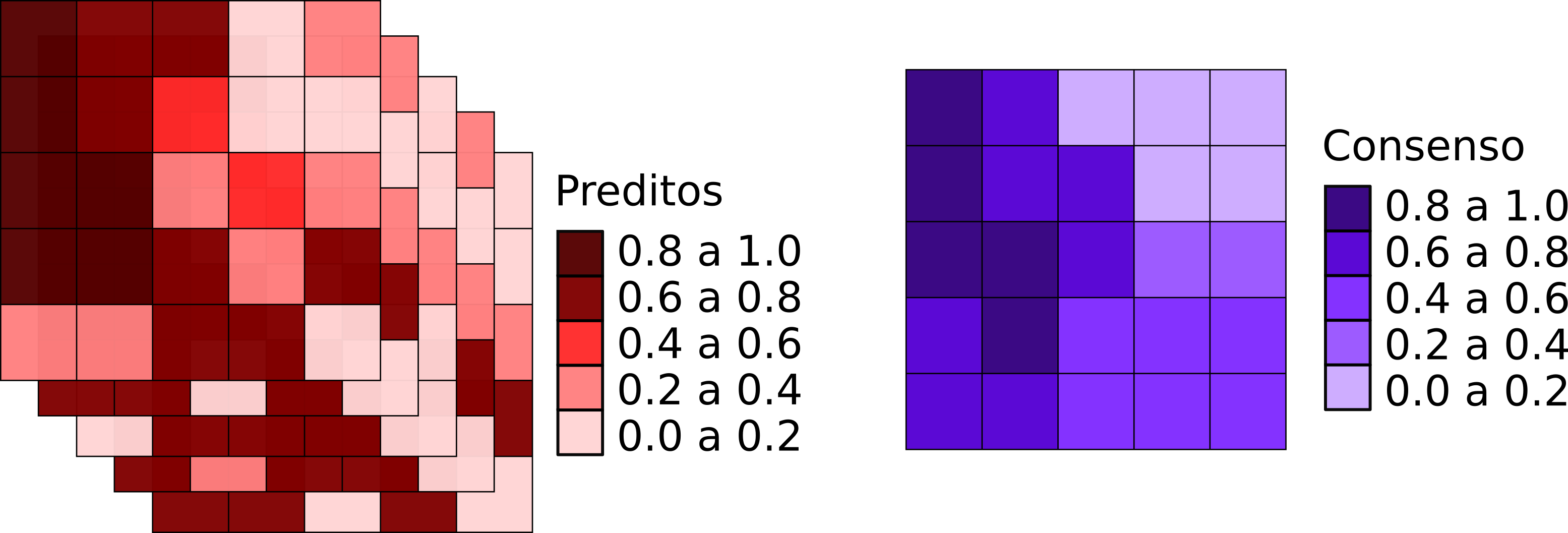
Média ponderada: calcular a média ponderada dos valores de adeq.
- Padronização dos valores de adequabilidade [0 a 1]
- Cálculo da média ponderada pelos valores de AUC ou TSS de cada pixel
\[consenso = \frac{\sum_{i=1}^{N}(w_i . pixel_i)}{\sum_{i=1}^N(w_i)}\]
Consenso (ensemble)
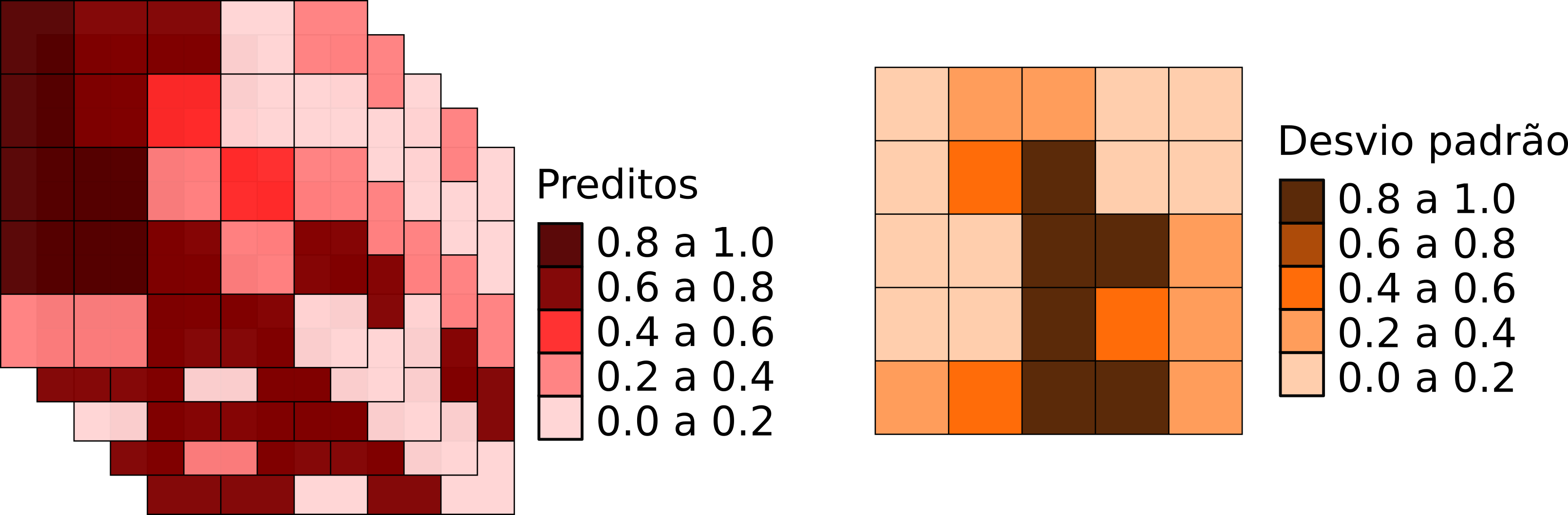
Incertezas: calcular um valor de incerteza para cada pixel
- Padronização dos valores de adequabilidade [0 a 1]
- Cálculo do desvio padrão ou partição da variância de cada pixel
\[ desvio = \sqrt{\frac{1}{N}\sum_{i=1}^{N} (pixel_i - \overline{pixel_i})^2} \]
Consenso (ensemble)
Incertezas: calcular um valor de incerteza para cada pixel
- Padronização dos valores de adequabilidade [0 a 1]
- Cálculo do desvio padrão ou partição da variância de cada pixel
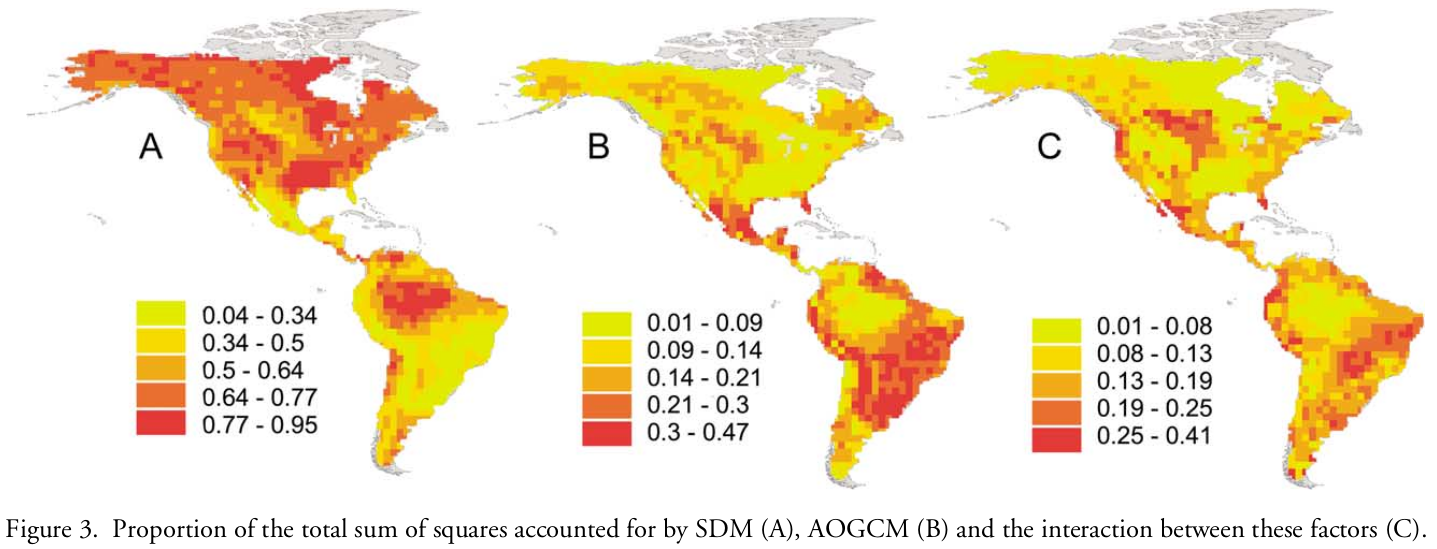
Aplicações
Conservação
Conservação
Impacto da mineração sobre anuros e aves na Serra do Espinhaço
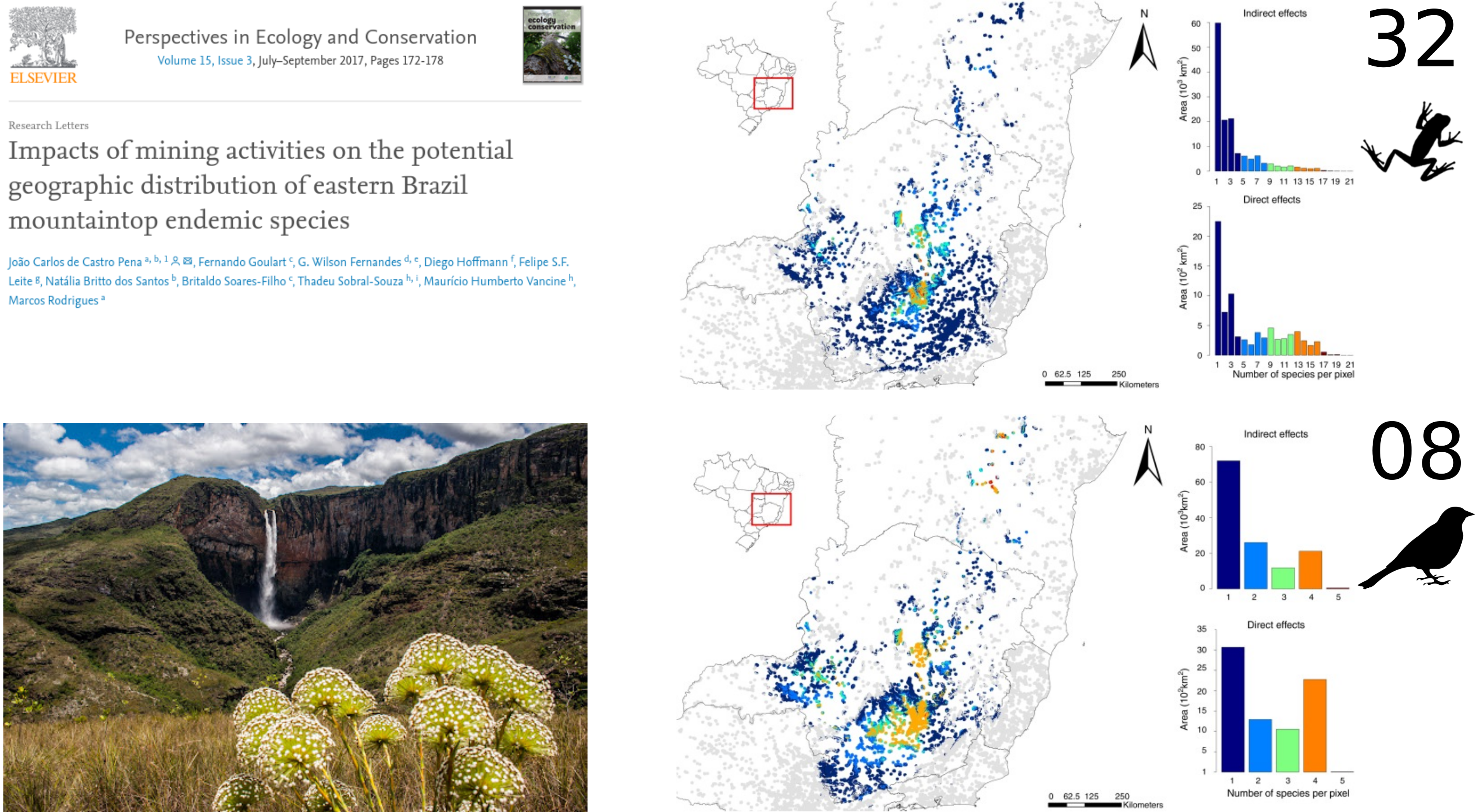
Conservação
Eficiência das áreas protegidas da Amazônia e Mata Atlântica
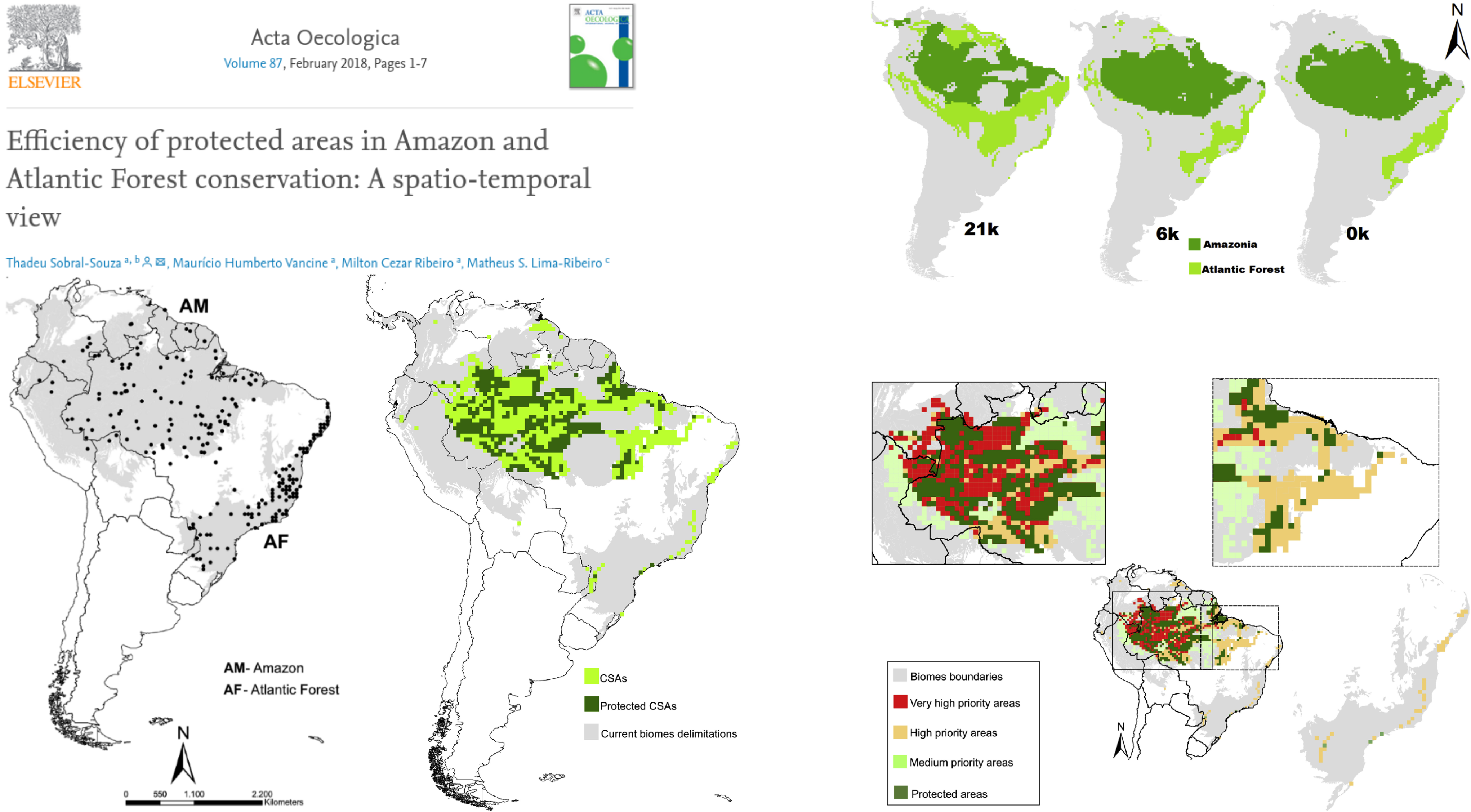
Relações ecológicas
Relações ecológicas
Distribuição de macroalgas epífitas e hospedeiras em Cuba
Ecologia da Paisagem
Ecologia da Paisagem
Clima, paisagem e riqueza de borboletas na Mata Atlântica
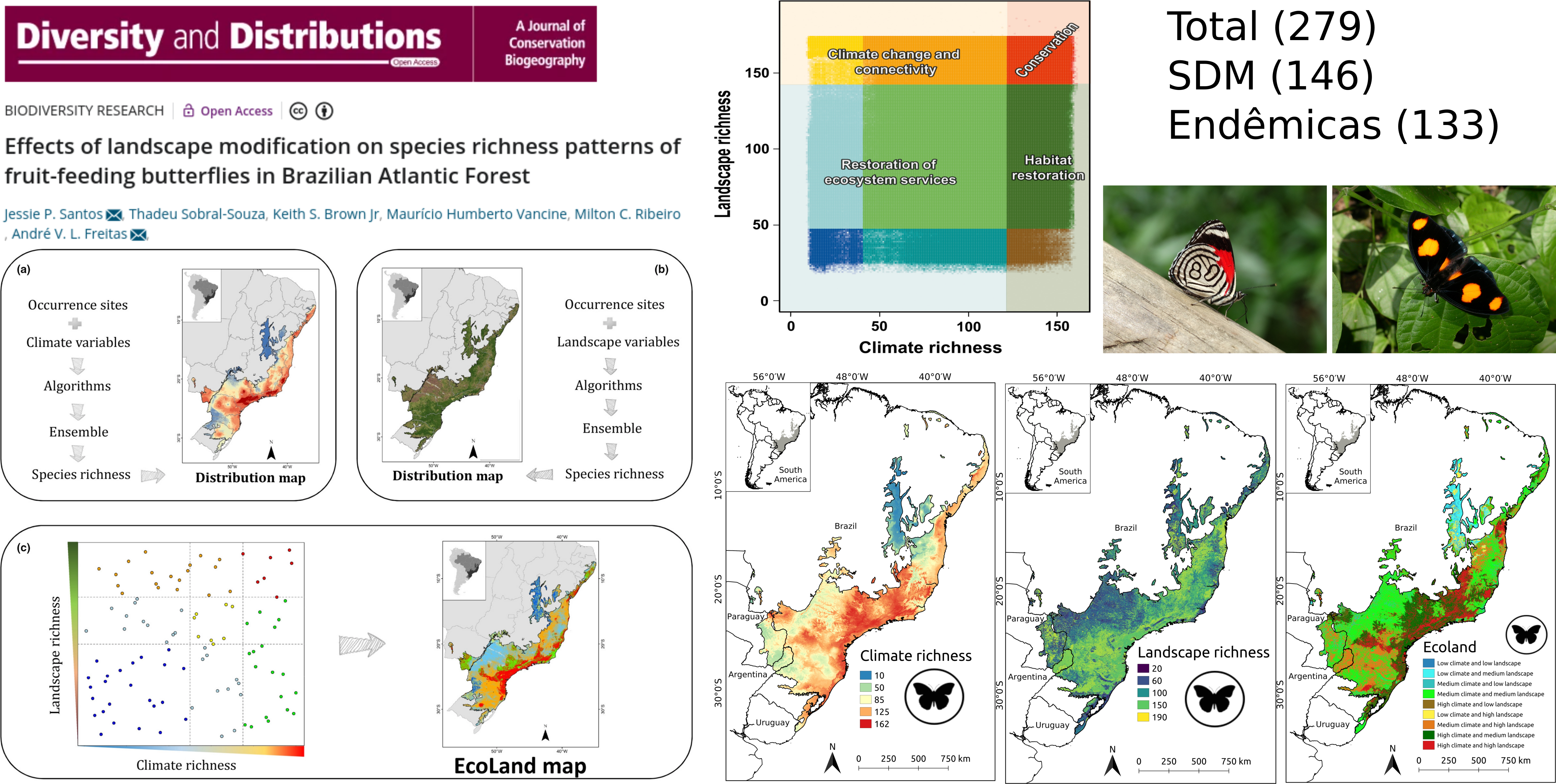
Ecologia da Paisagem
Regiões prioritárias para conservação das queixadas no Brasil
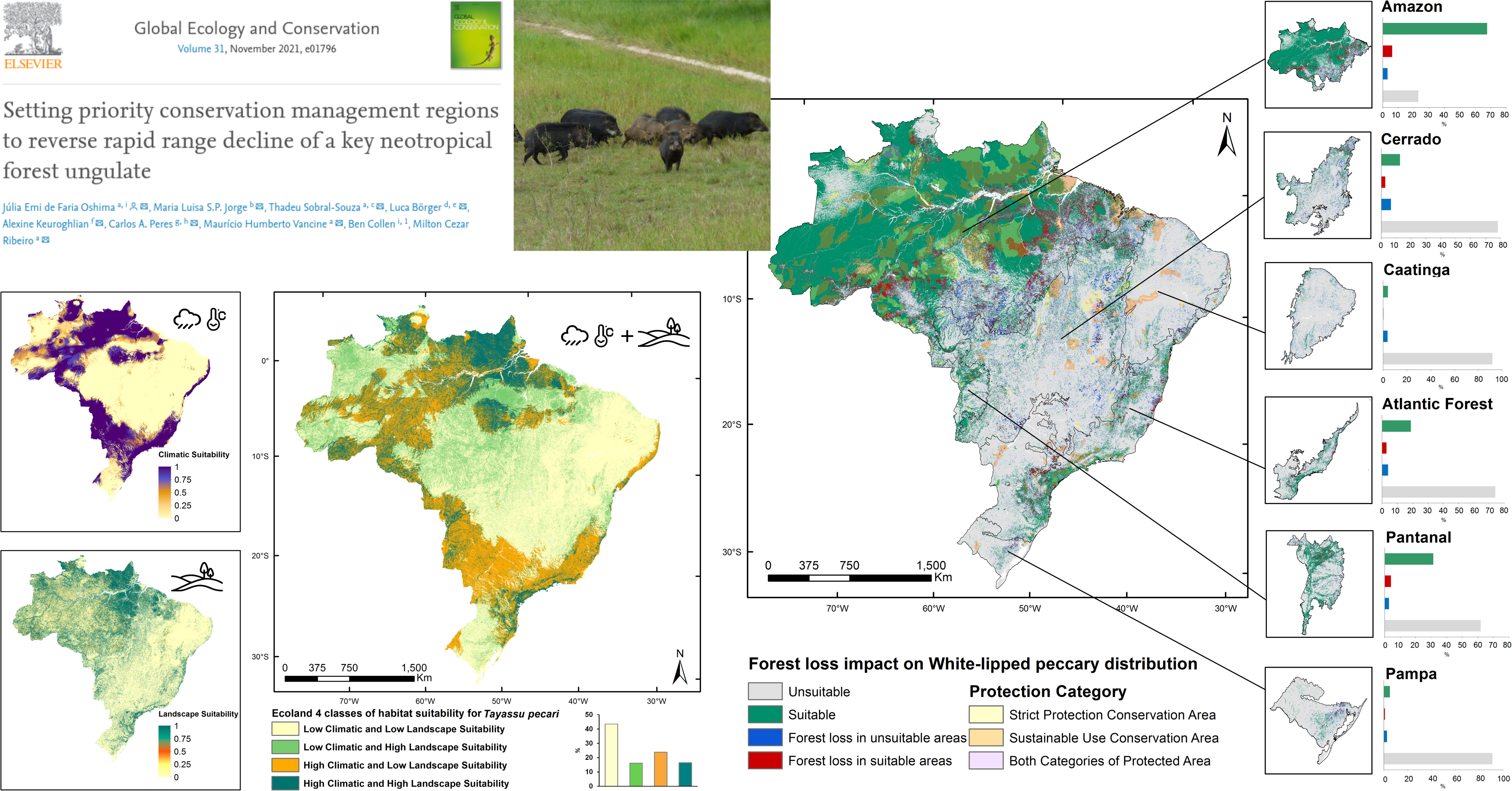
Ecologia da Paisagem
Expansão da cana sobre o tamanduá em São Paulo
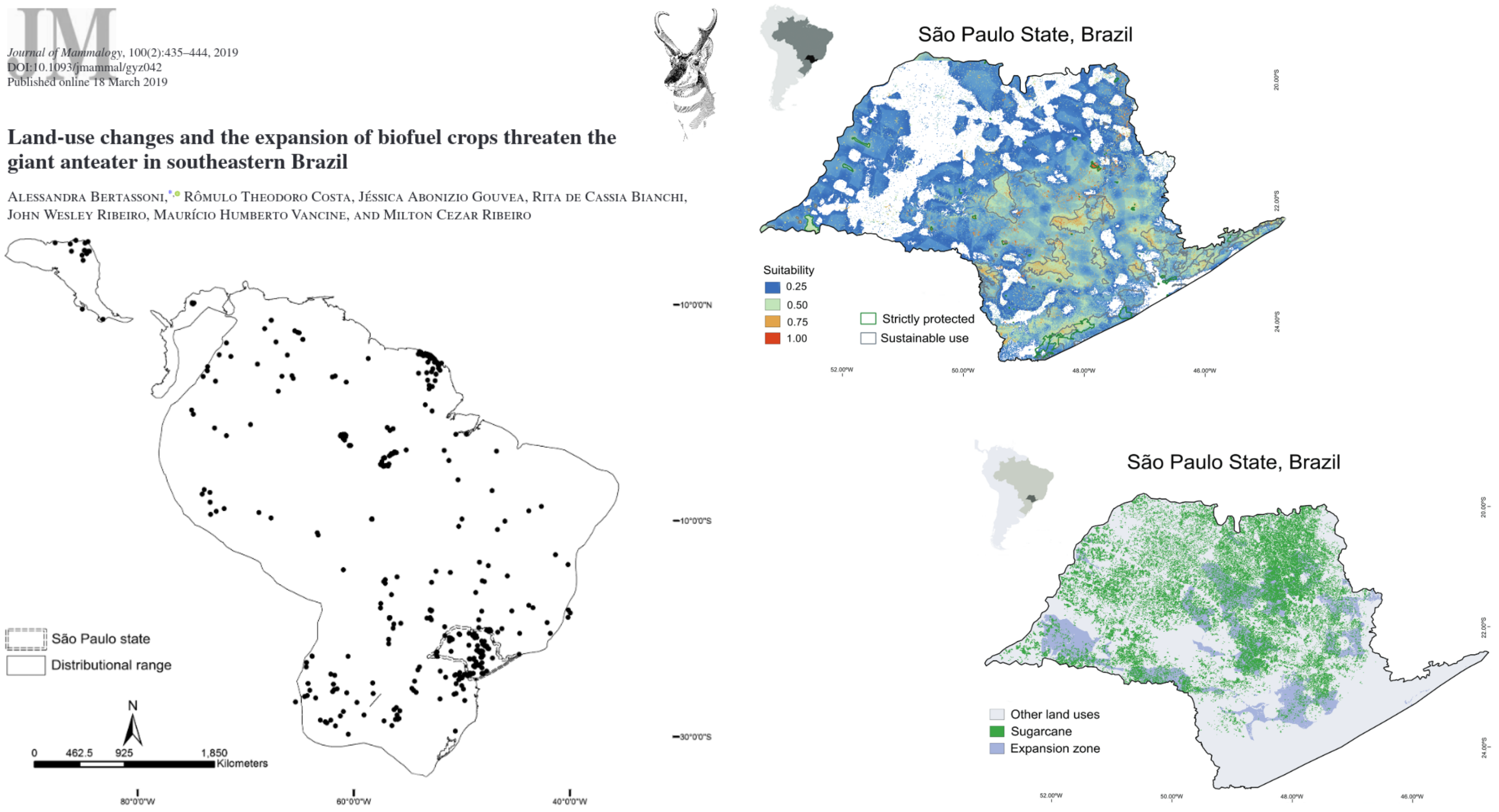
Doenças
Doenças
Áreas de transmissão de Chagas (Trypanosoma cruzi) no Paraná
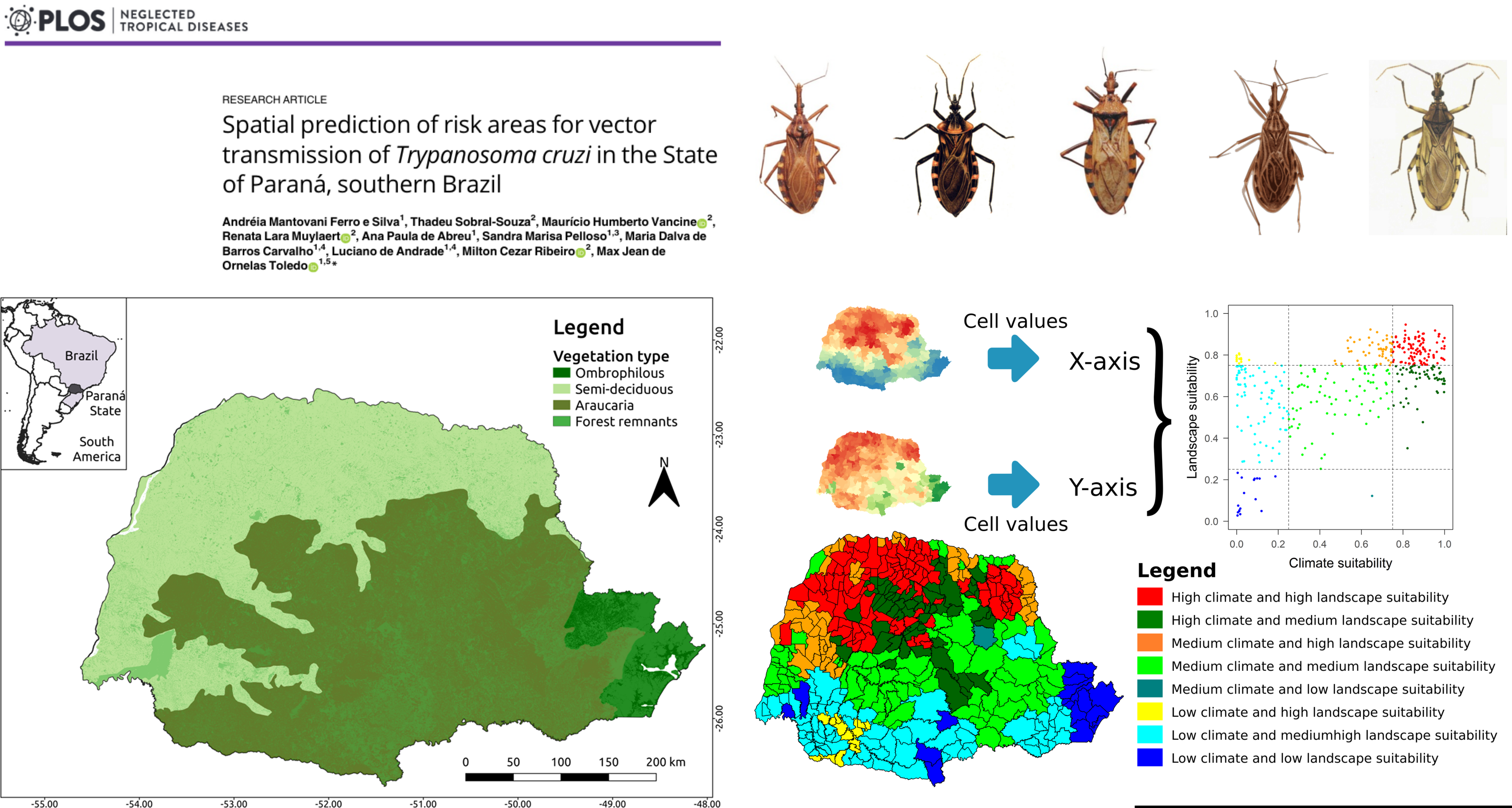
Doenças
Mudanças climáticas e distribuição de morcegos que hospedam vírus da SARS
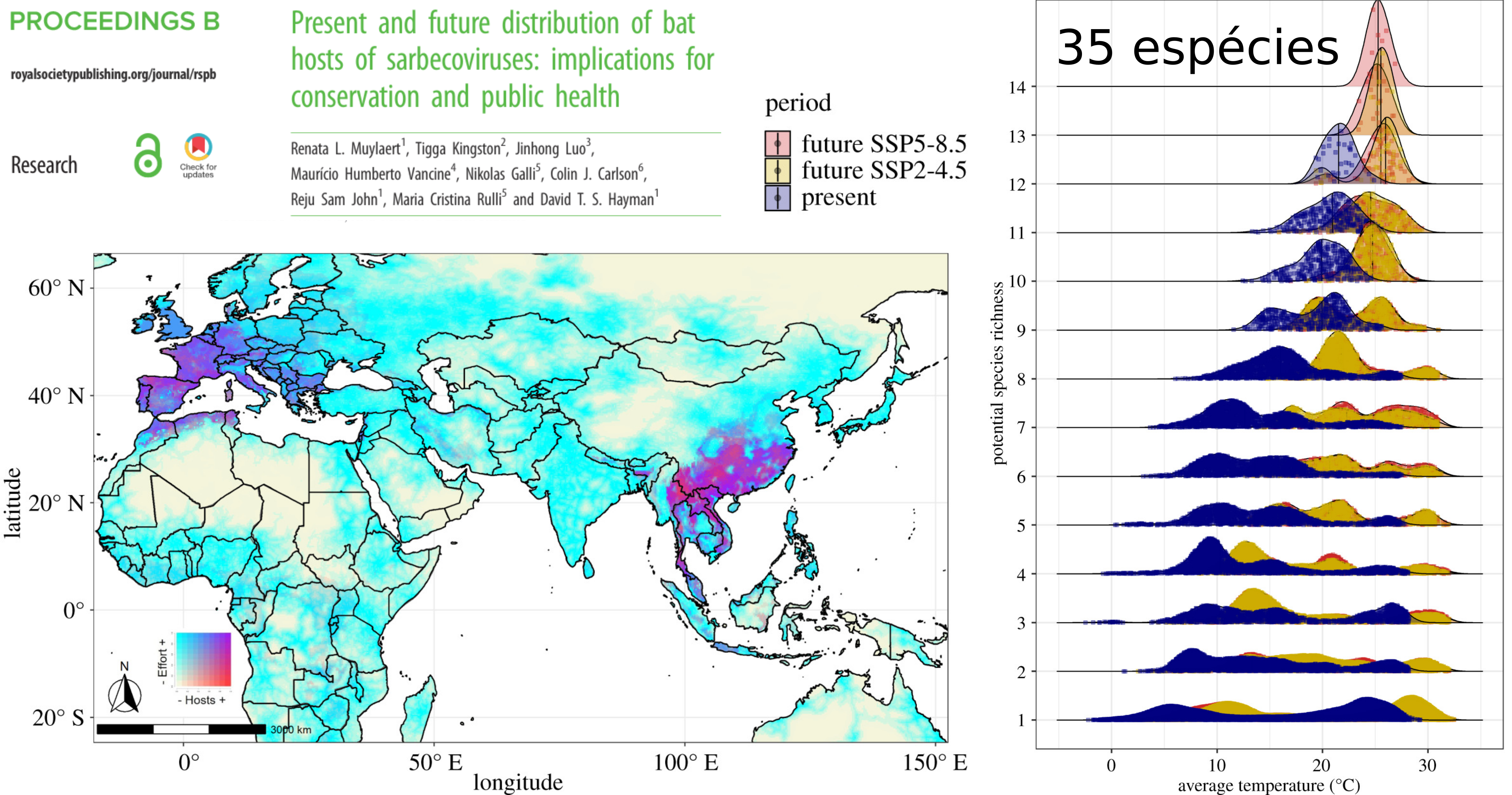
Invasão biológica
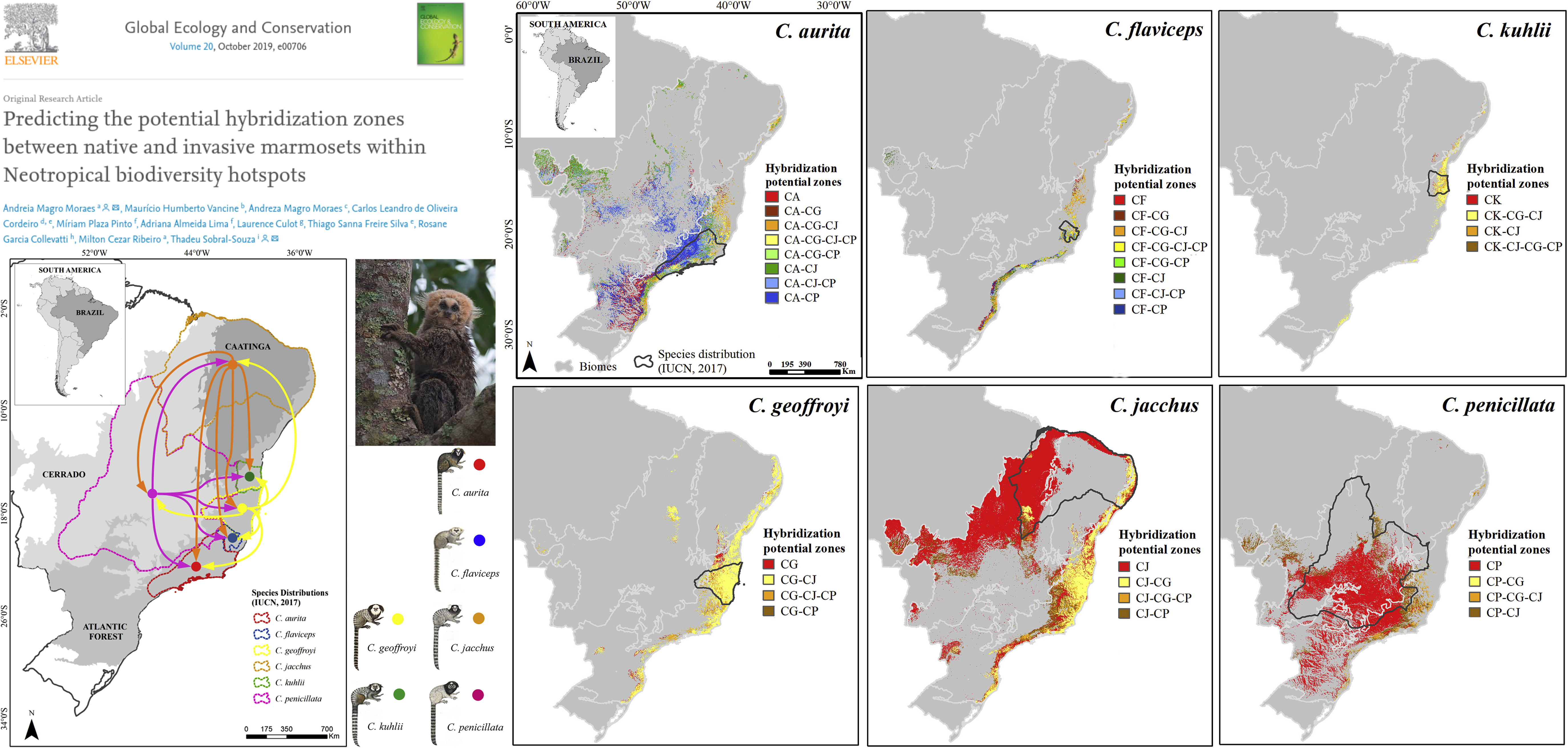
Invasão biológica
Zonas de hibridização potencial de saguis no Brasil
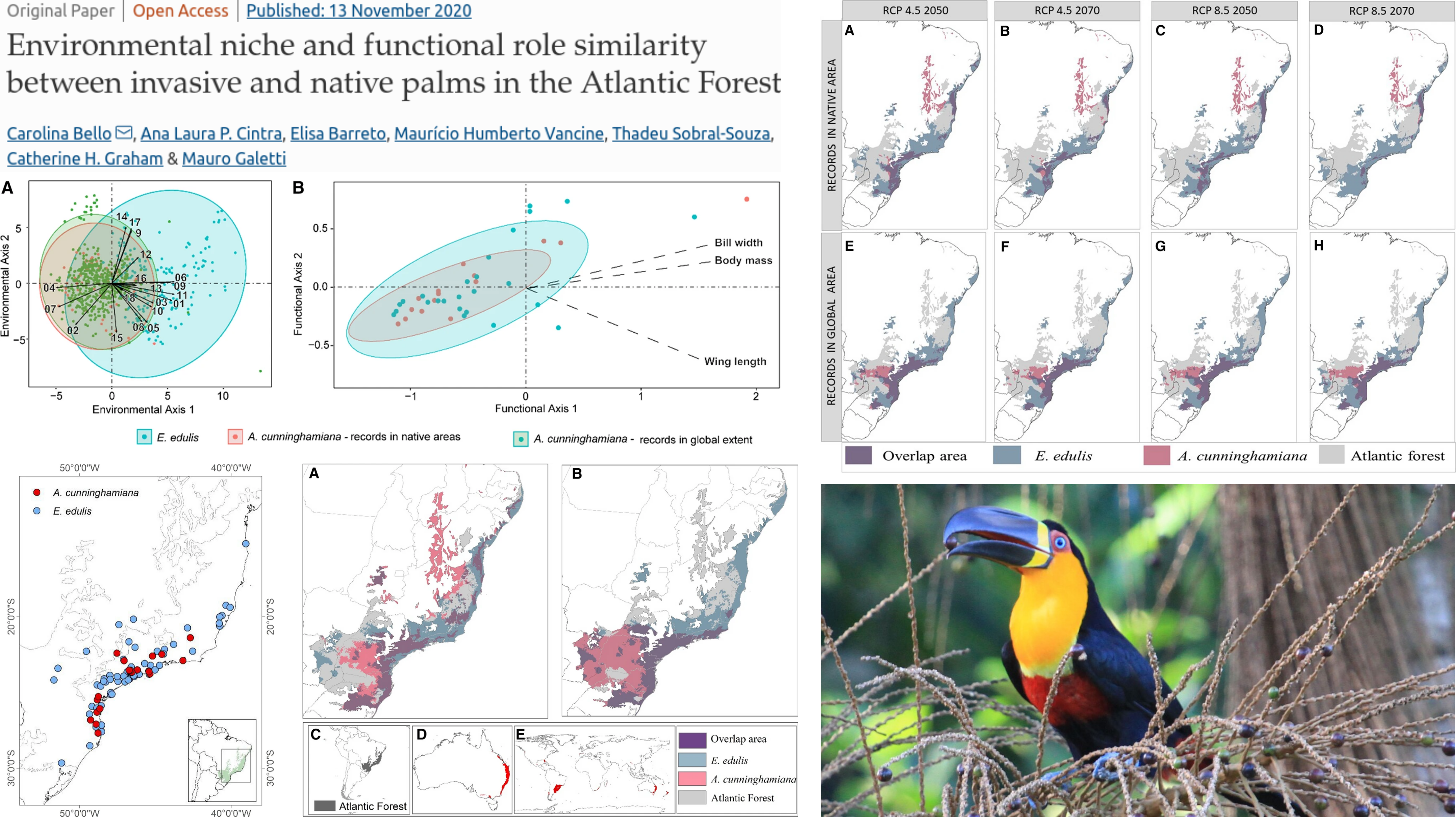
Invasão biológica
Sobreposição de palmeira nativa e invasora na Mata Atlântica
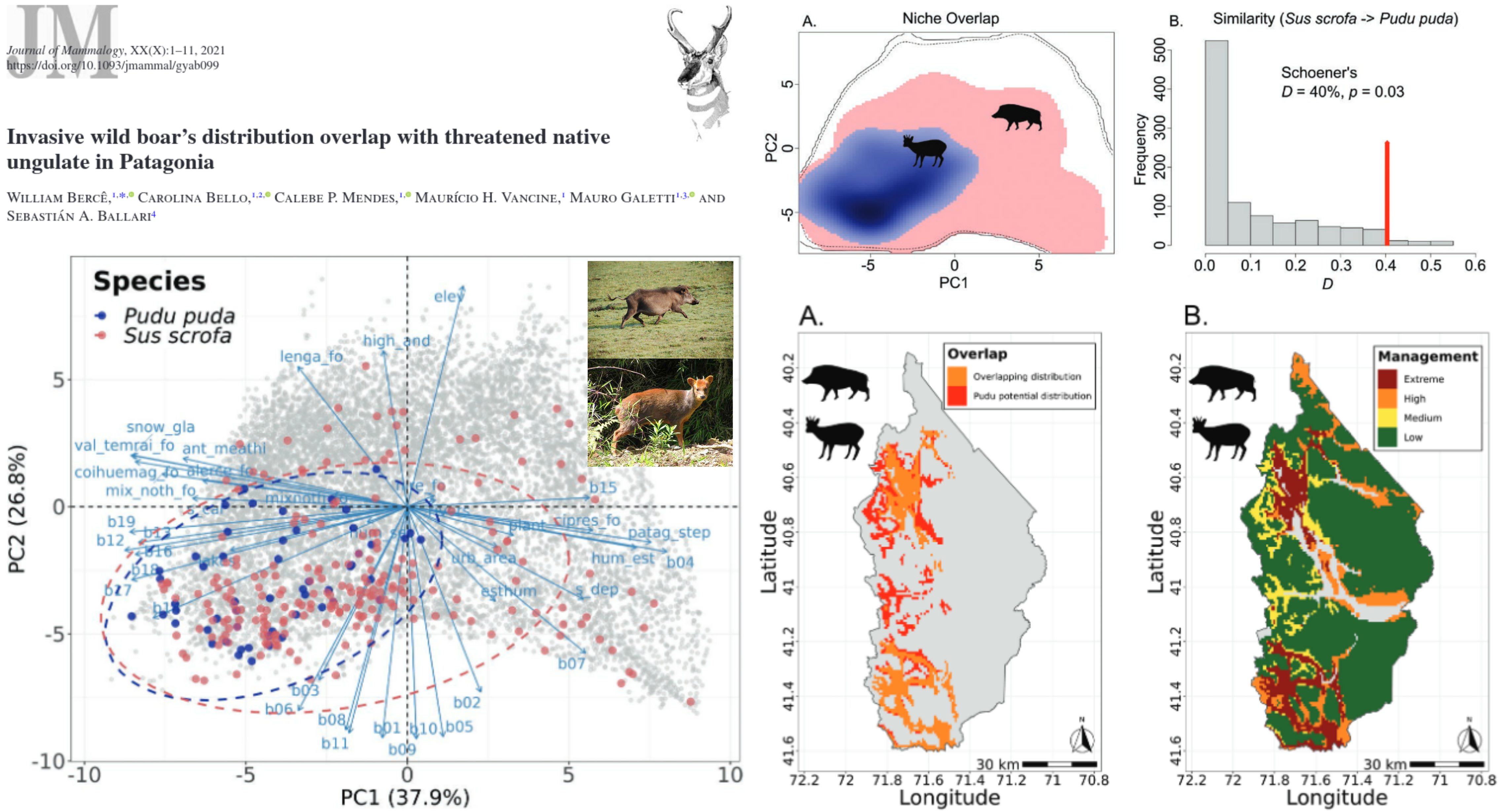
Invasão biológica
Áreas de sobreposição de um cervídeo nativo e javaporco na Argentina
Mudanças Climáticas
Mudanças Climáticas
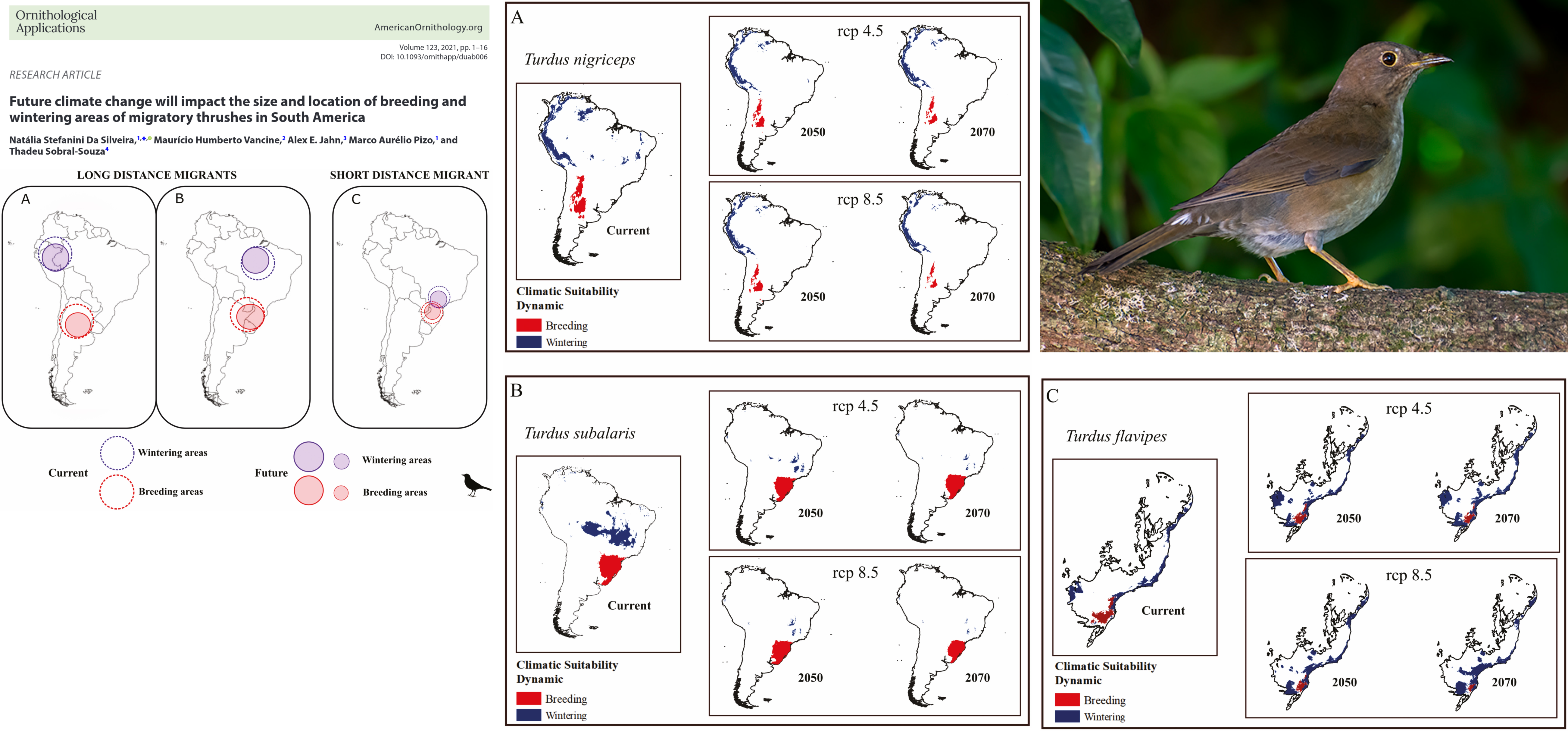
Mudanças climáticas e migração de sabiás na América do Sul
Mudanças Climáticas
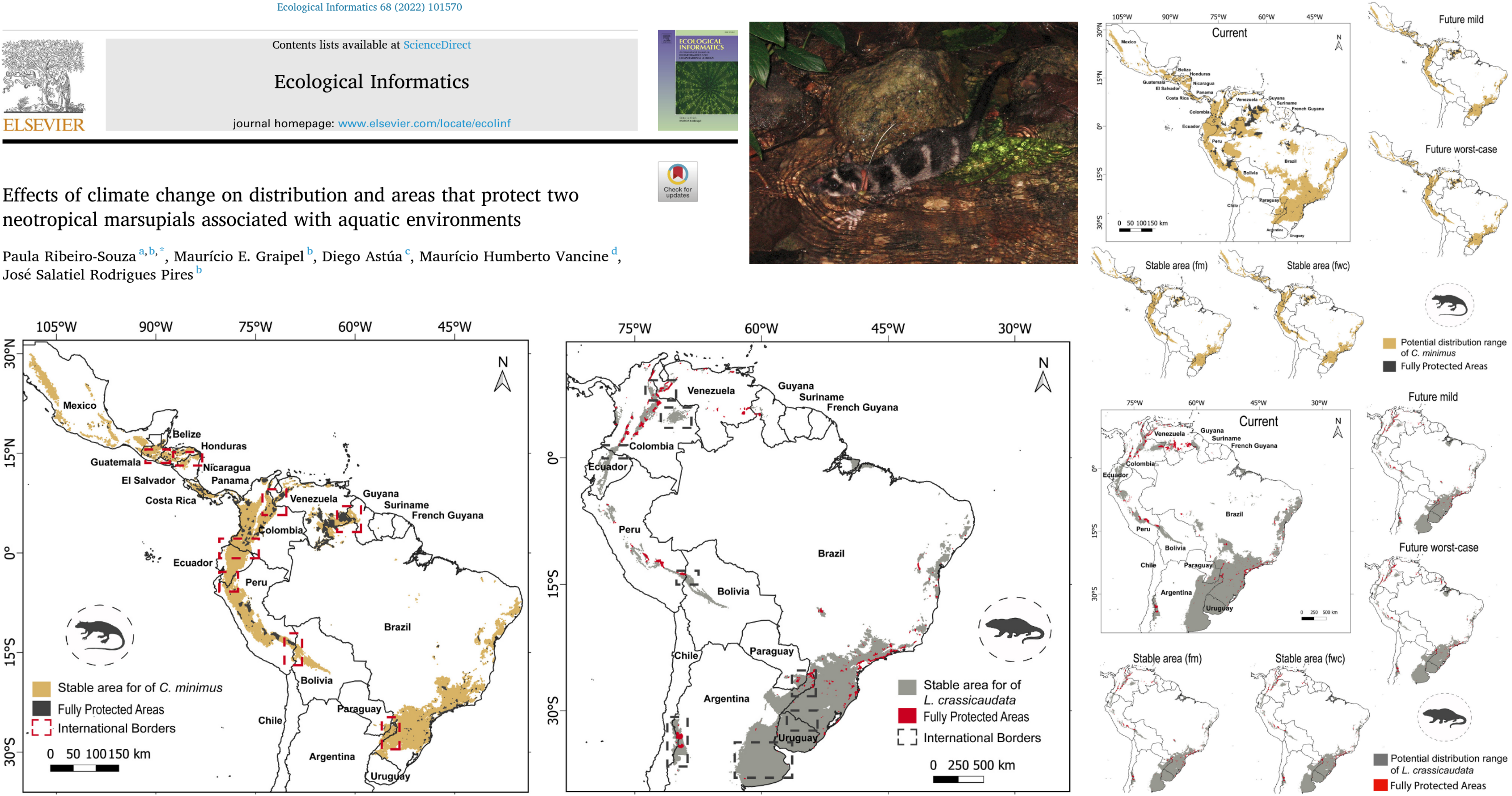
Mudanças climáticas sobre duas espécies de marsupiais na América do Sul
Mudanças Climáticas
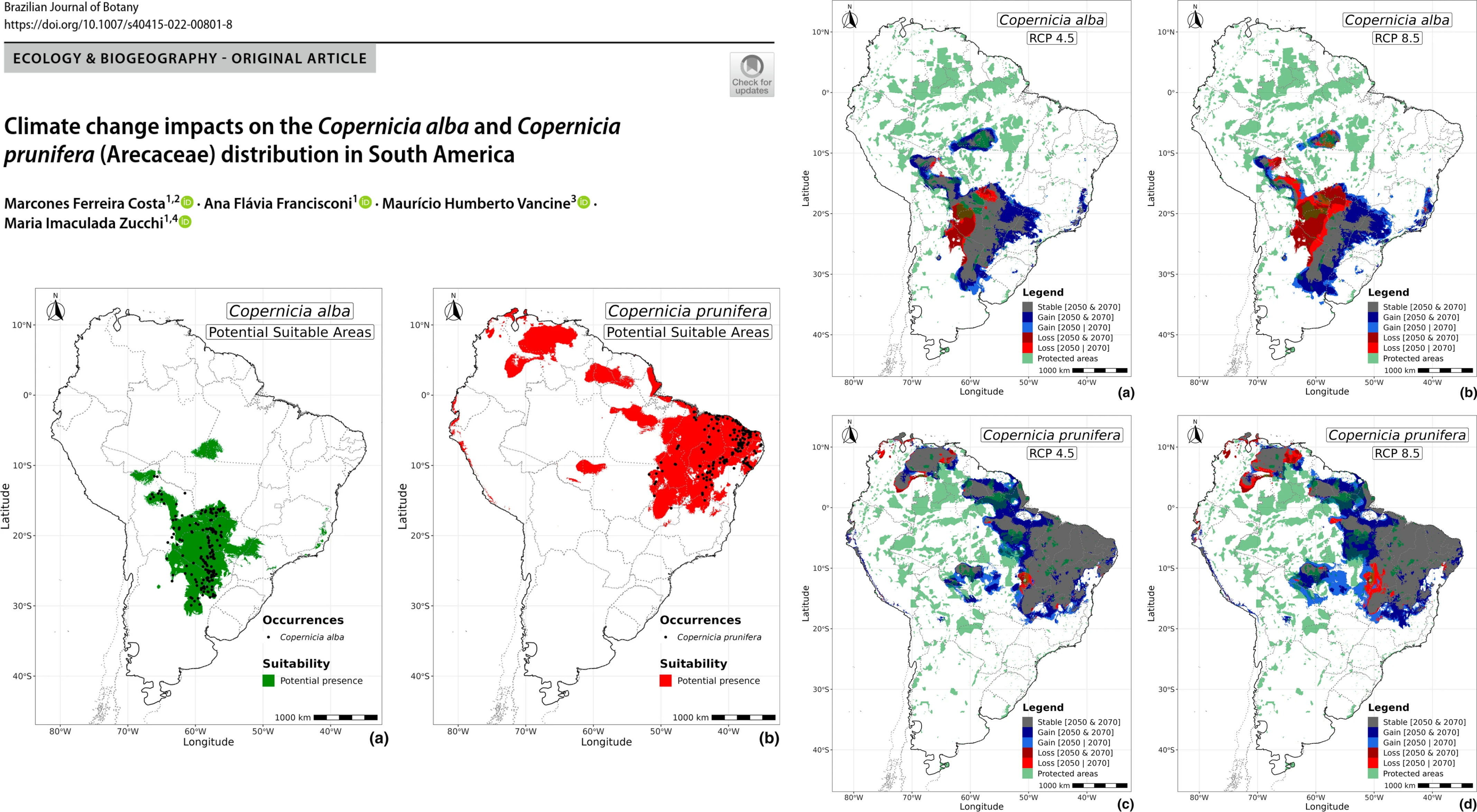
Mudanças climáticas sobre duas espécies de palmeiras na América do Sul
Mudanças Climáticas
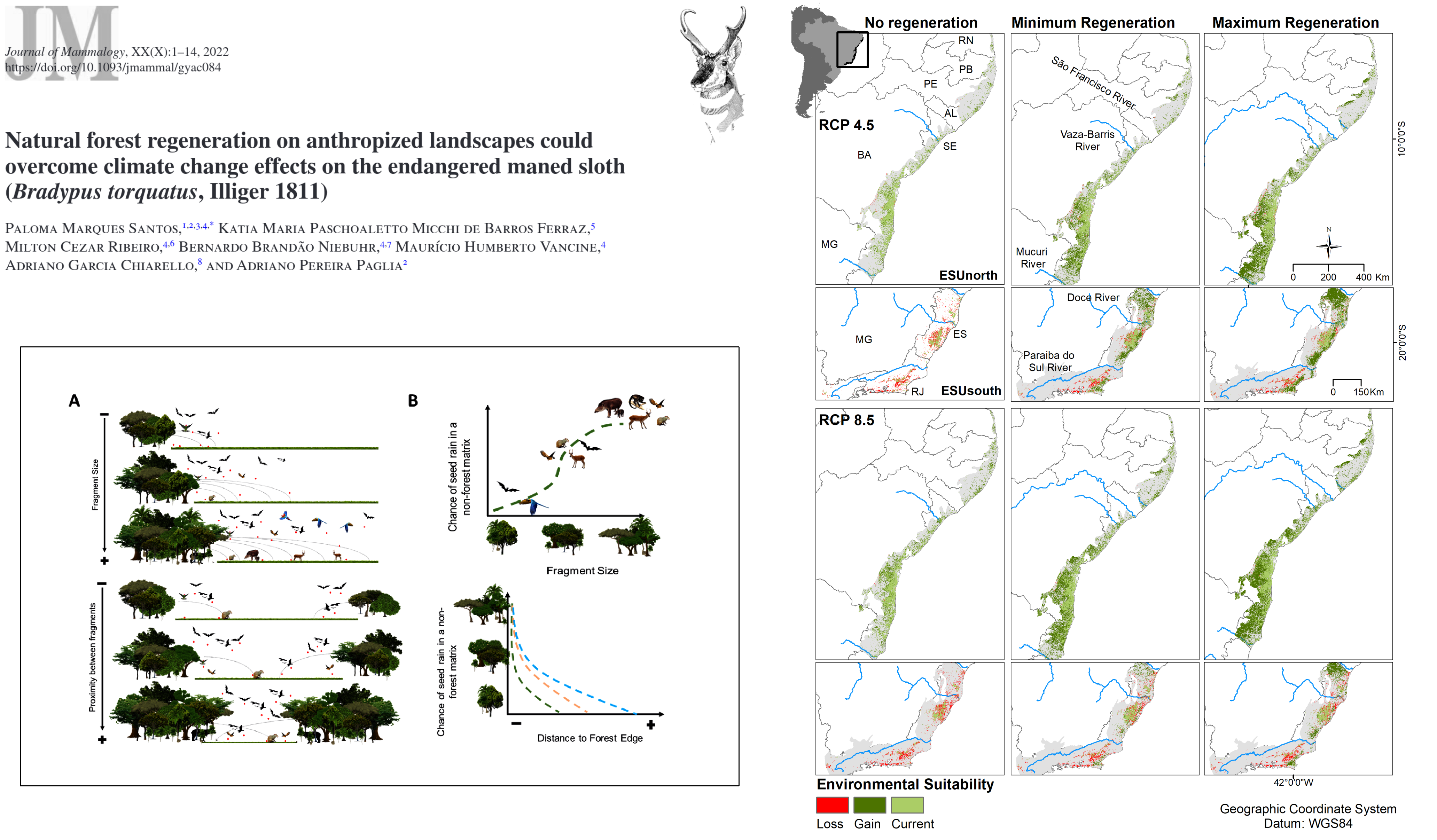
Mudanças climáticas, regeneração potencial e preguiça-de-coleira na Mata Atlântica
Material
Artigos
Livros
Muito obrigado!
Agradecimentos:
Orientador: Miltinho
Flavia Pinto
Thadeu Sobral-Souza
Matheus Lima-Ribeiro
João Giovanelli

Slides por Maurício Vancine, feitos com Quarto. Código disponível no GitHub.
